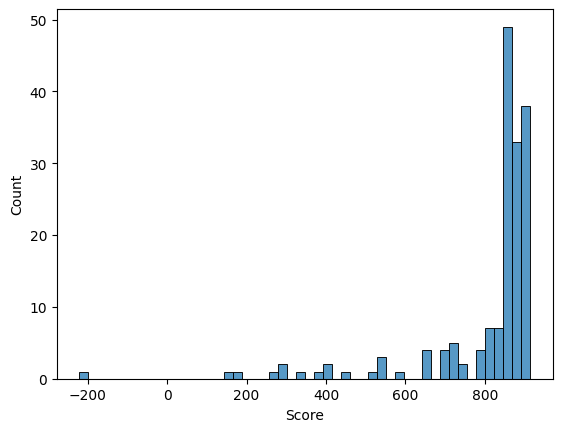
Pic.1. Гистограмма весов белков, видно, что за порог можно взять значение около 800
Для данного практикума взял домен Pyr_redox (PF00070). Он представляет собой небольшой NADH-связывающий домен внутри более крупного FAD-связывающего домена. Для данного домена есть 91 доменных архетиктур. Я выбрал доменную архетиктуру, содержащую 2 домена: Pyr_redox на в середине и FAD-depend_C на С-конце. Она включает 168 белков (репрезентативный - Q9SUH4).
Далее скачал последовательности подсемейства и выровнял их в Jalview алгоритмом Mafft. Удалил последовательности с идентичность больше 97%. Выравненные последовательности доступны по ссылке. Это будет позитивной выборкой.
Далее я обрезал все ненужное и оставил только участок с начала первого домена до конца второго. Удалил последовательности с идентичностью больше 97%. Выравненные последовательности доступны по ссылке. Это будет обучающей выборкой.
Для негативной выборки я объединил все белки из моей доменной архетиктуры, Pyr_redox и Pyr_redox_2 (55 белков), Pyr_redox и FAD_binding (39 белков), Pyr_redox и Pyr_redox_dim в один файл.
Для построения HMM-профиля, его калибровки и поиска по нему белков семейства в последовательностях выравнивания full был использован пакет HMMER со следующими командами:
hmm2build -g hmm.txt domen_seq.fasta
hmm2calibrate hmm.txt
hmm2search --cpu 1 hmm.txt full_2.fasta > hmm2search_out.txt
Профиль HMM доступен по ссылке
Файл с находками в итоговой выборке доступен по ссылке
С помощью скрипта, любезно предаставленным Каримовой Кариной, основные характеристики были собраны в таблицу.

Pic.1. Гистограмма весов белков, видно, что за порог можно взять значение около 800