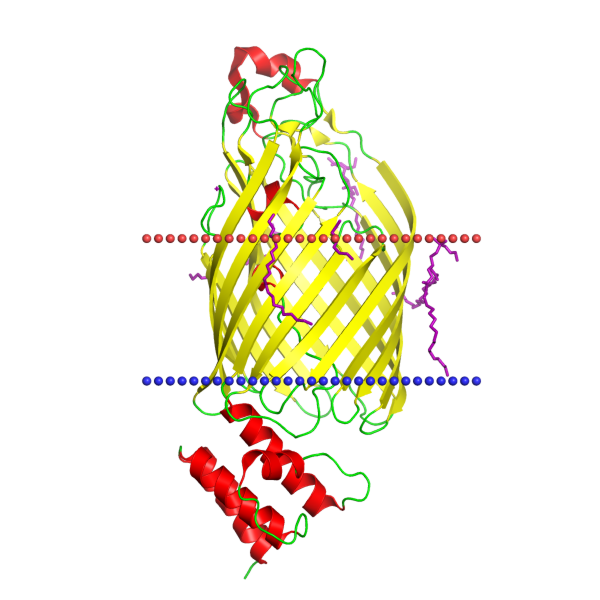
Для данного задания я выбрал белок Cellulose synthase operon protein C (channel BcsC) (белок C оперона целлюлозосинтазы), локализованный в наружней мембране грамотрицательной бактерии Escherichia coli. PDB - 6tzk, Uniprot - BCSC_ECOLI. Целлюлозосинтазный оперон, состоящий из белков A, B, C и D, отвечает за синтез внеклеточной бактериальной целлюлозы. Белок С, являющийся бэтта-бочкой, отвечает за транспорт целлюлозы через внешнюю мембрану наружу.

Координаты трансмембранных участков белка, приведенные в OPM:
1( 785- 793), 2( 807- 815), 3( 821- 829), 4( 866- 873), 5( 880- 884), 6( 894- 901), 7( 907- 917), 8( 944- 952),
9( 960- 968), 10( 980- 989),11( 999-1008),12(1030-1040),13(1046-1057),14(1094-1104),15(1112-1119),16(1125-1134).
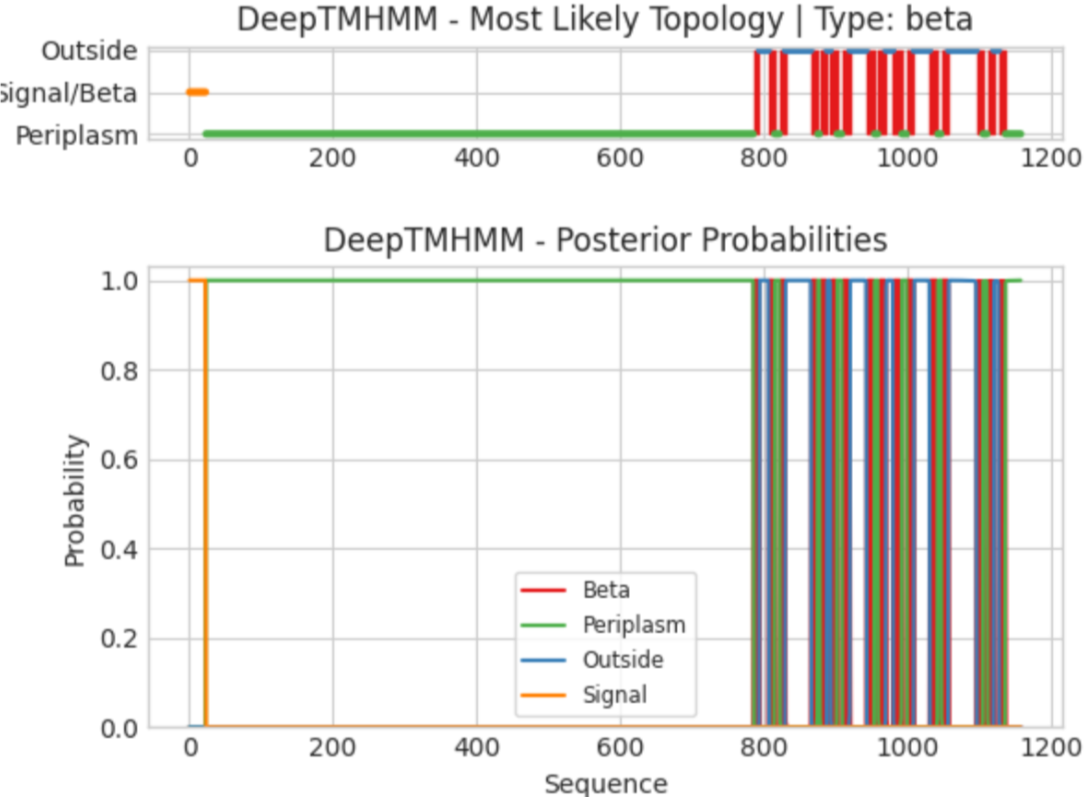
Далее я запустил программу DeepTMHMM для последовательности белка С, взятой в UniProt.
Координаты трансмембранных участков белка, предсказанные DeepTMHMM:
1( 786- 793), 2( 807- 814), 3( 822- 829), 4( 866- 874), 5( 878- 887), 6( 893- 901), 7( 909- 919), 8( 943- 953),
9( 959- 969), 10( 980- 991), 11( 999- 1008), 12( 1031- 1041), 13( 1047- 1056), 14( 1096- 1104), 15( 1111- 1119),
16( 1127- 1135).
Файл с результатами в текстовом виде доступен по ссылке.
Предсказание DeepTMHMM для белка С получилось хорошим - количество трансмембранных участков равно, одиннадцатый участок полностью совпадает, остальные участки различаются в 1-3 аминокислоты. Различия, вероятно, объясняются неточностью программ, так как "лишние" аминокислоты в большинстве случаев являютя нейтральными гидрофобными.
В данном задании рассмотрим трансмембранные участки в выданном мне альфа-спиральном белке Autoinducer 2 (AI-2) import system permease protein LsrC (белок-пермеаза LsrC системы импорта автоиндуктора 2). UniProt - LSRC_ECOLC. Он входит в состав комплекса ABC-переносчиков LsrABCD, который участвует в усвоении AI-2. AI-2 - это сигнальная молекула, используемая для определения кворума, процесса, который бактерии используют для общения и координации группового поведения в зависимости от плотности популяции. LsrC - интегральный мембранный белок, расположенный во внутренней мембране Escherichia coli.
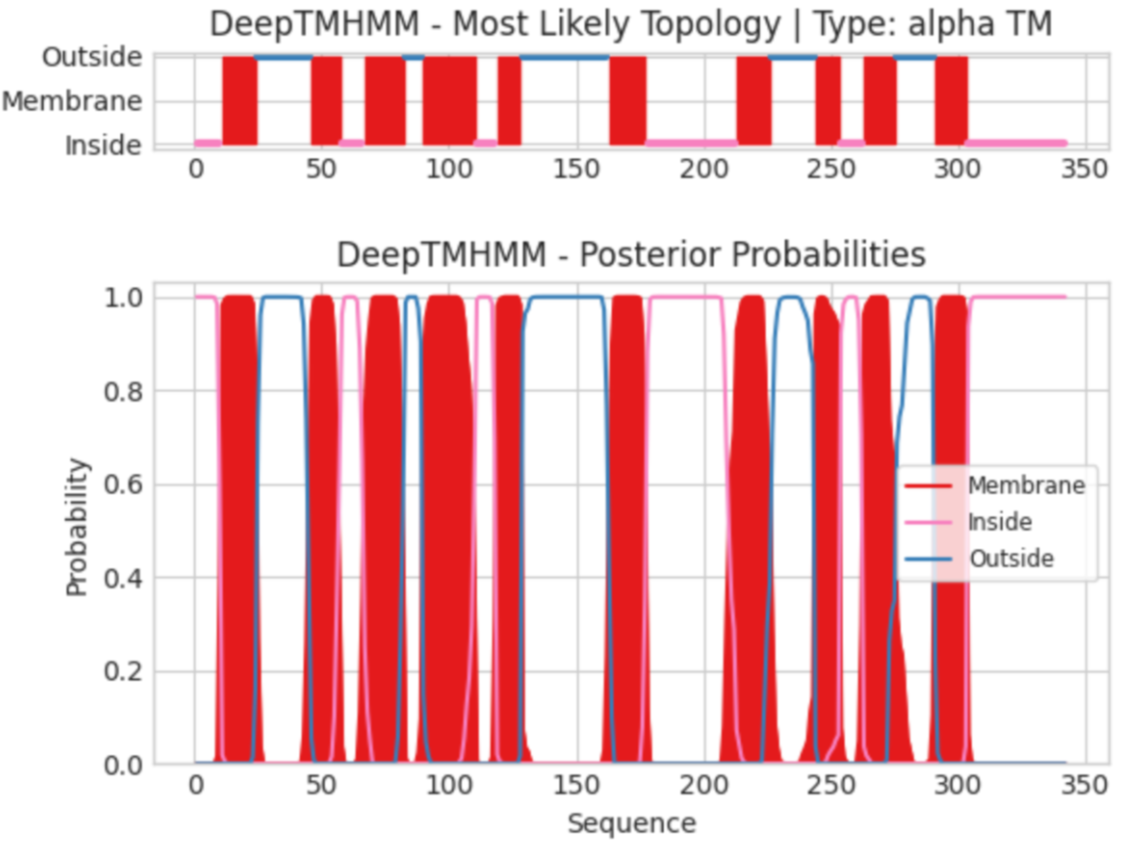
Далее я запустил программу DeepTMHMM для последовательности белка LsrC, взятой в UniProt.
Координаты трансмембранных участков белка, предсказанные DeepTMHMM:
1( 11- 24), 2( 46- 57), 3( 67-82), 4( 90- 110), 5( 119- 128), 6( 163- 177), 7( 213- 226), 8( 244- 253), 9( 263- 275),
10( 291- 303).
Файл с результатами в текстовом виде доступен по ссылке.
Теперь получим модель предсказания белка с помощью программы PPM 3.0. Для этого на вход я загрузил скачанную с UniProt предсказанную структуру AlphaFold со следующими параметрами. Количество мембран - 1, тип мембраны - внутреняя мембрана грамотрицательной бактерии, возможность изгиба - нет, топология N-конца - внутри клетки (предсказание DeepTMHMM).
Координаты трансмембранных участков белка, предсказанные PPM:
1( 4- 27), 2( 37- 60), 3( 65- 78), 4( 88- 113), 5( 117- 135), 6( 161- 180), 7( 204- 225), 8( 240- 251), 9( 262- 279),
10( 288- 308)
Файл в формате pdb, полученный с помощью PPM, доступен по ссылке.
Предсказания DeepTMHMM и PPM похожи: количество трансмембранных участков равно и большей своей частью перекрываются.