Секвенирование по Сэнгеру.
Задание 1
Задача: качественная расшифровка результатов секвенирования по Сэнгеру.
Капиллярный секвенатор выдает файлы с хроматограммой и автоматически прочтённой последовательностью в формате .ab1. (.abi, .ab, .scf). Мне были даны 2 файла в формате .ab1
(54_F.ab1,54_R.ab1), соответствующие прямому и обратному прочтению секвенируемой ДНК.
Для просмотра хроматограмм и редактирования автоматического прочтения я использовала программу Chromas (Lite).Оба заданных файла были открыты в программе Chromas.
Для обратной цепи была открыта комплементарная последовательность (Edit>Reverse+Complement). На данном этапе было сделано выравнивание последовательностей в Jalview и сохранены в формате fasta.
Затем две хроматограммы были выровнены с сохранением масштаба с помощью поиска подслов (find).
Сначала были удалены нечитабельные участки хроматограммы.
- Прямая цепь
- 5' нечитаемый участок: 1-51
- 3' нечитаемый участок: 753-936
- Обратная цепь
- 5' нечитаемый участок: 1-138
- 3' нечитаемый участок: 900-953(в программе 904-953)
В общем случае мое мнение о нечитаемых концах совпадало с выбором программы. Уровень шумов небольшой, в основном последовательность нормально считывается,
сигнал хороший.
Редактирование
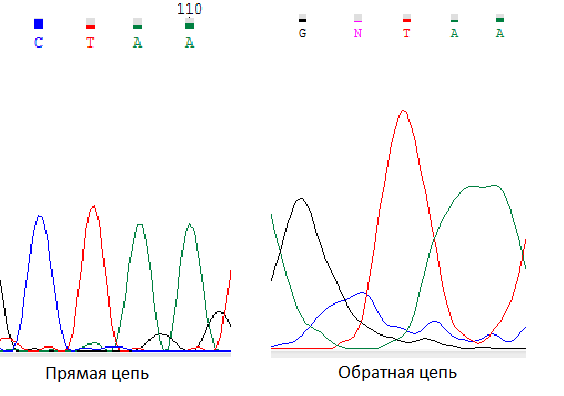
1) Обозначенные буквой N нуклеотиды
был не определен
программой в обратной цепи, так как в ней
на этой позиции наблюдается слабый сигнал,
который легко могут быть спутаны с шумом.
В прямой последовательности на этой позиции
присутствует достаточно выраженный пик
цитозина при слабом
сигнале шума.
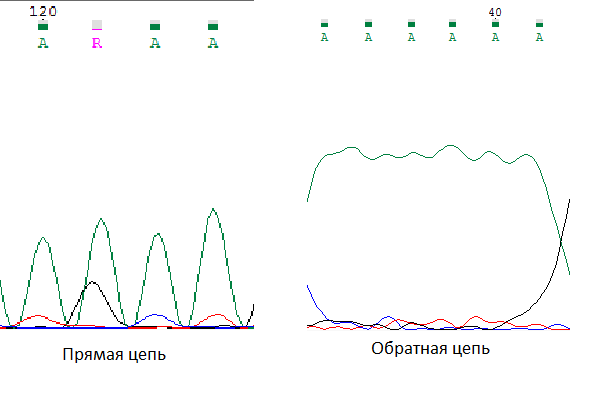
2) На месте нуклеотида R в обратной цепи стоит аденин. Соответсственно
из-за присутствия сильного пика гуанина программе не удалось
однозначно определить нуклеотид. Это возможно исправить за счет обратной цепи. Хоть в ней неявно выражены пики,
программа все равно правильно определила последовательность нуклетидов.
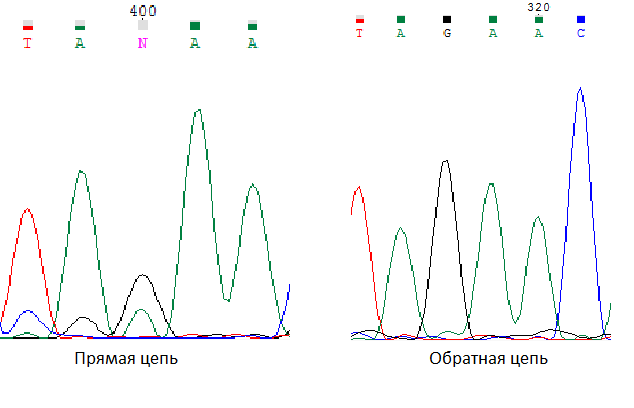
3) Обозначенный буквой N нуклеотид
был не определен программой в прямой
цепи, так как в ней на этой позиции
наблюдается сигнал, практически не
отличающийся по силе от шумового.
В обратной последовательности
присутствует лишь один выраженный пик
гуанина при практически отсутствующем
сигнале шума.
4) N нуклеотид также был неопределен программой из-за лишнего пика гуанина, в обратной цепи он отсутствует.
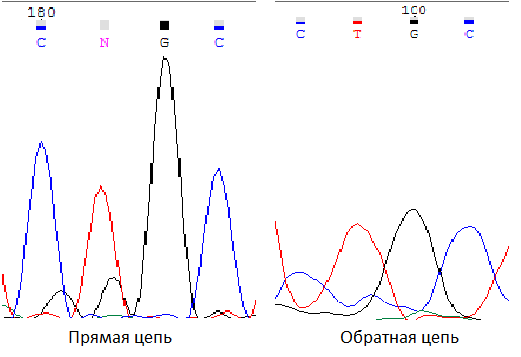
Получилось, что основным исправлением последовательностей была замена нуклеотида.
Редактированные последовательности представлены вверху практикума.
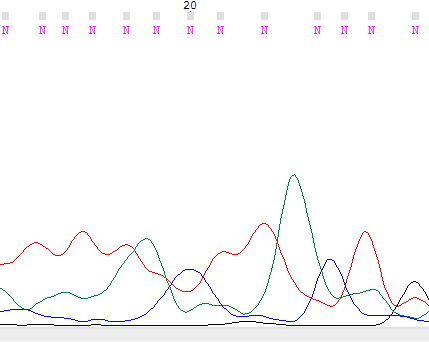
Нечитаемый фрагмент хромотограмы.
Фрагмент взят из директории bad диска P.
Если посмотреть на изображение, то можно сказать, что по цвету возможно определить, сигнал какого нуклеотида наблюдается на хроматограмме. Но нельзя точно сказать в какой последовательности они идут.
, потому что пики находятся друг под другом.
К тому же, невозможно разобрать, какой сигнал яляется шумовым, а какой истинным, ведь все сигналы почти одного уровня. Всё, что можно заключить из этой хроматограммы,
это то, что в этом участке ДНК есть аденин, тимин, цитозин и гуанин, но для анализа последовательности эта информация недостаточна.