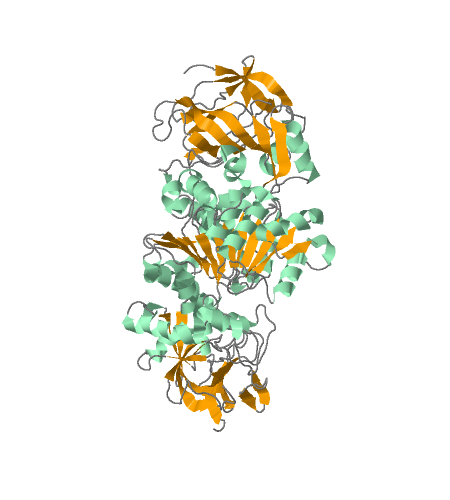
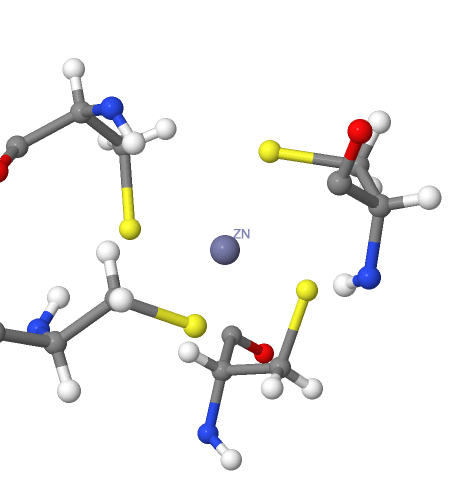
Атлас контактов биомолекулы Alcohol dehydrogenase class-P
Общая характеристика
Название: Alcohol dehydrogenase class-P
Организм: Arabidopsis thaliana
Ген: ADH1
Молекулярный вес: 82.36 кДа [1,2] (димер)
Код фермента: 1.1.1.1[3]
Класс: 1, Оксидоредуктазы
Подкласс: 1.1, Алкогольоксидоредуктазы (взаимодействующие с CH-OH группой донора)
Под-подкласс: 1.1.1, алкоголь: NAD(P)оксидоредуктазы (взаимодействующие с NAD или NADP в качестве акцептора)
Код четвертого уровня: 1.1.1.1, Алкогольдегидрогеназа
PDB: 4RQT
Uniprot ID: P06525
Алкогольдегидрогеназа класса P Arabidopsis thaliana (L.) (ADH) специализируется на катализе окисления первичных спиртов до соответствующих им альдегидов посредством восстановления кофермента никотинамида адениндинуклеотида (NAD). Наиболее активно идет взаимодействие с этанолом (EtOH). Также способна окислять вторичные спирты до соответствующих кетонов[4,5]. Является димером двух идентичных субъединиц.
Энзимология
Каждая субъединица ADH содержит один сайт связывания кофермента NAD, один активный центр и два иона цинка, один из которых находится в активном центре и важен для катализа. Каталитический ион цинка координирован аминокислотами CYS47, CYS177 и HIS69.
Рисунок 2 наглядно демонстрирует работу активного активного центра ADH на примере EtOH. Наиболее широко распространенный механизм окисления первичного спирта ADH следующий[4,6]: Кофермент NAD связывается с активным центром ADH, далее происходит связывание спирта с каталитическим цинком посредством кислорода; спирт депротонируется что, приводит к образованию связанного с цинком алкоксид-иона (1а); тем временем гидрид-анион переходит от иона алкоксида к NAD, восстанавливая его, что также образует связанный с цинком альдегид (1б); после этого альдегид высвобождается из активного центра ADH; протон, ранее отделившийся от спирта через треонин переходит на кислород при втором атоме NAD (2), затем на кислород при третьем атоме (3). Оттуда он уходит на гистидин (4*). После этого протон сольватируется (5) и покидает активный центр ADH, также как и NADH. Стоит отметить, что, несмотря на то, что в обзорах данного белка не было упоминаний о переходе протона на HIS48 (4*), признак наличия данного шага является очень консервативным для ADH среди ортологов у разных царств[4,6] и паралогов в виде ADH коричного спирта, кодируемых генами CAD4 и CAD5[7]. Предполагаемое место HIS51 в данном белке занимает HIS48.
Физико-химические характеристики лигандов
Цинк
Название по IUPAC: Zinc(2+)
Химическая формула: [Zn+2]
Брутто-формула: Zn
Молярная масса: 65.41 г/моль
Сульфат-ион
Название по IUPAC: Sulfate
Химическая формула: [O-S(=O)(=O)O-]
Брутто-формула: O4 S
Молярная масса: 96.06 г/моль
Ацетат-ион
Название по IUPAC: Acetic acid
Химическая формула: CC(=O)O
Брутто-формула: C2 H4 O2
Молярная масса: 60.05 г/моль
Апплет - визуализация внутримолекулярных взаимодействий
Анализ внутримолекулярных взаимодействий
Структура молекулы
Исследуемый белок представляет собой димер из двух идентичных субъединиц, отобразить которые удалось с помощью команды load=4rqt.cif filter "assembly 1".На основании структуры (рис.1) можно определить, что алкогольдегидрогеназа относится к типу α/β-белков, так как альфа-спирали (выделены зеленым) и бета-листы (выделены оранжевым)в структуре чередуются. Стартовый скрипт демонстрирует общий вид молекулы с раскраской по структуре, на котором в виде шаро-стержневых моделей выделены лиганды, которых в составе мономера 6: 3 сульфат-аниона, 2 катиона цинка (каталитический и некаталитический) и ацетат-ион. Второе изображение стартого скрипта показывает аминокислотные остатки, содержащие атомы, находящиеся на расстоянии не менее 5 ангстрем от лигандов, и предположительно участвующие в их связывании с белком.
Строение активного центра, в котором находится атом цинка ZN402, было определено на основании анализа данных Uniprot и PDB-структуры 4RQU, а также структуры аналогичных алкогольдегидрогеназ других организмов. Рисунок 3 отражает положение в пространстве аминокислотных остатков HIS69, CYS177, CYS47, THR49, HIS48, PHE322, THR272 и VAL295. Возможный механизм работы фермента описан выше.
Кристаллы ADH согласно протоколу[5] получали при 22 C в виде висящих капель с использованием 2,0 М сульфата аммония, 0,1 М ацетата (pH 5,9) и 1% глицерина в качестве осадителя. Этим объясняется наличие в pdb-файле сульфат-ионов и ацетата, которые не играют какой-либо роли в деятельности ADH. Предположительно, ацетат остался в активном центре, связанный с каталитическим ионом цинка, благодаря форме, похожей на таковую у субстрата данного фермента, EtOH.
Сайт связывания NAD+ для 4RQT был найден следующим образом. Изучая статьи по данной теме, мы выяснили, что взаимодействие алкогольдегидрогеназы Arabidopsis thaliana (L.) было рассмотрено ранее и отражено в PDB-структуре с ID 4RQU [8], которая была проанализирована нами. Иллюстрация взаимодействия NAD с аминокислотными остатками фермента представлена на рисунке 4, соответствующие аминокислотные остатки указаны в таблице 1, отражающей данные об аминокислотных остатках, обеспечивающих осуществление биологической функции белка.
| Позиция | Аминокислота | Описание |
|---|---|---|
| Металл-связывающие | ||
| 47 | Cys | ZN[402] (Zinc 1), каталитический |
| 49 | Thr | ZN[402] (Zinc 1), каталитический |
| 69 | His | ZN[402] (Zinc 1), каталитический |
| 99 | Cys | ZN[401] (Zinc 2) |
| 102 | Cys | ZN[401] (Zinc 2) |
| 105 | Cys | ZN[401] (Zinc 2) |
| 113 | Cys | ZN[401] (Zinc 2) |
| 177 | Cys | ZN[402] (Zinc 1), каталитический |
| Нуклеотид-связывающие | ||
| 44 | His | NAD |
| 45 | Thr | NAD |
| 200 | Gly | NAD |
| 201 | Ala | NAD |
| 203 | Gly | NAD |
| 222 | Asp | NAD |
| 223 | Phe | NAD |
| 227 | Arg | NAD |
| 268 | Thr | NAD |
| 291 | Val | NAD |
| 318 | Phe | NAD |
| 368 | Arg | NAD |
| Субстрат-связывающие | ||
| 49 | Thr | EtOH |
| Белок-связывающие | ||
| 103 | Arg | ADH |
| 104 | His | ADH |
| 109 | Glu | ADH |
| 110 | Ser | ADH |
| 111 | Asn | ADH |
| 112 | Met | ADH |
| 117 | Arg | ADH |
| 193 | Lys | ADH |
| 266 | Asp | ADH |
| 287 | Asp | ADH |
| 301 | Asp | ADH |
| 304 | Phe | ADH |
| 306 | Thr | ADH |
| 307 | His | ADH |
| 314 | Glu | ADH |
| 315 | Arg | ADH |
| 317 | Leu | ADH |
| 319 | Gly | ADH |
| 321 | Phe | ADH |
| Иные | ||
| 48 | His | Каталитический |
Водородные связи
Межмолекулярные или внутримолекулярные водородные связи возникают тогда, когда вблизи водорода, связанного с электроотрицательным атомом, оказывается другой электроотрицательный атом, имеющий неподеленую электронную пару, и происходит электростатическое притяжение между этим атомом и водородом и смещение электронной пары в направлении атома водорода. Они служат основой формирования вторичной структуры белка - альфа-спиралей и бета-листов. Скрипт "Водородные связи" последовательно выводит изображения альфа-спирали и бета-листа с обозначенными водородными связями, их длинами и значениями углов N-H-O. Для поиска водородных связей использовалась команда hbonds calculate; hbonds 20. Среднее значение длины водородной связи в альфа-спирали - 3.04 ангстрема, угол N-H-O - 151.05 градусов. Для бета-листа соответствующие значения составляют 2.82 ангстрема и 159.25 градусов. Согласно источникам,[9, 10] стандартная длина водородной связи варьирует в пределах 2.7-3.0Å, и с ее увеличением снижается прочность, что позволяет утверждать, что для бета-листов данного белка характерна большая прочность, чем для альфа-спиралей. Угол связи для обоих структур соответствует водородной связи средней силы
Ионные взаимодействия
Ионные взаимодействия в молекулах белков могут возникать при условии, если вблизи отрицательно заряженных радикалов аминокислот (аспарагиновой или глутаминовой кислоты) оказываются положительно заряженные (гистидин, аргинин, лизин). Выбрать участки молекулы, где в теории может реализовываться такое взаимодействие, можно с помощью команд select within(GROUP, (his*.ND1, his*.NE2, lys*.NZ, arg*.NH2, arg*.NH1) and within(3.5, (asp*.OD1, asp*.OD2, glu*.OE1, glu*.OE2))) и select within(GROUP, (asp*.OD1, asp*.OD2, glu*.OE1, glu*.OE2) and within(3.5, (his*.ND1, his*.NE2, lys*.NZ, arg*.NH2, arg*.NH1))), выделяющих аминокислотные остатки, заряженные участки которых расположены не далее 3.5 ангстрем друг от друга (первая команда выделяет положительно заряженные, вторая - отрицательно заряженные). Также список потенциально способных к взаимодействию пар аминокислотных остатков можно получить с помощью программы [12]. Скрипт "Солевые мостики" последовательно выводит изображения трех пар аминокслотных остатков, относительное пространственное расположение заряженных атомов которых допускает взаимодействие. Расстояние между кислородом и азотом разных аминокислотных остатков составляет в среднем около 2.89 ангстрем. На основании полученного изображения можно заключить, что ионные связи соединяют между собой разные элементы вторичной структуры.
Ковалентные взаимодействия
Среди ковалентных взаимодействий, формирующих структуру белка, можно выделить пептидные связи между аминокислотными остатками в первичной структуре, S-S-связи в дисульфидных мостиках и координационные взаимодействия с лигандами - катионами металлов. В рассматриваемой структуре отсутствуют дисульфидные мостики, так как все остатки цистеина расположены на значительных расстояниях друг от друг, что демонстрирует скрипт "Цистеин". Отсутствие S-S-связей подтверждает команда ssbonds on. Достаточно близко располагаются остатки цистеина под номерами 99, 102, 105, 113, а также 47 и 177, но их атомы серы образуют связи с катионами цинка zn401 и zn402, что демонстрирует скрипт "Координационные связи". Согласно данным Uniprot, zn402, образующий связи с двумя аминокислотными остатками цистеина, гистидином и ацетатом, обусловливает каталитическую активность белка. Оба иона цинка находятся в тетраэдрическом окружении, длина связи между цинком и атомами окружения составляет в среднем 2.34 ангстрема.
Взаимодействия с участием ароматических систем
π-π-взаимодействия возникают между ароматическими системами, имеющими плоскую форму, за счет межмолекулярного перекрывания p-орбиталей в π-сопряженных системах. Выявить предполагаемые места возникновения такого взаимодействия можно, выделив все радикалы ароматических аминокислот командой select phe, tyr, trp, далее требуется рассмотреть их взаимное расположение. В зависимости от положения выделяют параллельно сдвинутый и Т-стэкинг. С помощью программы можно получить список пар аминокислотных остатков, между которыми в теории могут возникать не только ароматические взаимодействия, но также π-катионные и сульфо-ароматические. В исследуемой структуре можно отметить несколько примеров Т-стэкинга (скрипт "Стэкинг"), пример параллельно сдвинутого стэкинга и несколько участков, где могут иметь место другие типы взаимодействий с участием ароматических систем.
Взаимодействие с лигандами
В исследуемой структуре присутствуют лиганды 3 видов: сульфат-анионы, 2 катиона цинка (каталитический и некаталитический) и ацетат-анион, связанный с атомом цинка в активном центре. Определить аминокислотные остатки, находящиеся вблизи лигандов и выделить их в структуре можно с помощью команды select within(GROUP, within(5.0, ligand) and protein). Особенности расположения аминокислотных остатков, находящихся на расстоянии не далее 5 ангстрем от лиганда, позволяют судить о ео способе связывания с молекулой белка. Как было рассмотрено выше в разделе, посвященном ковалентным взаимодействиям, катионы цинка имеют координационное окружение, представленное в одном случае 4 аминокислотными остатками цистеина, а в другом - двумя остатками цистеина, гистидином и ацетат-ионом. Таким образом, взаимодействие цинка с белком ковалентное. Рассматривая окружение сульфат-аниона, можно отметить присутствие поблизости положительно заряженных аминокислотных остатков аргинина и гистидина, что может свидетельствовать о существовании ионного взаимодействия. Также возможно образование водородных связей, так как вблизи элетроотрицательных атомов кислорода сульфат-аниона находятся -NH- и NH2-группы аргинина и гистидина, и возможно смещение электронной пары кислорода на свободную орбиталь атомов водорода. Стэкинга и других видов гидрофобных взаимодействий не наблюдается, в связи с отсутствием у лигандов потенциальной возможности образовывать подобного вида контакты.
Анализ расположения атомов в гидрофобном ядре
| Атом PHE67 | Атом другой аминокислоты | Расстояние (Å) |
|---|---|---|
| [PHE]67:A.CA | [PHE]149:A.CG | 4,69 |
| [PHE]67:A.CA | [PHE]149:A.CD2 | 4,15 |
| [PHE]67:A.CA | [PHE]143:A.CB | 4,00 |
| [PHE]67:A.CB | [ASP]50:A.CB | 4,44 |
| [PHE]67:A.CB | [PHE]149:A.CE2 | 4,07 |
| [PHE]67:A.CB | [LEU]22:A.CD1 | 5,81 |
| [PHE]67:A.CD1 | [PHE]143:A.CG | 3,59 |
| [PHE]67:A.CD1 | [PHE]143:A.CE2 | 3,95 |
| [PHE]67:A.CD1 | [PHE]143:A.CE1 | 4,10 |
| [PHE]67:A.CD2 | [TRP]54:A.CB | 4,15 |
| [PHE]67:A.CD2 | [LEU]22:A.CD2 | 4,84 |
| [PHE]67:A.CD2 | [ARG]65:A.CA | 5,43 |
| [PHE]67:A.CD2 | [ARG]65:A.C | 4,72 |
| [PHE]67:A.CE1 | [ASP]50:A.C | 5,23 |
| [PHE]67:A.CE1 | [PHE]53:A.CB | 3,84 |
| [PHE]67:A.CE1 | [ARG]65:A.CZ | 4,94 |
| [PHE]67:A.CE2 | [ALA]15:A.CB | 3,60 |
| [PHE]67:A.CE2 | [ARG]65:A.CA | 5,18 |
| [PHE]67:A.CE2 | [PHE]63:A.CE1 | 4,20 |
| [PHE]67:A.CG | [ASP]50:A.CA | 4,98 |
| [PHE]67:A.CG | [ARG]65:A.CG | 5,13 |
| [PHE]67:A.CG | [PHE]362:A.CZ | 6,01 |
| [PHE]67:A.CG | [ILE]66:A.CA | 4,97 |
| [PHE]67:A.CZ | [ARG]65:A.CB | 3,87 |
| [PHE]67:A.CZ | [TRP]54:A.CA | 3,59 |



Гидрофобные взаимодействия в молекулах белков возникают вследствие того, что энтропийный фактор вынуждает полярный растворитель уменьшать площадь соприкосновения с неполярными молекулами растворенных веществ. При нахождении белка в воде неполярные радикалы аминокислот сближаются и погружаются внутрь молекулы. Совокупность таких радикалов получила название гидрофобного ядра.
Обнаружить гидрофобные участки белка можно с помощью команды select hydrophobic, выделяющей гидрофобные аминокислотные остатки. Найти отдельные ядра можно с помощью следующей программы [13]. В результате работы с ней с последующей передачей полученных данных в команду select и spacefill было получено изображение (рис. 11). Для анализа плотности расположения атомов в гидрофобном ядре был выбран остаток PHE67.
Для выделения атомов молекулы белка, находящихся на расстоянии не далее 7 ангстрем от выбранного остатка, была использована команда select within(GROUP, within(7.0, MyResidue) and not MyResidue and protein), где MyResidue - обозначение выбранного остатка. Изображения атомов, находящихся на меньших расстояниях, с учетом их ван-дер-ваальсовых радиусов были получены командами select (GROUP, within(1.0, MyResidue) and not MyResidue), где первый аргумент within - расстояние от выбранного остатка в ангстремах, и spacefill. Из полученных изображений (скрипт "Гидрофобное ядро") видно, что атомы практически полностью покрывают поверхность остатка уже на расстоянии 5 ангстрем и окончательно - на расстоянии 6 ангстрем.
Чтобы определить, возможно ли поместить внутрь гидрофобного ядра молекулу воды, рассчитаем ее приблизительный диаметр: значение ван-дер-ваальсового радиуса для кислорода составляет 1.4Å, тогда, пренебрегая водородом, для молекулы НОН получим 2.8Å. Чтобы молекула воды могла поместиться, например, между двумя атомами углерода аминокислотных остатков, расстояние между углеродными атомами должно складываться как минимум из двух ван-дер-ваальсовых радиусов углерода и диаметра молекулы воды: 1.85*2 + 2.8 = 6.5Å. Для атомов углерода и азота такое расстояние равно 1.85 + 2.8 + 1.54 = 6.19Å, для двух атомов азота - 1.54*2 + 2.8 = 5.88Å. Все три значения значительно больше наблюдаемого на практике расстояния между ближайшими атомами аминокислотных остатков (среднее арифметическое измеренных расстояний - 4.54Å), следовательно, в выбранное гидрофобное ядро молекулу воды поместить нельзя. Даже рассматривая участок с визуально более "рыхлой" структурой, можно понять путем измерений, что расстояние слишком мало, чтобы вместить даже один атом кислорода, не говоря о сложности проникновения воды в данный участок ядра вследствие нахождения остатка в его внутренней части.
Личный вклад
Богомаз Олеся, Суздаленко Анна - поиск информации о сайте связывания НАД и написание соответствующего скрипта
Кузнеченкова Екатерина - анализ межмолекулярных контактов, верстка сайта
Никонов Михаил - поиск информации о строении и функционировании активного центра, энзимологии, функциях лигандов
P.S. Каждый этап работы выполнялся при активном участии всех членов команды
Ссылки на источники:
- UniProtKB - P06525 (ADH1_ARATH)
- Protein Molecular Weight Calculator - Science Gateway
- "Enzyme nomenclature 1992 : recommendations of the Nomenclature Committee of the International Union of Biochemistry and Molecular Biology on the nomenclature and classification of enzymes" - International Union of Biochemistry and Molecular Biology. Nomenclature Committee
- "Isolation and biochemical analysis of ethyl methanesulfonate-induced alcohol dehydrogenase null mutants of arabidopsis thaliana (L.) Heynh." - Jacobs M., Dolferus R., Van den Bossche D. Biochem. Genet. 26:105-122(1988) [PubMed] [Europe PMC] [Abstract]
- "Purification and enzymatic characterization of alcohol dehydrogenase from Arabidopsis thaliana." - Cheng F., Hu T., An Y., Huang J., Xu Y. Protein Expr. Purif. 90:74-77(2013) [PubMed] [Europe PMC] [Abstract]
- "Computational Studies of the Mechanism for Proton and Hydride Transfer in Liver Alcohol Dehydrogenase." - Pratul K. Agarwal, Simon P. Webb, and Sharon Hammes-Schiffer JACS. 122, 19: 4803-4812(2000)
- "Crystal structures and catalytic mechanism of the Arabidopsis cinnamyl alcohol dehydrogenases AtCAD5 and AtCAD4" - Youn, B., Camacho, R., Moinuddin, S. G. A., Lee, C., Davin, L. B., Lewis, N. G., & Kang, C.Organic & Biomolecular Chemistry, 4(9): 1687(2006)
- Structural insight into the conformational change of alcohol dehydrogenase from Arabidopsis thaliana L. during coenzyme binding - FangFang Chen, Ping Wang, Yan An, JianQin Huang, YingWu Xu - Biochimie 33-39 - 2015
- HighPrecision Measurement of Hydrogen Bond Lengths in Proteins by Nuclear Magnetic Resonance Methods - Harris - 1999 - Proteins: Structure, Function, and Bioinformatics.
- Steiner, T. The Hydrogen Bond in the Solid State / T. Steiner // Angew. Chem. Int. Ed. - 2002. - V. 41. - P. 48-76.
- ChemDraw JS
- Поиск контактов
- Поиск гидрофобных ядер