Поиск информации о белке с использованием базы данных Uniprot
Общая информация о белке в Uniprot
| Раздел UniProtKB | Swiss-Prot |
|---|---|
| UniProt ID | FHMCD_RHILO |
| UniProt AC | Q988C8; B6ZH65; |
| EMBL AC | AB362565; BAH02784.1; -; Genomic_DNA; BA000012; BAB53022.1; -; Genomic_DNA |
| PDB ID | 4OM8 (структура известна для всего белка) |
| RecName | Full: 5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate 5-dehydrogenase Short: FHMPC dehydrogenase EC=1.2.1.100 |
| Длина | 309 AA |
| Молекулярная масса | 33061 MW |
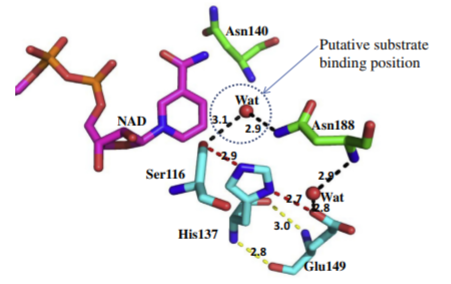
Апплет демонстрирует структуру белка 5-формил-3-гидрокси-2-метилпиридин 4-карбоксилат 5-дегидрогеназы. Синим цветом показана цепь А, зеленым - цепь В. В шаростержневой модели показаны лиганды: ACT - ацетат-ион, BME - бета-меркаптоэтанол, GOL - глицерол, NAD - никотинамид-аденин-динуклеотид. В записи Uniprot вторичная структура для одной из идентичных цепей белка описана в поле FT. Для каждой альфа-спирали (HELIX), поворота (TURN) и бета-листа (STRAND) указан диапазон номеров аминокислотных остатков, их образующих. Для одной цепи белка определены структуры 15 альфа-спиралей, 12 бета-листов и 3 поворотов. Также в данном поле указаны диапазон номеров аминокислотных остатков цепи (CHAIN), участки связывания НАД (NP_BIND, BINDING) и активный центр (SITE). Исследуемый белок имеет нарушенный фенотип, поэтому для него также указаны участки, структура которых изменена мутацией (MUTAGEN), причем в качестве примечания в записи содержится информация о том, какая замена произошла в результате мутации и как она повлияла на функциональную активность.
Организм
Организм: Mesorhizobium japonicum
Таксономическое положение: царство Bacteria, тип Proteobacteria, класс Alphaproteobacteria, порядок Rhizobiales, семейство Phyllobacteriaceae, род Mesorhizobium, вид Mesorhizobium japonicum.
Mesorhizobium japonicum - симбиотическая азотфиксирующая грам-отрицательная палочковидная бактерия. Впервые была обнаружена в Японии, а образец для секвенирования генома был взят из корневых клубеньков лотоса японского.[4] Геном представлен хромосомой и двумя плазмидами.[1] Температурный оптимум для нормальной жизнедеятельности бактерии составляет 28 градусов по Цельсию, при этом она способна продолжать активный рост при температурах от 15 до 34 градусов. Оптимальное значение рН составляет 7, активный рост идет при рН от 6 до 8. Данная бактерия чувствительна к действию большинства антибиотиков, проявляет резистентность только к эритромицину и клоксациллину.[4]
Исследование протеома бактерии
| Организм | Mesorhizobium japonicum | Nitratireductor basaltis |
|---|---|---|
| ID протеома | UP000000552 | UP000053675 |
| Количество белков | 7255 | 3449 |
| Количество белков в Swiss-Prot | 613 | 0 |
| Трансмембранные белки | annotation:(type:transmem) AND organism:"Mesorhizobium japonicum (strain LMG 29417 / CECT 9101 / MAFF 303099) (Mesorhizobium loti (strain MAFF 303099)) [266835]" AND proteome:up000000552 | annotation:(type:transmem) AND organism:"Nitratireductor basaltis [472175]" AND proteome:up000053675 |
| Количество трансмембранных белков | 1312 | 716 |
| Количество трансмембранных белков в Swiss-Prot | 53 | 0 |
| Ферменты | ec:* AND organism:"Mesorhizobium japonicum (strain LMG 29417 / CECT 9101 / MAFF 303099) (Mesorhizobium loti (strain MAFF 303099)) [266835]" AND proteome:up000000552 | ec:* AND organism:"Nitratireductor basaltis [472175]" AND proteome:up000053675 |
| Количество ферментов | 898 | 708 |
| Количество ферментов в Swiss-Prot | 404 | 0 |
| Азотфиксация | goa:("nitrogen fixation [9399]") AND organism:"Mesorhizobium japonicum (strain LMG 29417 / CECT 9101 / MAFF 303099) (Mesorhizobium loti (strain MAFF 303099)) [266835]" AND proteome:up000000552 | goa:("nitrogen fixation [9399]") AND organism:"Nitratireductor basaltis [472175]" AND proteome:up000053675 |
| Количество белков азотфиксации | 28 | 7 |
| Количество белков азотфиксации в Swiss-Prot | 6 | 0 |
Для конкретного вида Mesorhizobium japonicum референсный протеом в базе данных отсутствует. Для сопоставления был взят референсный протеом близкородственного вида Nitratireductor basaltis, относящийся к тому же семейству, что и рассматриваемый организм. Основанием для выбора этого представителя послужило наличие у него гена, кодирующего исследуемый фермент, с тем же названием, что и у Mesorhizobium japonicum. Родовое название этой бактерии указывает на её экологическую роль - участие в процессах денитрификации, следовательно она, как и Mesorhizobium japonicum, вовлечена в круговорот азота. Известно, что многие денитрификаторы также способны к азотфиксации[5], поэтому в качестве одного из направлений исследования допустимо взять присутствие в протеоме азотфиксирующей и нитратредуцирующей бактерий белков, так или иначе вовлеченных в данный процесс. Протеом Nitratreductor basaltis не содержит белков со статусом reviewed, что может свидетельствовать о пока еще недостаточной степени изученности данного организма. При рассмотрении протеомов других видов Mesorhizobium также было отмечено, что белков, внесенных в Swiss-Prot достаточно мало, и в основном они принадлежат Mesorhizobium japonicum.
Выше в таблице 2 представлены результаты поисковых запросов по протеомам выбранных организмов. Протеом Nitratireductor basaltis по количеству белков составляет меньше половины от протеома Mesorhizobium japonicum, и в долях, занимаемых белками определенных функциональных групп, также наблюдаются различия. При этом если в долях трансмембранных белков различие не очень значительно - 18% у Mesorhizobium japonicum и 21% у Nitratireductor basaltis - то для ферментов это значение уже различается сильнее: 12% у Mesorhizobium japonicum и 20.5% у Nitratireductor basaltis. Последний факт можно объяснить, при исследовании протеома Nitratireductor basaltis больше внимания уделялось потенциальным ферментам, поэтому известное их количество всего на 200 меньше, чем для исследуемой бактерии, хотя общий размер протеома меньше вдвое. Белки, вовлеченные в процесс азотфиксации, действительно были найдены у обоих представииелей, причем в протеоме Mesorhizobium japonicum их доля составила 0.4%, а у Nitratireductor basaltis - 0.2%, что вполне согласуется с принадлежностью их к определенным экологическим группам.
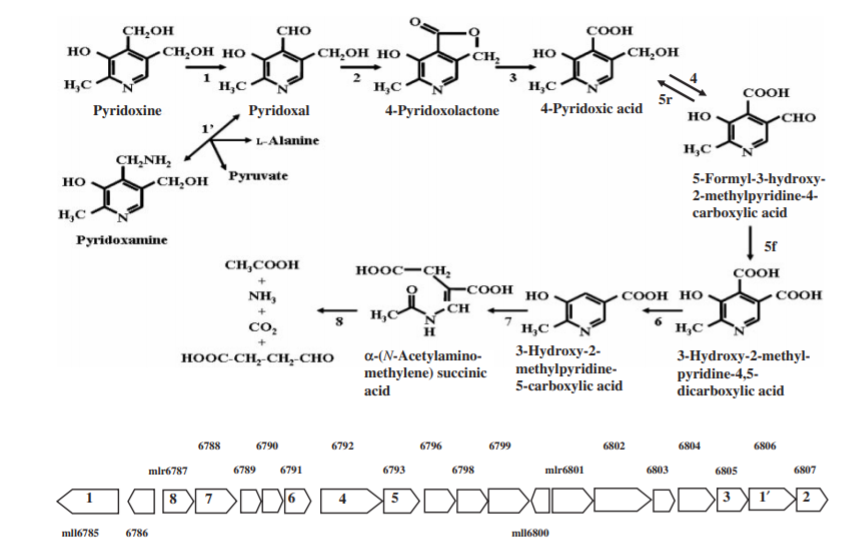
Особенностью метаболизма данной бактерии является способность осуществлять разрушение витамина В6 посредством 9 ферментативных реакций[2], которые представлены на схеме ниже:
Функциональные особенности белка
Описываемый белок вовлечен в метаболизм жирных кислот и катаболизм витамина В6, согласно данным из "электронной аннотации", основанным на описании функций гомологичных белков (IEA: InterPro). Экспериментальная проверка этих данных не проводилась. Структура рассматриваемого образца нарушена мутацией, затрагивающей аминокислотные остатки HIS137 (замена на лизин) и GLU149 (замена на глутамин). Эксперимент показал, что в позиции, соответствующей HIS137, приводит к потере ферментативной активности, а в случае GLU149 - к изменчивости оптимума рН в зависимости от субстрата. [2]
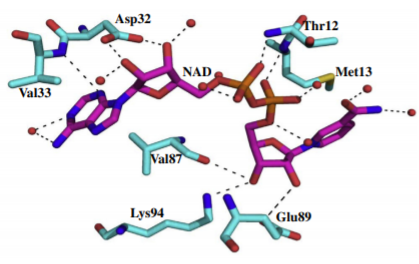
Участвует в разрушении пиридоксина (витамина В6), катализирует окисление 5-формил-3-гидрокси-2-метилпиридин-4-карбоксилата до 5-гидрокси-6-метилпиридин-3,4-дикарбоксилата при участии НАД+ или его восстановление до 4-пиридоксиновой кислоты при участии НАДН. Обе реакции протекают практически необратимо. Оптимальное значение рН - 8.5, температура - 55 градусов по Цельсию. Представляет собой гомодимерный белок, не содержащий металла в качестве кофактора. Относится к семейству 3-гидроксиацил-СоА дегидрогеназ. В активном центре находятся аминокислотные остатки HIS137 и GLU149, основная роль в катализе принадлежит остатку гистидина, глутамат является вспомогательным. [2] Связывание НАД обеспечивается 12-13, 32-33, 87-89 и 94 аминокислотными остатками.[3]
В записи Uniprot информация о функции и свойствах белка содержится в поле СС. В разделе FUNCTION перечислены процессы, в которых участвует белок, далее в разделах CATALYTIC ACTIVITY, число которых соответствует количеству катализируемых процессов, содержатся уравнения реакций и указания на направление их протекания в живых организмах. Для каждого частвующего в реакции вещества указан идентификатор CHEBI. В разделе BIOPHYSICOCHEMICAL PROPERTIES перечислены кинетические параметры процессов: константы Михаэлиса-Ментен (для FHMPC она составляет 48.2 uM в присутствии НАД и 24.9 uM в присутствии НАДН, для НАД - 34.3 uM, для НАДН - 12.4 uM), количество молекул субстрата, связывающихся с активным центром фермента в единицу времени (204 частицы в секунду для реакции окисления и 217 - для реакции восстановления), оптимальные значения рН и температуры. В разделе PATHWAY перечислены метаболические пути, в которых участвует фермент, SUBUNIT - описывает четвертичную структуру, DISRUPTION PHENOTYPE - указывает на наличие мутаций, SIMILARITY - принадлежность к определенному семейству белков.
Анализ кластеров UniRef
| Раздел UniRef | Кластер | Всего белков | Белков из базы Swiss-Prot | Белков из UniParc |
|---|---|---|---|---|
| UniRef50 | UniRef50_Q988C8 | 254 | 1 | 73 |
| UniRef90 | UniRef90_Q988C8 | 72 | 1 | 23 |
| UniRef100 | UniRef100_Q988C8 | 1 | 1 | 0 |
Во всех кластерах данный белок является репрезентативной последоватеьностью (representative) и самой длинной последовательностью кластера, на основе которой проверяется принадлежность к кластеру других белков (seed), что связано с тем, что исследуемый белок является единственным, имеющим статус reviewed, в найденных объединениях. В кластере UniRef с долей совпадения 90% обнаружено только два белка, принадлежащих бактериям рода Phyllobacterium того же семейства, к которому принадлежит и рассматриваемый организм, остальные же принадлежат видам рода Mesorhizobium, как и исследуемый белок, что свидетельствует о невысокой распространенности подобных белков. В кластере с долей совпадения 50% разнообразие видов уже выше, причем среди них встречаются принадлежащие типу Actinobacteria. Можно отметить, что подобные белки чаще встречаются у рода Streptomyces, в остальном - у единичных представителей.
Запросы в Uniprot
Запросы были направлены на:
- поиск информации о данном белке, обнаруженном у организмов других родов и семейств того же типа
- выяснение, был ли белок организма выделен экспериментально, или данные о нем были получены на основе сходства его структуры с уже изученными
- анализ сайта связывания с НАД у белков разных организмов
- поиск информации о генах с таким же названием у других организмов
- исследование распространенности белков, осуществляющих катаболизм витамина В6
| Tекст запроса | Swiss-Prot | TrEMBL | Комментарий |
|---|---|---|---|
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase | 1 | 21 | найдены все записи о данном белке у разных организмов |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase taxonomy:proteobacteria | 1 | 13 | найдены записи о данном белке, обнаруженном у организмов в рамках типа |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase taxonomy:streptomyces | 0 | 3 | найдены записи о данном белке, обнаруженном у организмов в рамках рода Streptomyces |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase NOT taxonomy:phyllobacteriaceae | 0 | 14 | найдены записи о данном белке, обнаруженном у организмов, не принадлежащих к семейству исследуемого организма |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase NOT taxonomy:mesorhizobium | 0 | 17 | найдены записи о данном белке, обнаруженном у организмов, не принадлежащих к роду исследуемого организма |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase NOT organism:"mesorhizobium japonicum" | 0 | 21 | найдены записи о данном белке, обнаруженном у организмов других видов, отличных от исследуемого |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase existence:"Evidence at protein level [1]" | 1 | 0 | выделен и изучен данный конкретный белок |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase existence:"Inferred from homology [3]" | 0 | 20 | описание функций основано на данных о гомологичных белках |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase existence:"Predicted [4]" | 0 | 1 | функция белка предсказана |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase annotation:(type:np_bind nad length:[1 TO 2]) | 1 | 0 | длина участка связывания НАД - 1-2 аминокислотных остатка |
| name:"5-formyl-3-hydroxy-2-methylpyridine 4-carboxylate" name:dehydrogenase annotation:(type:np_bind nad length:[4 TO 6]) | 0 | 21 | длина участка связывания НАД - 4-6 аминокислотных остатков |
| taxonomy:phyllobacteriaceae gene:fhmpcd1 | 1 | 1 | поиск по более крупным таксонам не показал большего количества результатов, данный ген кодирует один и тот же белок у Nitratireductor basaltis и Mesorhizobium japonicum |
| taxonomy:mesorhizobium gene:fhmpcd1 | 1 | 0 | в пределах рода данный ген встречается только у одного организма - Mesorhizobium japonicum |
| taxonomy:bacteria goa:("vitamin b6 catabolic process") | 9 | 0 | помимо рода Mesorhizobium, среди бактерий белки, участвующие в катаболизме витамина В6, обнаружены у Microbacterium luteolum и Escherichia coli |
| taxonomy:mesorhizobium goa:("vitamin b6 catabolic process") | 6 | 0 | в рамках данного рода обнаружено 6 белков, участвующих в катаболизме витамина В6 |
| goa:("vitamin b6 catabolic process") NOT taxonomy:bacteria | 7 | 35 | были найдены белки, участвующие в процессах катаболизма витамина В6 у представителей других царств |
| goa:("vitamin b6 catabolic process") NOT taxonomy:bacteria cofactor:(chebi:"Mg(2+) [18420]") | 4 | 0 | найденные белки были проверены на наличие сайта связывания с НАД, что не привело к положительному результату, но было выяснено, что для части из них характерно наличие кофактора |
В запись о данном белке в Uniprot изменения были внесены в общей сложности 132 раза, в том числе 4 раза с момента внесения его в Swiss-Prot. В базу данных Swiss-Prot он был внесен 17.06.2020, до этого имевшаяся о нем информация обновлялась автоматически.
Список использованной литературы
- Complete genome structure of the nitrogen-fixing symbiotic bacterium Mesorhizobium loti - T Kaneko, Y Nakamura, S Sato, E Asamizu, T Kato, S Sasamoto, A Watanabe, K Idesawa, A Ishikawa, K Kawashima, T Kimura, Y Kishida, C Kiyokawa, M Kohara, M Matsumoto, A Matsuno, Y Mochizuki, S Nakayama, N Nakazaki, S Shimpo, M Sugimoto, C Takeuchi, M Yamada, S Tabata - DNA Res. (2000) 7:331-338.
- Gene identification and characterization of 5-formyl-3-hydroxy-2-methylpyridine 4-carboxylic acid 5-dehydrogenase, an NAD+-dependent dismutase. - Yokochi N., Yoshikane Y., Matsumoto S., Fujisawa M., Ohnishi K., Yagi T. - J. Biochem. 145:493-503(2009) [PubMed] [Europe PMC]
- Crystal structure of 5-formyl-3-hydroxy-2-methylpyridine 4-carboxylic acid 5-dehydrogenase, an NAD-dependent dismutase from Mesorhizobium loti. - Mugo A.N., Kobayashi J., Mikami B., Yoshikane Y., Yagi T., Ohnishi K. - Biochem. Biophys. Res. Commun. 456:35-40(2015) [PubMed] [Europe PMC]
- Mesorhizobium japonicum в базе данных BacDive
- Умаров М.М. Азотфиксация в ассоциациях организмов//Проблемы агрохимии и экологии. - 2009 - №2 - С.22-26