Сборка de novo
Код доступа чтений, из которых мне нужно было составить сборку, - SRR4240361. Архив с чтениями был скачан в рабочую директорию с помощью следующей команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/001/SRR4240361/SRR4240361.fastq.gz
Далее нужно было обработать исходные чтения для дальнейшего преобразования их в сборку генома. Сначала были удалены возможные остатки адаптеров: сначала все файлы из директории с последовательностями адаптеров собраны в один, затем на основе этого чения были "очищены" с помощью trimmomatic. При записи команды нужно указать, что чтения одноконцевые (SE), задать исходный и конечный файл и запустить "step" ILLUMINACLIP, которому в свою очередь подается файл с последовательностями адаптеров. Далее с правых концов чтений были удалены нуклеотиды с качеством ниже 20 ("step" TRAILING команды trimmomatic), а также были удалены чтения длиной менее 32 нуклеотидов (MINLEN:32).
cat /mnt/scratch/NGS/adapters/* >> adapters.fa
java -jar /usr/share/java/trimmomatic.jar SE SRR4240361.fastq.gz trimmed.fastq.gz -trimlog trim.log ILLUMINACLIP:adapters.fa:2:7:7
java -jar /usr/share/java/trimmomatic.jar SE trimmed.fastq.gz trimmed2.fastq.gz -trimlog trim2.log TRAILING:20 MINLEN:32
В результате запуска двух команд trimmomatic были получены следующие результаты:
Input Reads: 7272621 Surviving: 7238089 (99.53%) Dropped: 34532 (0.47%)
(после удаления адаптеров размер файла уменьшился с 193M до 192M)
Input Reads: 7238089 Surviving: 6834335 (94.42%) Dropped: 403754 (5.58%)
(после удаления коротких чтений размер конечного файла стал равен 178M)
Из них можно следать вывод, что адаптеров в чтениях уже практически не было, так как в первый раз "отфильтровалось" всего 0.47% чтений. В связи с несоответствием по длине или качеству также удалена достаточно малая часть, значит, исходные данные можно охарактеризовать высоким качеством.
Далее были запущены команды velveth и velvetg, первая из которых подготавливает k-меры (k=31) на основе исходного файла и создает папку с данными velveth, на которой далее запускается velvetg и осуществляет сборку на основе k-меров. Файл velvet.log содержит информацию о полученной сборке, в частности значение N50: 25683
velveth velveth 31 -fastq.gz trimmed2.fastq.gz -short
velvetg velveth &> velvet.log
В результате работы velvetg были получены файлы stats.txt и contigs.fa. Первый содержит информацию о длине и покрытии каждого контига и другие количественные данные, второй - длину, покрытие и последовательность каждого контига. На основе этих файлов можно выделить 4 контига наибольшей длины (в задании речь шла о трех, но один из контигов в процессе работы с BLAST не выровнялся на хромосому бактерии, и мне показалось, что взять следующий по длине контиг более логично, чем запускать программу с разными параметрами для разных контигов):
| ID | длина | покрытие | fasta |
|---|---|---|---|
| 6 | 49238 | 26.66 | contig 1 |
| 2 | 45555 | 26.45 | contig 2 |
| 34 | 43866 | 23.52 | contig 3 |
| 11 | 37067 | 28.38 | contig 4 |
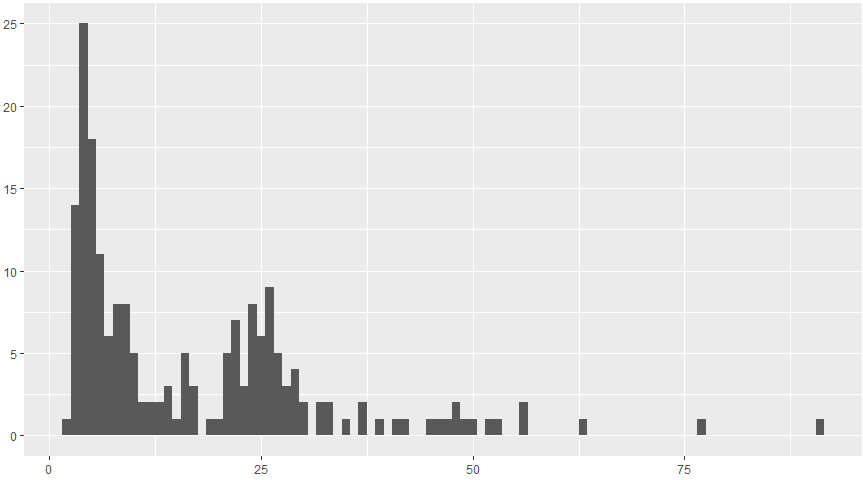
Если рассматривать статистические данные на основе файла stats.txt, возникает проблема наличия в нем очень коротких "контигов" вплоть до одного нуклеотида, которые мешают увидеть общую картину. После преобразования данных (удаления строк с контигами длины меньше 32) они значительно сокращаются. Среднее значение покрытия составляет около 17, максимальное и минимальное - 90.7 и 2.2 соответственно. Гистограмма, примерно отражающая распределение числа контигов с разным покрытием после обработки данных, представлена рядом на рисунке.
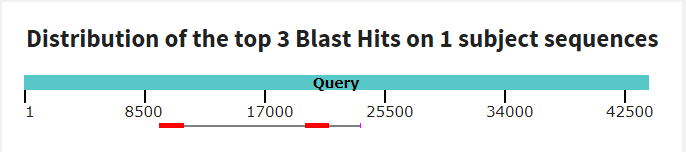
Далее было произведено сопоставление контигов с последовательностью заданной хромосомы. Как было уже упомянуто, для второго по длине контига значимых участков сходства с данной хромосомой найдено не было, поэтому далее будет представлена информация о результатах работы megablast с контигами 1, 3 и 4. Запуск blastn с контигом 2 даёт в результате выравнивание с идентичностью 70.81% и покрытием 61%, значит, вероятно, речи о глобальных ошибках прочтения не идёт, просто данный участок характеризуется большим количеством точечных мутаций, вследствие чего не срабатывает алгоритм megablast.
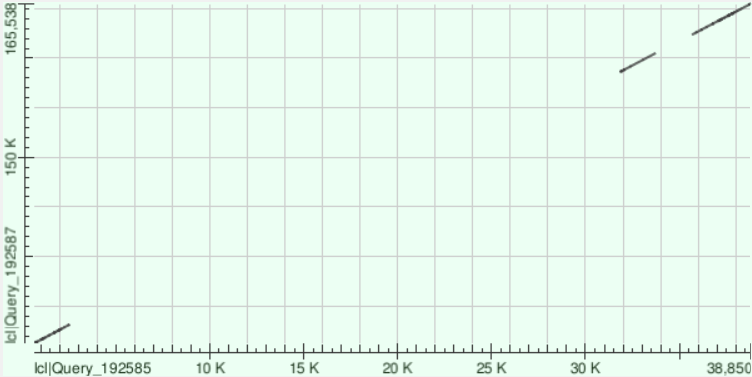
Для первого контига был получен следующий результат. Он имеет три общих участка с хромосомой: 158589-160512 (15 гэпов, 366 однонуклеотидных различий), 162363-165538(95 гэпов, 711 однонуклеотидных различий), 131249-133142(76 гэпов, 452 однонуклеотидных различия). В целом локальное выравнивание достаточно хорошее, идентичность 80.26% при пократии 14%. Расположение выравнянных участков в контиге видно из схемы ниже: это концевые участки. DotPlot также показывает, что в начале и в конце прочтения находятся высококонсервативные районы, а между ними совпадающие элементы отсутствуют, что можно охарактеризовать как крупную делецию.
Для двух других контигов, как видно из соответствущих графиков, имеем похожую ситуацию: несколько консервативных участков, между которыми, вероятно, крупные делеции. Вероятно, такое явление могло возникнуть вследствие ошибки в процессе сборки.
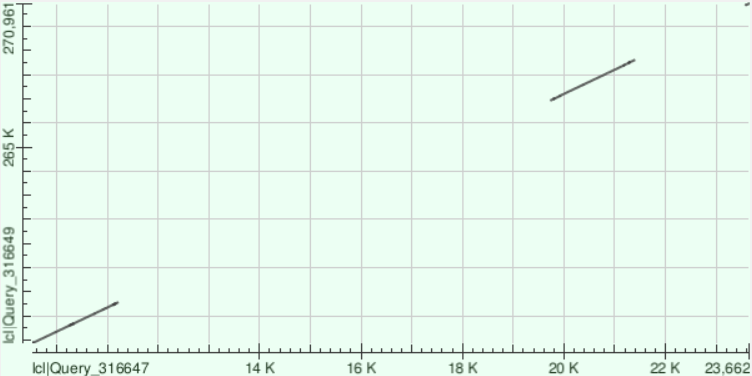
Для контига 3 результат следующий: участки 266926-268617(42 гэпа, 104 однонуклеотидных различия), 256868-258570(40 гэпов, 373 различия) и короткий 270883-270961 (1 гэп, 4 различия). Можно отметить высокую консервативность отдельных фрагментов: идентичность составляет 91.44% при покрытии 7%.
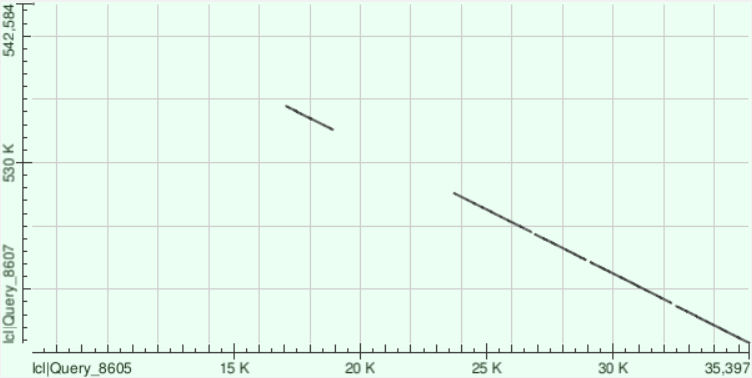
В результате для контига 4 видим достаточно длинный участок совпадения, прерываемый короткими делециями. Это участки 524536-527614 (78 гэпов, 689 различий), 515741-518675(114 гэпов, 616 различий), 518938-522162(130 гэпов,768 различий), 532575-534461 (83 гэпа, 379 различий), 522296-524368(85 гэпов, 454 различия), 542494-542584(5 гэпов, 4 различия). Для этого контига имеем наибольшее покрытие - 35%, но не такую высокую идентичность - 75.41%. Примечательно, что точки, отображающие идентичность позиций в двух последовательностях, лежат не на прямой, проходящей через точку пересечения осей, или параллельной ей. Это свидетельствует о том, что, вероятно, последовательность контига представляет собой обратную (комплементарную) последовательность.