Комплексы ДНК-белок
Задание 1. Предсказание вторичной структуры заданной тРНК
Упражнение 1
Для предсказания инвертированных участков тРНК с помощью программы einverted была скачана последовательность заданной тРНК в формате fasta и выполнен следующий код:
einverted rcsb_pdb_1EXD.fasta
При значении параметра Minimum score threshold, равном 50 по умолчанию, пары не были найдены, поэтому оно было изменено на 0. В результате был получен файл, содержащий 6 найденных пар оснований, соответствующих акцепторному стеблю тРНК, что согласуется с результатами работы find_pair, представленными в следующем файле, но позволяет характеризовать einverted как менее эффективную программу для исследования вторичной структуры РНК.
Упражнение 2
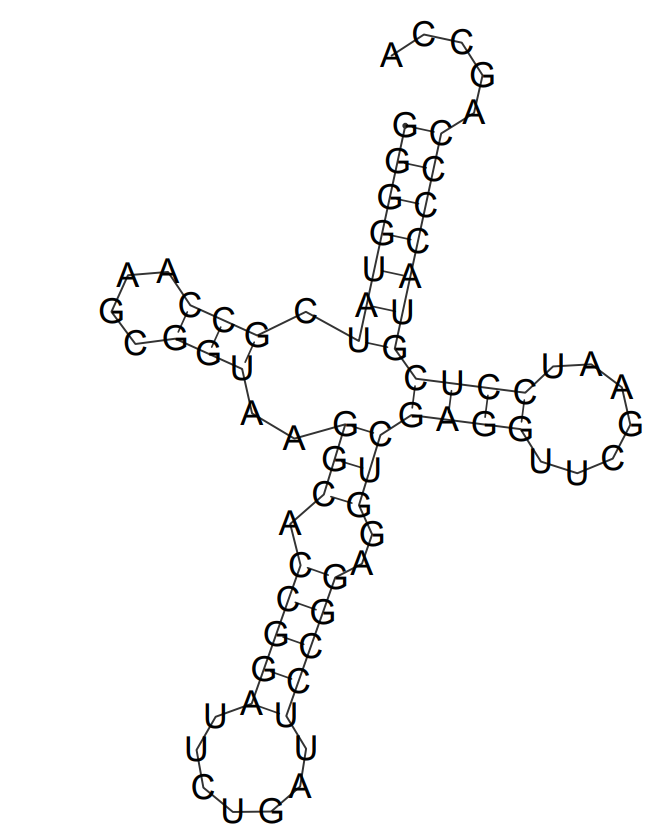
Далее было выполнено предсказание структуры тРНК по алгоритму Зукера. Синтаксис:
export PATH=${PATH}:/home/preps/golovin/progs/bin
cat rcsb_pdb_1EXD.fasta | RNAfold --MEA
ps2pdf 1EXD_1_Chain_dp.ps
Полученную структуру можно просмотреть в файле по ссылке, либо на рисунке, представленном на данной странице. Результаты работы RNAfold частично согласуются с результатами find_pair, но есть различия: акцепторный стебель содержит те же 6 пар и дополнительно неканоническую пару U-G, D-стебель в обоих предсказаниях содержит 3 пары, но в предсказании find_pair размер D-петли на несколько нуклеотидов больше, поэтому можно видеть сдвиг в номерах "спаренных" нуклеотидов. Также результат алгоритма Зукера содержит неканоническую пару U-G, что отражено в таблице. Т-стебель практически полностью совпадает в выдаче обеих программ, но по алгоритму Зукера она получается короче на один нуклеотид. Заметны различия в строении антикодонового стебля: по результату работы find_pair, он состоит из 4 канонических и 4 неканонических пар, следующих в структуре непрерывно, по алгоритму RNAfold же наблюдается выпетливание и всего одна неканоническая пара. Часть нуклеотидов, отнесенных find_pair к D-стеблю, отнесена к антикодоновому. Таким образом, можно отметить высокую точность алгоритма Зукера и его эффективность в предсказании структур тРНК.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-902-907-3' 5'-966-971-3' всего 6 пар, все канонические | 5'-902-907-3' 5'-966-971-3' всего 6 пар, все канонические | 5'-902-908-3' 5'-965-971-3' всего 7 пар, 6 канонических и неканоническая U-G |
| D-стебель | 5'-910-912-3' 5'-923-925-3' всего 3 пары, канонические | - | 5'-910-912-3' 5'-917-919-3' всего 3 пары, 2 канонические и одна неканоническая U-G |
| T-стебель | 5'-949-953-3' 5'-961-965-3' всего 5 пар, канонические | - | 5'-950-953-3' 5'-961-964-3' всего 4 пары, все канонические |
| Антикодоновый стебель | 5'-937-944-3' 5'-926-933-3' всего 8 пар, из них 4 неканонические, в том числе пары A-A и U-U | - | 5'-922-924, 926-930-3' 5'-945-947, 938-942-3' всего 8 пар, 7 канонических и одна неканоническая G-U |
| Общее число канонических пар нуклеотидов | 22 | 6 | 19 |
Задание 2.Поиск ДНК-белковых контактов в заданной структуре
Упражнение 1
С помощью программы Jmol можно выделить определенные множества атомов для расчета в дальнейшем расстояний между ними и потенциальных контактов молекул белка и ДНК. По следующим ссылкам можно увидеть скрипт для задания множеств атомов и скрипт для демонстрации ДНК и выделенных атомов.
Упражнение 2
В рамках данного упражнения были проанализированы ДНК-белковые контакты разной природы. Определив атомы кислорода и азота как полярные, а серы, углерода и фосфора как неполярные, можно найти количество соответствующих контактов с учетом вхождения этих атомов в тот или иной структурный элемент нуклеотида. По ссылке доступен скрипт, позволяющий определить количество полярных и неполярных контактов ДНК с белком. По результатам работы программы, представленным в таблице, можно отметить, что большинство ДНК-белковых контактов являются неполярными и образуются между неполярными атомами белка и дезоксирибозой ДНК. Также большое количество потенциальных контактов можно обнаружить между белком и остатком фосфорной кислоты. Количество контактов с азотистыми основаниями невелико и примерно одинаково для большой и малой бороздки, эти контакты с высокой долей вероятности являются специфическими и обеспечивают распознавание белком конкретной ДНК по ее последовательности.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 6 | 137 | 143 |
| остатками фосфорной кислоты | 23 | 30 | 53 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 9 | 11 |
| остатками азотистых оснований со стороны малой бороздки | 2 | 7 | 9 |
Упражнение 3
С помощью программы nucplot была получена схема ДНК-белковых контактов для заданной структуры. В ней отражены возможные контакты аминокислот с азотистыми основаниями, дезоксирибозой и остатками фосфорной кислоты. По ссылке можно просмотреть файл со схемой. Для данной структуры можно отметить симметричность, так как белок состоит из двух идентичных субъединиц, и аминокислоты с одинаковыми номерами цепей А и В образуют идентичные контакты с разными цепями ДНК. Помимо "прямых" контактов отмечены также контакты, осуществляющиеся через посредство молекул воды.
Упражнение 4
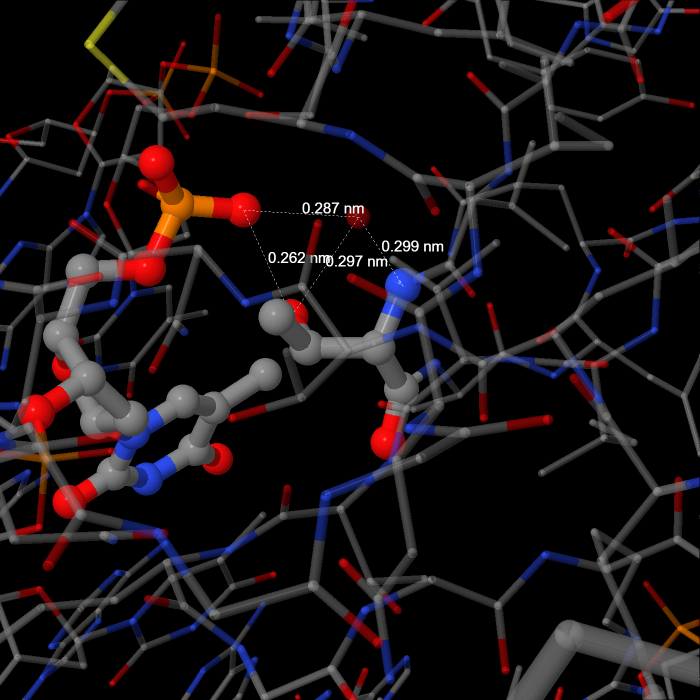
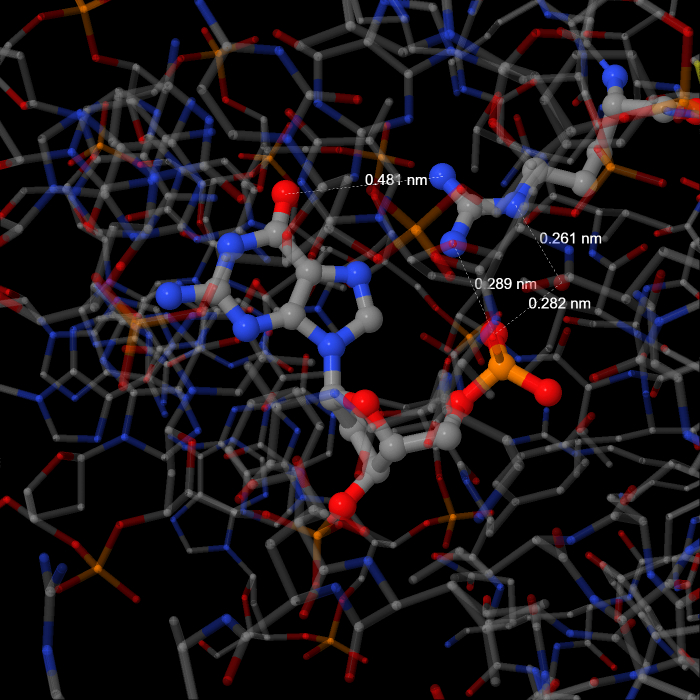
Так как субъединицы белка идентичны, логично рассмотреть контакты только одной из них. В числе остатков, образующих несколько контактов, можно выделить Thr194 цепи А, образующий две полярные связи (через азот N и кислород OG1) через посредство молекулы воды и одну напрямую через кислород OG1 с фосфатной группой, соединяющей 503 и 504 нуклеотиды. В качестве одного из значимых для распознавания белком структуры ДНК остатков можно рассмотреть Arg67 цепи А, кторый образует контакт с гуанином 517-го нуклеотида через аминогруппу. Также он образует контакты через аминогруппу и через азот NE через посредство молекулы воды с остатком фосфорной кислоты, соединяющим 516 и 517 нуклеотиды. С помощью программы Jmol были получены изображения этих контактов, представленные на рисунках ниже.