Выравнивание геномов
Задание 1. Выбор прокариотического организма с полностью собранным геномом
В качестве объектов для исследования генома были выбраны 5 штаммов Lactobacillus acidophilus: DSM 20079 (CP020620.1), YT1 (CP025200.1), LA-G80-111 (CP054559.1), FSI4 (CP010432.1), АТСС 53544 (CP022449.1). Lactobacillus acidophilus - часть микрофлоры желудочно-кишечного тракта человека, используется в промышленноси в производстве йогурта, сыра и других ферментированных продуктов. Организм был выбран таким образом с расчетом на качественную сборку и аннотацию, так как представители рода Lactobacillus давно известны и активно используются в биотехнологии.
Задание 2. Построение нуклеотидного пангенома с помощью NPG-explorer
Для анализа геномов был составлен следующий входной файл, в котором перечислены название и АС штаммов из GenBank. Далее были запущены команды NPG-explorer, с их протоколами выполнения можно ознакомиться по ссылкам: Prepare, MakePangenome. Команды -g npge.conf, Examine и PostProcessing не выдают ничего на stdout и stderr. Перед запуском MakePangenome в файле npge.conf значение параметра WORKERS (число задействованных процессоров) было изменено на 1, значение MIN_IDENTITY (минимальная доля консервативных позиций), согласно рекомендации Examine - на 0.894.
В результате были получены следующие файлы:
- дерево геномов
- описание генов
- описание мутаций в блоках
- консенсусы блоков
- описание блоков
- выравнивание блоков
- таблиц характеристик блоков
Задание 3. Описание стабильного ядра нуклеотидного пангенома
Стабильное ядро нуклеотидного пангенома включает 140 s-блоков (8809250 нуклеотидов), процент нуклеотидов в ядре от общего числа всех нуклеотидов составляет 87.4%, от общей длины блоков - 79.74%. Процент консервативных колонок в объединенном выравнивании s-блоков составляет 99.4472%. Минимальная длина фрагмента (задана параметром MIN_LENGTH) составляет 100 нуклеотидов, максимальная - 168701.
Задание 4. Описание крупных делеций
Файл pangenome.info был проанализирован с помощью Excel, в результате чего был найден h-блок наибольшей длины - h4x14073 входит в g-блок g5x609709 и включает 14073 нуклеотида. Этот блок подтверждает делецию в геноме штамма YT1, так как присутствует в остальных четырех. Имена закодированных в этом блоке генов, делетированных у штамма YT1:
- HUE89_02490 tRNA-Thr (73 пары нуклеотидов)
- CGZ81_04110 restriction endonuclease (4506 пар нуклеотидов)
- CGZ81_04115 hypothetical protein (1749 пар нуклеотидов)
- CGZ81_04120 DNA methyltransferase (870 пар нуклеотидов)
- CGZ81_04125 methylase (684 пары нуклеотидов)
- CGZ81_04130 hypothetical protein (702 пары нуклеотидов)
- CGZ81_04135 hypothetical protein (351 пара нуклеотидов)
- CGZ81_04140 chromosome partitioning protein ParA (1128 пар нуклеотидов
- CGZ81_04145 hypothetical protein (414 пар нуклеотидов)
- CGZ81_04150 replication initiation protein (1083 пары нуклеотидов)
- CGZ81_04155 hypothetical protein (198 пар нуклеотидов, есть перекрывание длиной 20 нуклеотидов с предыдущим )
- CGZ81_04160 integrase (1203 пары нуклеотидов)
Идентификаторы генов взяты из генома штамма АТСС 53544 и отличаются у других, но гены полностью совпадают. Часть из них представляют собой не аннотированные гены, а только предполагаемые участки, которые могут ими являться (обозначены как hypothetical protein). В числе кодируемых белков эндонуклеаза рестрикции, белок, распределяющий хромосомы при делении клетки, инициатор репликации и интеграза. Возможно, речь может идти о репликационном аппарате вируса, так как интеграза обеспечивает вставку вирусной ДНК, эндонуклеаза рестрикции - двуцепочечный разрыв ДНК для последующей вставки или перекомбинации, другие ферменты связаны также с удвоением ДНК и делением клетки. Наличие метилазы и ДНК-метилтрансферазы нетипично для бактерии, так как метилирование ДНК является способом регуляции экспрессии генов эукариот. Согласно найденному источнику, метилирование ДНК у бактерий лежит в основе явления хозяйской рестрикции-модификации, которое заключается в том, что выращенный в клетках штамма бактериофаг приобретает специфичность - способность заражать только конкретного хозяина. Однако, если указанный участок попал в геном бактерии вследствие встречи с бактериофагом (тогда неаннотированные гены, вероятно, кодируют белки бактериофага), более ожидаемо было бы увидеть вставку в одном из штаммов, а не делецию.
Можно отметить, что все делеции, кроме двух (блоки h4x171 и h4x307) произошли в геноме YT1, исключения - в геноме DCM 20079. Делеция, на которую указывает блок h3x9393, произошла одновременно в геноме YT1 и DCM 20079. Блок длиной 307 нуклеотидов содержит ген CGZ81_01975 tRNA-Lys длиной 73 нуклеотида, блок длиной 171 нуклеотид - фрагмент гена CGZ81_09435 transposase (идентификаторы из генома АТСС 53544).
Рассмотрим гены блока h3x9393, делетированные у двух штаммов: (идентификаторы из генома ATCC 53544)
- CGZ81_03190 site-specific integrase (1035 пары нуклеотидов)
- CGZ81_03195 hypothetical protein (189 пар нуклеотидов)
- CGZ81_03200 replication protein (1101 пара нуклеотидов)
- CGZ81_03205 hypothetical protein (531 пара нуклеотидов)
- CGZ81_03210 cell division protein (1077 пар нуклеотидов, перекрывается с предыдущим)
- CGZ81_03215 hypothetical protein (312 пар нуклеотидов, перекрывается с предыдущим)
- CGZ81_03220 hypothetical protein (885 пар нуклеотидов)
- CGZ81_03225 methylase (2820 пар нуклеотидов)
- CGZ81_03230 head protein (372 пары нуклеотидов)
- CGZ81_03235 hypothetical protein (231 пара нуклеотидов)
Данный набор генов также напоминает вставку из вирусного генома: есть белок бактериофага head protein, сайт-специфичная интеграза для вставки в геном, белки, отвечающие за репликацию ДНК и деление клетки, а также метилирование ДНК. Вероятно, общий предок выбранных штаммов был "заражен" бактериофагом, но у части потомков этот фрагмент генома был вновь удален. Если обратить внимание на дерево геномов, видно, что штаммы YT1 и DCM 20079, у которых данный фрагмент не обнаруживается, находятся на одной "ветке" и отделились позже остальных, что подтверждает гипотезу.
Задание 5. Описание перестановок синтений
Из представленной выше схемы видно, что порядок блоков в геноме штамма YT1 на данном участке полностью не соответствует порядку блоков в геномах остальных штаммов. Такой значительный "беспорядок" мог бы объясняться ошибкой при расшфровке генома и определении ориджина репликации, но здесь видно, что часть блоков (q5x108, q5x107, q5x1906) расположены в геноме YT1 ближе к началу, чем для остальных штаммов, тем временем другие (q5x26904, q5x09709) - ближе к концу, порядок блоков полностью другой. Вероятно речь идет о множественных транслокациях. Отличие этого генома от остальных было заметно еще на этапе выполнения задания 4, когда в нем было обнаружено наибольшее количество делеций. Это отражено и в дереве геномов, где YT1 имеет самую длинную "ветку", что указыает на то, что после расхождения в эволюции предков YT1 и DCM 20079 (для них показана наибольшая близость, что можно подтвердить и наличием общей делеции) до формирования YT1 произошло несопоставимо больше эволюционных событий, чем для всех остальных штаммов. При этом можно отметить, что родство штаммов при построении дерева, по-видимому, больше определяется сходством блоков, а не их порядком, иначе было бы ожидаемо увидеть YT1 на отдельной "ветке" как относительно более далекого родственника.
Задание 6. Ошибки аннотации гена
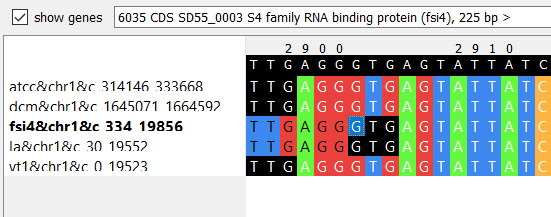
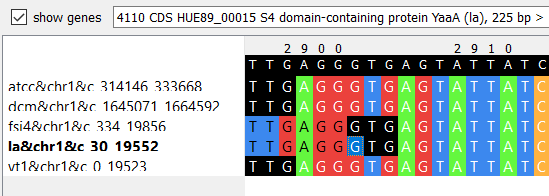
Предполагаемая ошибка аннотации найдена в блоке s5x19525 глобального блока g5x184060. Можно видеть, что при очень высокой доле идентичных нуклеотидов (на рассматриваемом участке различия отсутствуют) на разных фрагментах одного блока обнаруживаются разные гены. В штаммах ATCC 53544 и DCM 20079 на рассматриваемом участке находится первый кодон гена РНК-связывающего белка, в то время как у штаммов FSI4 и LA-G80-111 ген не определяется, а у YT1 - ген S4 домен-содержащего белка, который по функции также является РНК-связывающим. На 6 нуклеотидов дальше начинаются гены, детектированные у штаммов FSI4 и LA-G80-111 - также гены S4 РНК-связывающих белков, длина этих генов ровно на 6 нуклеотидов меньше указанных ранее. Таким образом, если ошибка имела место, она не повлияла на распознавание функции кодируемого белка, и данной выборки недостаточно, чтобы сказать, какой вариант из найденных скорее является ошибочным.
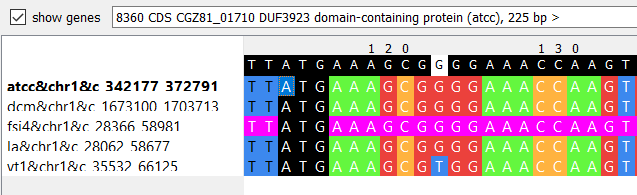
Далее на предмет ошибок аннотации был исследован блок s5x30625 того же глобального блока. Например, на указанном выше участке видно, что на четырех из пяти фрагментах геномов найден ген DUF3923 домен-содержащего белка, в то время как в геноме DCM 20079 на этом же участке при полном соответствии последовательности ген найден, но белок не определен. С высокой вероятностью можно предполагать, что здесь должен находиться тот же белок, что и в родственных штаммах.
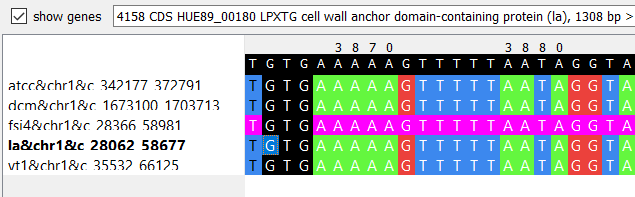
В этом же блоке представлена обратная ситуация: в четырех из пяти фрагментах найден ген, но не определен белок, но в геноме LA-G80-111 на этом участке закодирован белок, заякоривающийся в клеточной стенке. В данном случае может иметь место ошибочное распознавание белка либо, аналогично предыдущему случаю, некачественное распознавание генов в четырех штаммах.
Выводы
В результате проделанной работы можно отметить в первую очередь впечатляющий объем функционала программы, которая позволяет осуществить полный автоматизированный анализ геномов. Среди выбранных геномов, как показали результаты, есть 4 очень близких и имеющих идентичный набор и порядок блоков, и один более далекий "родственник", в геноме которого в результате множественных эволюционных событий блоки поменяны местами, но сохраняют идентичность. Доля длины стабильных блоков по отношению к общей составляет почти 80%, что также указывает на высокую гомологию. По набору блоков можно судить о количестве делеций и вставок, причем для выбранных 5 геномов наиболее характерна ситуация делеции в одном из геномов (чаще всего YT1, который также отличается множественными транслокациями) или вставки также в один из геномов (вставками условно считаем u-блоки, наибольшее их число аналогично у YT1). Перестройки генома предположительно могут быть связаны с встраиванием в ДНК бактерии генов бактериофагов.