Поиск ортологов и паралогов
Составление списка гомологичных белков, включающих паралоги
Для поиска гомологов белка CLPX_ECOLI в заданных организмах была создана локальная база данных, содержащая иx протеомы. Эта база данных была подана на вход программе blastp с порогом на e-value 0.001, далее из выдачи программы были выбраны идентификаторы белков, и соответствующие последовательности собраны из базы данных в новый файл с помощью seqret. Все действия от создания базы данных до выравнивания найденных последовательностей выполняются следующим скриптом:
cat *.fasta > proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out proteomes
blastp -query clpx_ecoli.fasta -db proteomes -evalue 0.001 -out clpx.blast
grep '|' clpx.blast | grep -v '>'| grep -v 'Query'| awk '{print $1}' | awk -F '|' '{print $3}' > names.txt
while read line; do echo 'proteomes.fasta:'$line; done < names.txt > myprots.list
seqret @myprots.list myprots.fasta
muscle -in myprots.fasta -out myprotsalign.fasta
В результате работы blastp был получен следующий результат, содержащий приведенный ниже список белков:
| Sequence | Score | E-value |
|---|---|---|
| sp|Q820F8|CLPX_STRAW ATP-dependent Clp protease ATP-binding sub... | 535 | 0.0 |
| sp|A1TCB3|CLPX_MYCVP ATP-dependent Clp protease ATP-binding sub... | 523 | 0.0 |
| sp|P9WPB9|CLPX_MYCTU ATP-dependent Clp protease ATP-binding sub... | 519 | 0.0 |
| sp|Q9CBY6|CLPX_MYCLE ATP-dependent Clp protease ATP-binding sub... | 519 | 0.0 |
| sp|Q6NFU7|CLPX_CORDI ATP-dependent Clp protease ATP-binding sub... | 518 | 0.0 |
| sp|Q6AFZ6|CLPX_LEIXX ATP-dependent Clp protease ATP-binding sub... | 509 | 1e-180 |
| sp|B0RAS4|CLPX_CLAMS ATP-dependent Clp protease ATP-binding sub... | 491 | 2e-173 |
| sp|Q8G5R1|CLPX_BIFLO ATP-dependent Clp protease ATP-binding sub... | 432 | 9e-150 |
| tr|Q8G871|Q8G871_BIFLO Protease OS=Bifidobacterium longum (stra... | 51.2 | 7e-07 |
| tr|Q6NFB1|Q6NFB1_CORDI ATP-dependent Clp protease ATP-binding s... | 45.8 | 3e-05 |
| sp|Q8G6B7|RUVB_BIFLO Holliday junction ATP-dependent DNA helica... | 42.7 | 2e-04 |
| tr|Q6ACQ0|Q6ACQ0_LEIXX ATP-dependent zinc metalloprotease FtsH ... | 43.1 | 2e-04 |
| tr|Q8G3S2|Q8G3S2_BIFLO ATP-dependent zinc metalloprotease FtsH ... | 43.1 | 2e-04 |
| tr|A1TG29|A1TG29_MYCVP ATPase AAA-2 domain protein OS=Mycolicib... | 43.1 | 2e-04 |
| tr|Q82QV8|Q82QV8_STRAW Putative AAA family ATPase OS=Streptomyc... | 41.6 | 4e-04 |
| tr|Q82EE9|Q82EE9_STRAW ATP-dependent zinc metalloprotease FtsH ... | 41.6 | 6e-04 |
| tr|A1TG43|A1TG43_MYCVP ATP-dependent zinc metalloprotease FtsH ... | 41.6 | 6e-04 |
| tr|Q82EB8|Q82EB8_STRAW Putative ATP-dependent Clp protease OS=S... | 41.6 | 7e-04 |
| sp|P9WQN3|FTSH_MYCTU ATP-dependent zinc metalloprotease FtsH OS... | 41.2 | 7e-04 |
| tr|B0RHW4|B0RHW4_CLAMS ATP-dependent zinc metalloprotease FtsH ... | 41.2 | 8e-04 |
Уже из этих результатов видно, что в первую очередь были найдены гомологи с такой же функцией - АТФ-связывающие субъединицы АТФ-зависимой протеазы Clp всех рассматриваемых организмов. У этих находок высокие значения Score и ничтожно малый E-value. Далее идут белки с другими функциями, значения Score для которых уже на порядок ниже. Самая многочисленная группа среди них - АТФ-зависимые цинк-металлопротеазы.
Реконструкция и визуализация
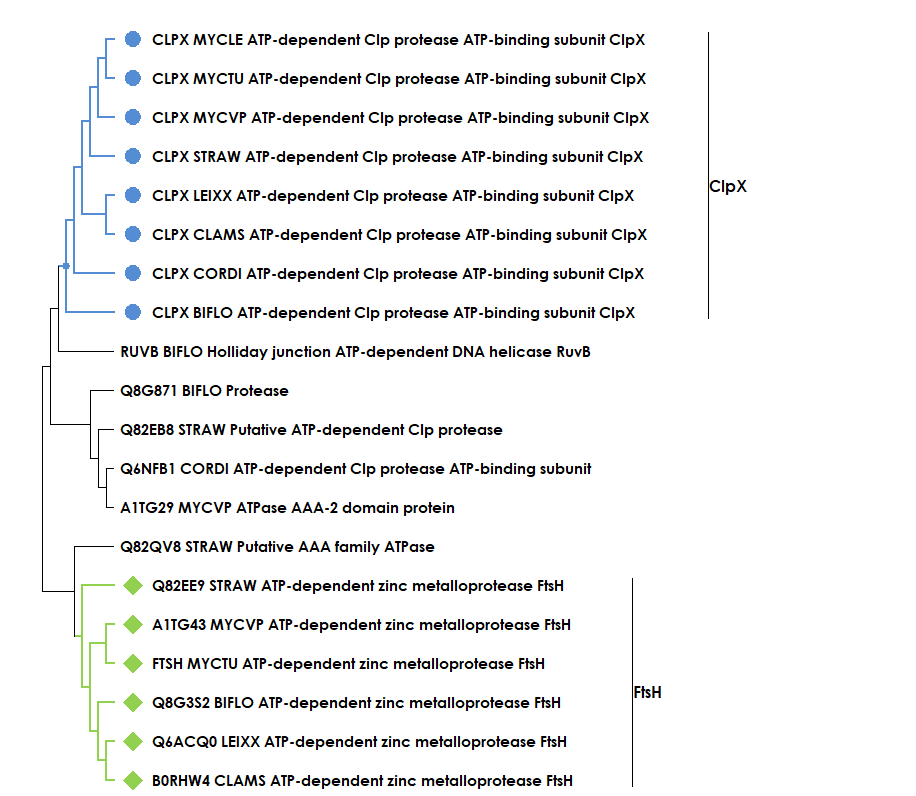
Реконструкция филогении была произведена с помощью программы MEGA с алгоритмом Maximal Likehood. Было получено следующее дерево, визуализация которого представлена ниже.
Из найденных белков паралогами являются, к примеру, CLPX_MUCTU и FTSH_MUCTU, CLPX_LEIXX и Q6ACQ0_LEIXX, CLPX_CLAMS и B0RHW4_CLAMS. Ортологами являются CLPX_BIFLO и CLPX_CORDI, A1TG43_MYCVP и FTSH_MUCTU, Q6ACQ0_LEIXX и B0RHW4_CLAMS. Можно выделить две большие группы ортологов - АТФ-связывающие субъединицы Clp протеаз (ClpX) и АТФ-зависимые цинковые металлопротеазы (FtsH). Для первой группы можно отметить, что в неё вошли белки всех рассматриваемых организмов, но филогения белков не в точности соответствует филогении бактерий. Сохраняются ветви, выделяющие LEIXX и CLAMS, а также MYCTU, MYCLE и MYCVP. Во второй группе присутствуют только 6 из 8 организмов, но филогения сохраняется: выделяются ветви CLAMS, LEIXX и BIFLO; MYCVP и MYCTU и ветвь, выделяющая отдельно STRAW.