Сигналы и мотивы
Задание 1. Сигнал митохондриальной локализации.
Сигнал митохондриальной локализации - закодированный в геноме короткий пептид длиной от 15 до 70 аминокислот, среди которых чередуются гидрофобные и положительно заряженные, образуя так называемую амфипатическую спираль, у которой одна сторона гидрофобная, а другая - гидрофильная. Эта последовательность соответствует N-концу белка и oбеспечивает его транспорт в митохондрии. Специфичный транспорт внутрь митохондрии осуществляется разнообразными путями за счет связывания сигнального участка с рецепторами на поверхности мембраны митохондрии. Как только белок-предшественник с сигналом митохондриальной локализации достигает митохондриального матрикса, этот короткий пептид отщепляется митохондриальной процессинговой пептидазой. Сигнал высокоэффективен, молекулярно-биологические эксперименты показывают, что белки, в последовательность которых искусственно вносится сигнал молекулярной локализации, практически в полной мере переходят в митохондрии.
Источники:
- Bolender N, Sickmann A, Wagner R, Meisinger C, Pfanner N. Multiple pathways for sorting mitochondrial precursor proteins. EMBO Rep. 2008 Jan;9(1):42-9
- Natalia Bolender, Albert Sickmann, Richard Wagner, Chris Meisinger, Nikolaus Pfanner. Multiple pathways for sorting mitochondrial precursor proteins. EMBO Reports (2008)9:42-49
Задание 2. Позиционная весовая матрица для последовательности Козак человека.
Чтобы получить позиционную весовую матрицу для последовательности Козак человека, были выбраны 100 случайных генов, для которых известны координаты стартовых кодонов. Все гены находятся на 1 хромосоме и на прямой цепи для упрощения работы. С помощью скрипта (основа скрипта взята из подсказок к заданию) были вырезаны нужные участки хромосомы длиной 13 нуклеоидов: 7 позиций до ATG и 3 после. Файл coords.txt, который принимается на вход скриптом, содержит координаты генов в форме 1:thickStart:thickEnd:1. Полученные последовательности далее использовались для построения позиционной матрицы: первые 40 генов - обучение, остальные 60 - положительный контроль. В базе данных был найден GC-состав 1 хромосомы (42,3%), на основании которого определены ожидаемые частоты каждого нуклеотида. Псевдоотсчеты выбраны одинаковыми для каждого основания (0.1). Таким образом, общая формула в ячейке для основания b в позиции j последовательности: ln(([N(b,j]+0.1]/(N+0.4))/p(b)), где р(b) = 0.21 для G и C и 0.29 для А и Т.
В результате вышеописанных вычислений получена следующая матрица PWM:
| основание | ожидаемая частота | псевдоотсчёты | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | 0,29 | 0,1 | -2,3656 | -0,2527 | -0,8317 | -0,6527 | -0,1484 | -0,3691 | -0,5009 | 1,2304 | -4,7635 | -4,7635 | -0,6527 | -0,2527 | -1,719 |
| T | 0,29 | 0,1 | -0,3691 | -0,5009 | -1,3296 | -1,3296 | -1,719 | -0,8317 | -1,05 | -4,7635 | 1,2304 | -4,7635 | -0,3691 | -0,2527 | -0,1484 |
| G | 0,21 | 0,1 | 0,7009 | 0,1744 | 0,5765 | 0,5765 | 0,8625 | 0,0701 | -0,5089 | -4,4408 | -4,4408 | 1,5532 | 0,5765 | 0,1744 | 0,7009 |
| C | 0,21 | 0,1 | 0,508 | 0,508 | 0,7009 | 0,6406 | -0,0463 | 0,7577 | 1,044 | -4,4408 | -4,4408 | -4,4408 | 0,2688 | 0,355 | 0,2688 |
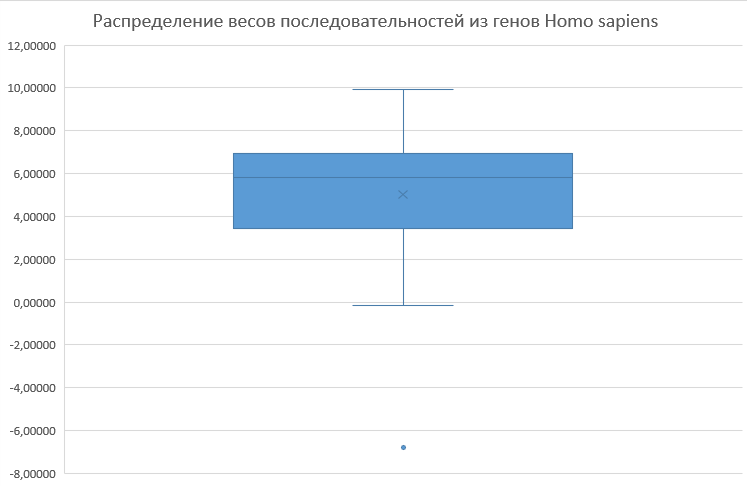
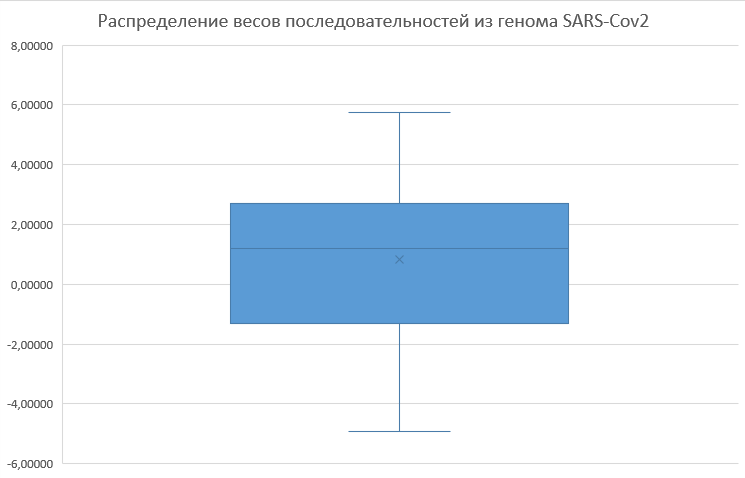
В качестве положительного контроля, как было упомянуто выше, использовались 60 генов из изначальной выборки, в качестве отрицательного - участки генома SARS-Cov2, содержащие ATG, но не ассоциированные с начальными участками генов. Для подготовки выборки последовательностей из генома вируса использовался следующий скрипт. На вход скрипт принимает полный геном вируса и файл со списком координат ATG. В результате имеем следующий список последовательностей. Далее веса последовательностей из двух выборок рассчитываются по приведенной выше матрице с помощью программы. Она принимает на вход матрицу PWM (без заголовков, только однобуквенные обозначения азотистых оснований и значения, соответствующие им в каждой позиции) и файл с рассматриваемыми последовательностями по одной в строке.
Результат видим на графиках ниже: медиана распределения весов последовательностей положительного контроля находится на уровне чуть ниже 6, для отрицательного контроля - около 1.5. На мой взгляд, разница достаточно значительна, чтобы утверждать, что последовательность, предшествующая ATG в геноме человека, является специфической.
Задание 3. LOGO.
Расчет значений матрицы информационного содержания осуществляется по формуле N(b,j)/Nw(b,j), где w(b,j)=ln(N(b,j)/p(b)) - значение PWM без учета псевдоотсчетов.
Матрица информационного содержания имеет вид:
| основание | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0,0613 | -0,0571 | -0,1052 | -0,0989 | -0,0371 | -0,0743 | -0,0884 | 1,2379 | 0 | 0 | -0,0989 | -0,0571 | -0,0879 |
| T | -0,0743 | -0,0884 | -0,1014 | -0,1014 | -0,0879 | -0,1052 | -0,1065 | 0 | 1,2379 | 0 | -0,0743 | -0,0571 | -0,0371 |
| G | 0,2996 | 0,0436 | 0,2174 | 0,2174 | 0,4338 | 0,0155 | -0,0648 | 0 | 0 | 1,5606 | 0,2174 | 0,0436 | 0,2996 |
| C | 0,1788 | 0,1788 | 0,2996 | 0,2577 | -0,0098 | 0,343 | 0,6299 | 0 | 0 | 0 | 0,0742 | 0,107 | 0,0742 |
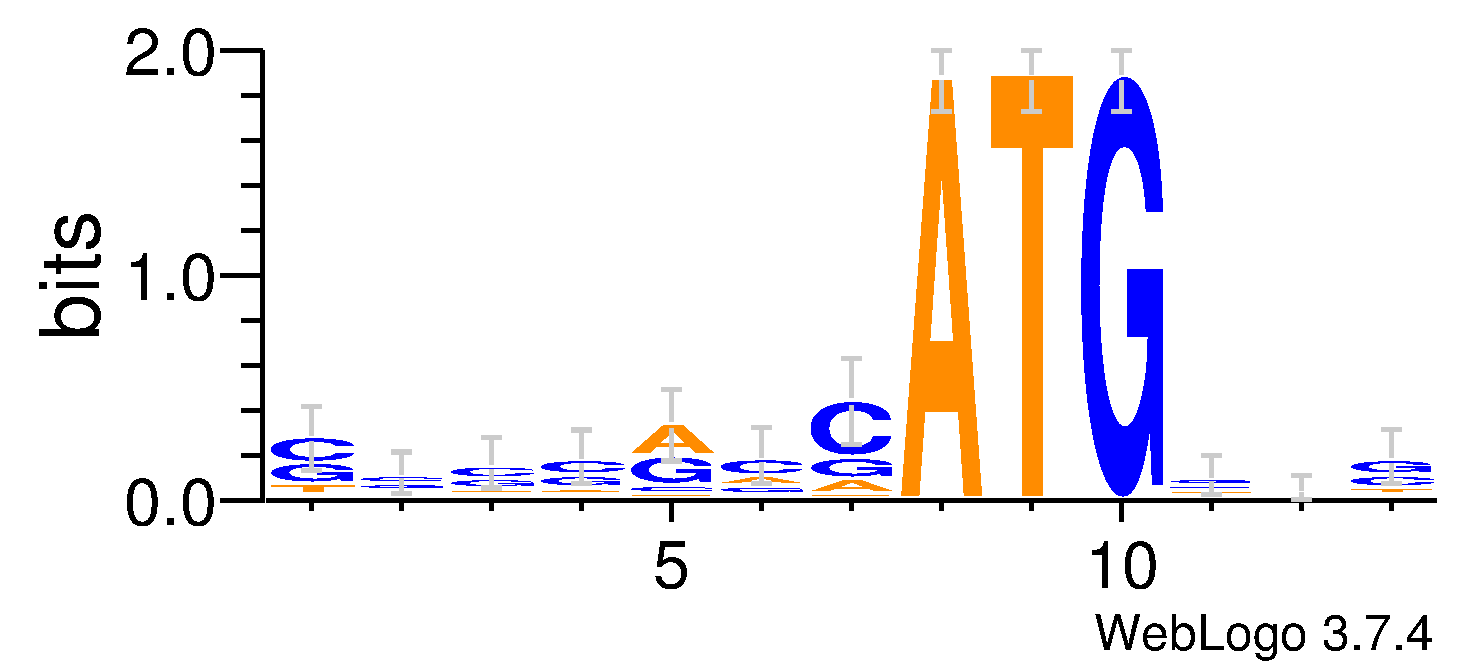
С помощью программы WebLOGO 3 была получена визуализация информационного содержания последовательности. Из схемы видно, что последовательность длиной 7 нуклеотидов, расположенная перед ATG в генах человека, имеет значимый информационный вес, иначе говоря, частоты встречаемости нуклеотидов в определенных позициях этой последовательности отличаются от таковых для всей последовательности, что может свидетельствовать о специфической функции этого участка.