Сигналы и мотивы - 3
Задание 1.
Механизм, позволяющий клетке уничтожать попавшую в неё чужерожную ДНК, не повреждая при этом соственную, носит название рестрикции-модификации. Он осуществляется одним либо несколькими ферментами, которые обладают нуклеазной и метилтрансферазной активностью. Функцей первых является разрезание чужеродной неметилированной ДНК в определеном участке - сайте рестрикции, - вторых - метилирование собственной ДНК.
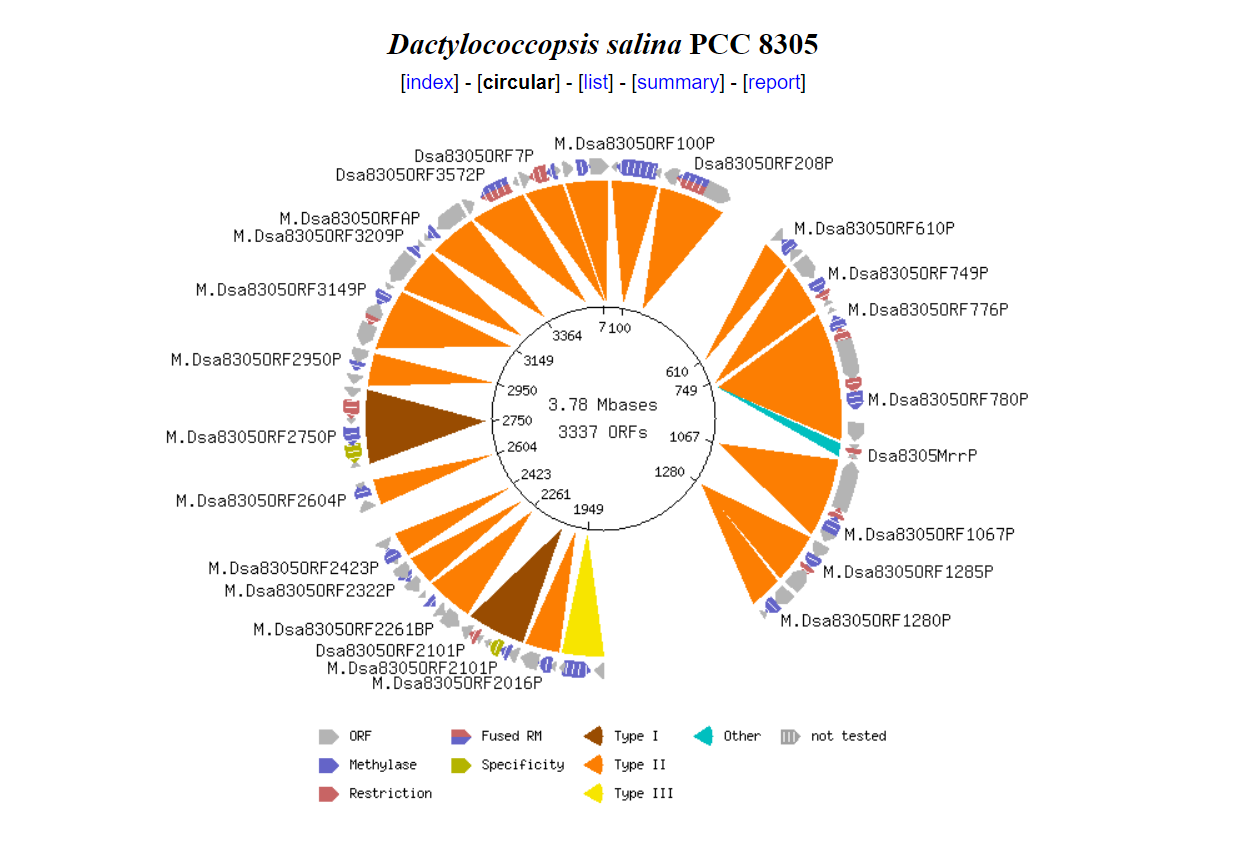
В качестве объекта для исследования в рамках данного задания был выбран штамм Dactylococcopsis salina PCC 8305, критерием выбора было достаточно большое количество генов систем рестрикции-модификации II типа (20). Эти системы характеризуются осуществлением метилтрансферазной и нуклеазной активности независимыми ферментами.Организм представляет собой мезофильную свободноживущую планктонную цианобактерию.
В разделе summary базы данных для выбранной бактерии представлены характеристики систем рестрикции-модификации, в том числе эндонуклеазы и их специфические сайты. Эта информация представлена в таблице ниже:
| сайт рестрикции | ферменты |
|---|---|
| GATC | M.Dsa8305ORF7P, Dsa8305ORF7P |
| CCWWGG | M.Dsa8305ORF12P |
| GCNGC | M.Dsa8305ORF610P |
| GGWCC | M.Dsa8305ORF749P, Dsa8305ORF749P |
| CCNGG | M.Dsa8305ORF776P, Dsa8305ORF776P |
| GTMKAC | M.Dsa8305ORF780P, Dsa8305ORF780P |
| CCRYGG | M.Dsa8305ORF1067P, Dsa8305ORF1067P |
| RGATCY | M.Dsa8305ORF1285P, Dsa8305ORF1285P |
| RGCGCY | M.Dsa8305ORF2016P |
| AATATT | M.Dsa8305ORF2261BP |
| CCWGG | M.Dsa8305ORF2322P |
| GGCC | M.Dsa8305ORF2423P |
| CCGG | M.Dsa8305ORF2604P |
| CGATCG | M.Dsa8305ORF3149P, Dsa8305ORF3149P |
| CTGCAG | M.Dsa8305ORFAP |
Для поиска недопредставленных сайтов рестрикции в геноме Dactylococcopsis salina с помощью скрипта был подготовлен список известных сайтов рестрикции. Скрипт принимает на вход предоставленный в задании файл и выбирает из 5-го столбца последовательности сайтов рестрикции, если они известны и имеют длину более 2 нуклеотидов. Полученный список принимает программа CBcalc. Я воспользовалась веб-версией (метод Карлина) и получила следующий результат. За недопредставленные я приняла сайты, для которых O/E ratio менее 0.8, и с помощью следующего скрипта я выбрала их последовательности (файл) и осуществила поиск эндонуклеаз, вносящих разрыв в эти сайты, по первоначальной таблице. В результате получился следующий список.
Задание 2.
В качестве объекта для поиска родственных последовательностей был выбран белок с идентификатором Q7VDL2 - probable septum site-determining protein MinC из Prochlorococcus marinus. Этот белок является ингибитором клеточного деления, так как нарушает формирование полярных Z-колец, которые в свою очередь служат каркасом для цитоскелетных белков. Механизм работы белка MinC заключается в том, что он дестабилизирует филаменты белка FtsZ, формирующего Z-кольца, и препятствует его полимеризации.
Для поиска гомологов данного белка был использован PSI-BLAST. Для стабилизации количества находок и установления значимой разности между худшей находкой выше порога на Е-value и лучшей находной ниже было выполнено 5 итераций, результат чего представлен в таблице ниже:
| номер итерации | число находок выше порога (0,005) | идентификатор худшей находки выше порога | E-value этой находки | идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 146 | Q9AG20.1 | 0.005 | A8GFG7.1 | 0.005 |
| 2 | 188 | B6JKX0.1 | 7e-08 | нет находок | - |
| 3 | 188 | Q9ZM51.1 | 2e-12 | A7H8E6.1 | 0.014 |
| 4 | 189 | A8MHK8.1 | 0.001 | A7H8E6.1 | 0.013 |
| 5 | 189 | A8MHK8.1 | 4e-10 | A7H8E6.1 | 0.009 |
На первой итерации запускается обычный BLAST по базе Swiss-Prot, поэтому вполне ожидаемо видеть, что Е-value худшей подходящей и лучшей не подходящей находок практически не отличаются (в данном случае, вероятно, за счет округления не отличается вообще). При втором запуске программы поиск новых белков осуществляется уже с помощью матрицы PSSM, построенной на основе полученного на первом шаге выравнивания, поэтому находки заметно лучше - самый высокий Е-value составляет 7е-08, а неудачные находки отсутствуют. На третьей итрации можно было бы остановиться, так как различие в E-value худшей подходящей и лучшей не подходящей находок значительно, а число найденных последовательностей не изменилось, но я решила проверить и запустила программу еще раз. На четвертой итерации добавляется еще одна находка и уменьшается разница Е-value граничных находок, но на 5-й итерации (и далее) наблюдаем стабилизацию.
Итого: порог на E-value менять не пришлось, стабилизировался результат по сути на третьей итерации, что говорит о том, что семейство характеризуется высокой степенью сходства и специфичности