База данных STRING
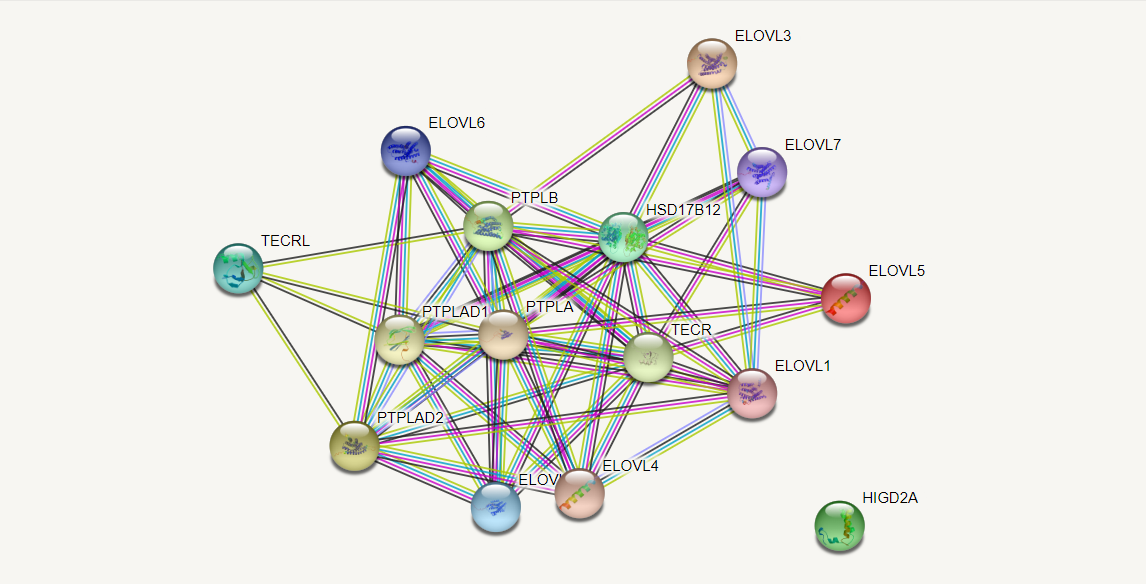
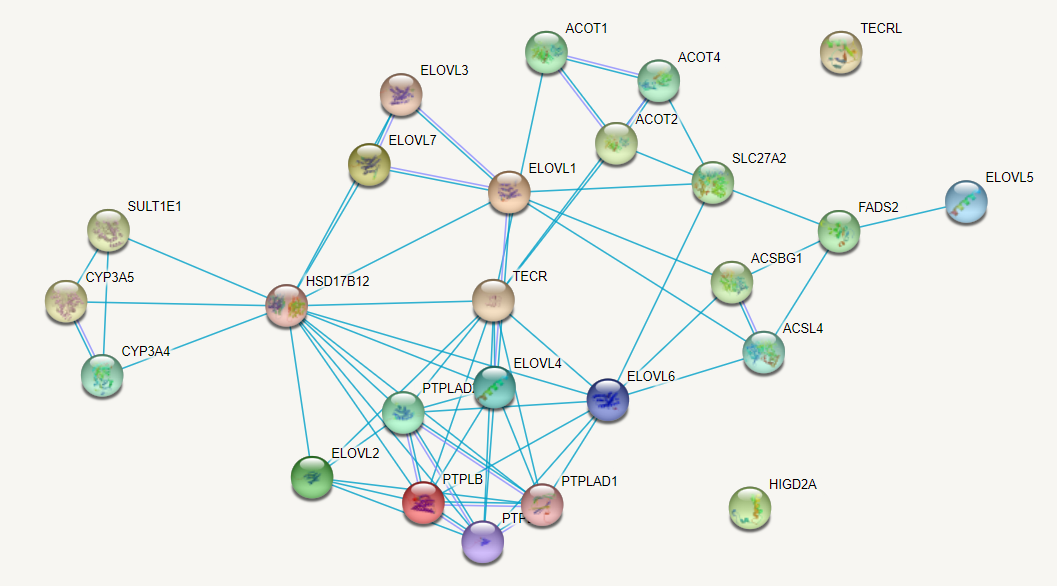
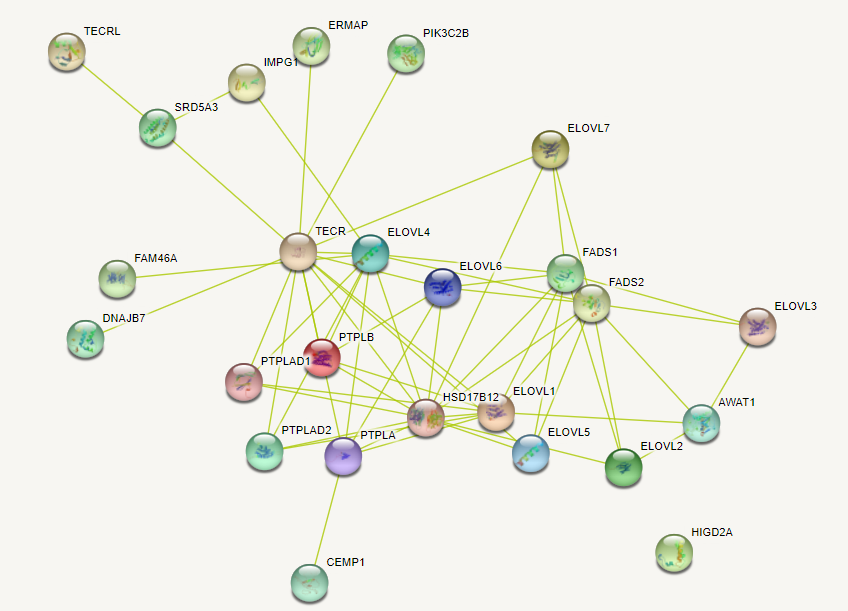
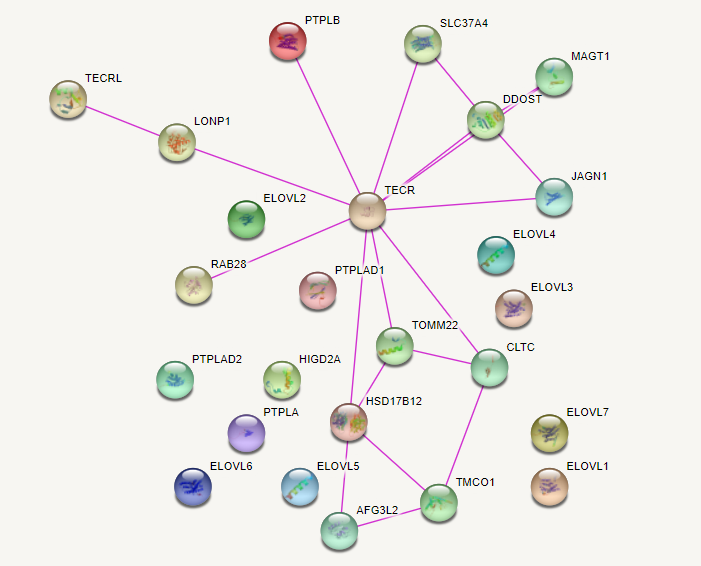
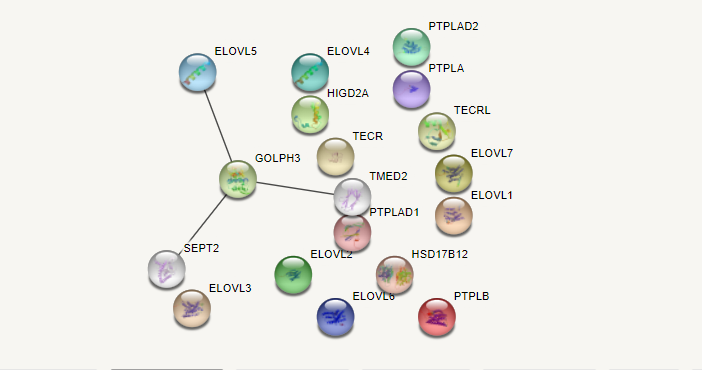
Для выполнения данного практикума мне был выдан следующий список генов. Для его исследования я воспользовалась базой данных STRING. Во вкладке Multiple proteins я ввела список генов, в качестве организма указала Homo sapiens. Сначала программой были найдены соответствующие генам белки Homo sapiens, затем проведен анализ. На схеме слева показаны все найденные виды взаимодействий между данными белками. Разными цветами показаны разные типы взаимодействий. Для всех 15 белков известны 3D-структуры, они также показаны на схеме. На картинках ниже показаны схемы по каждому виду взаимодействий отдельно. Видно, что наибольшее число связей наблюдается по упоминанию в статьях и в базах данных, при этом нет информации о соседнем расположении в геноме, совместном присутствии или слиянии генов.
О каждом типе взаимодействий можно найти подробную информацию. Например, о генах HSD17B12 (редуктаза длинноцепочечных 3-оксоацил-СоА) и ELOVL3 (участвует в элонгации длинноцепочечных жирных кислот) известно, что точных данных о их совместной экспрессии нет, но она наблюдается у их гомологов из других организмов, их биохимическое взаимодействие также не доказано, но есть данные о взаимодействии их гомологов, между ними есть ассоциации в курируемых базах данных, они упоминаются в одних и тех же абстрактах PubMed, при этом есть данные о их совместном присутствии у других организмов.
Во вкладке Experiments можно найти информацию о том, каким образом было показано биохимическое взаимодействие рассматриваемых белков. Например, для HSD17B12 и TECR было показано взаимодействие с высокой вероятностью методом биохимического анализа. Взаимодействие PTPLB и TECR показано двугибридным методом со средней степенью точности, и с аналогичносй точностью взаимодействие ELOVL1 и HSD17B12 было показано методом аффинной хроматографии. Для других белков имеются данные экспериментальной точности, полученные методами основанного на сближении биотинилирования и pull down.
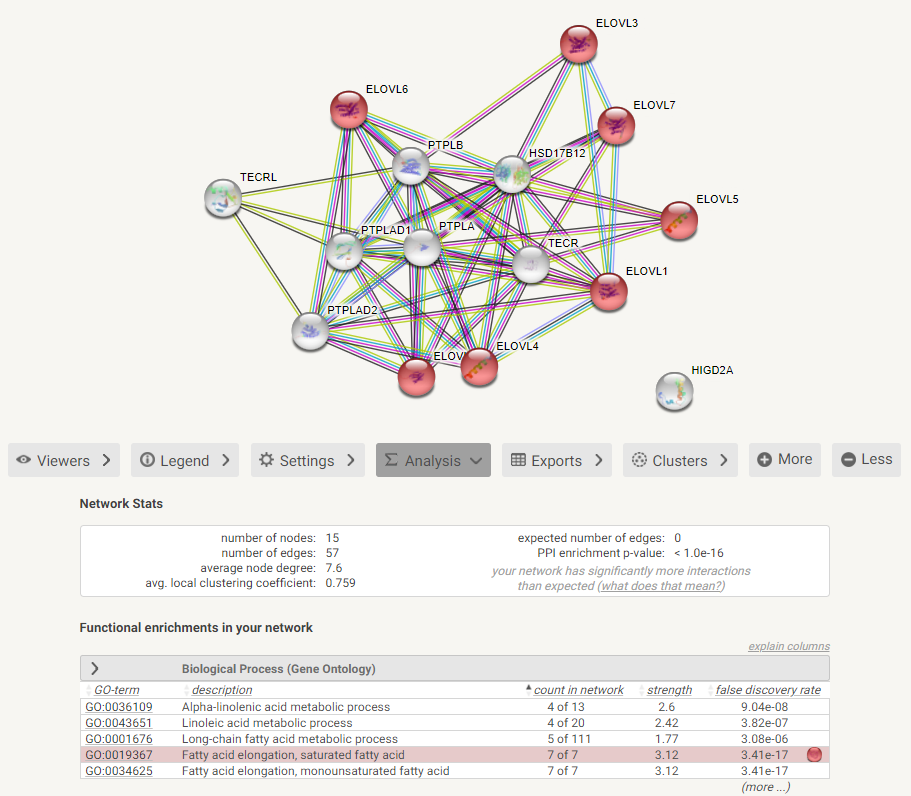
Анализ обогащения показал следующие результаты по разным разделам Gene Ontology:
- Биологические процессы: метаболизм альфа-линоленвой кислоты (FDR 9.04e-08), метаболизм линолевой кислоты (FDR 3.82e-07), метаболизм длинноцепочечных жирных кислот (FDR 3.08e-06), элонгация насыщенных и мононенасыщенных жирных кислот (FDR 3.41e-17)
- Молекулярные функции: дегидратация 3-гидрокисацил-СоА (FDR 7.20e-09)
- Местоположение в клетке: мембрана эндоплазматического ретикулума (FDR 8.69e-20)
Можно отдельно посмотреть на схеме, какие именно белки принадлежат какой-либо из функциональных групп. Например, как видно из рисунка, в элонгации насыщенных жирных кислот участвуют белки группы ELOVL. Также имеются ссылки на другие базы данных, в частности KEGG, что позволяет получить информацию о метаболических путях, в которые вовлечены указанные белки.
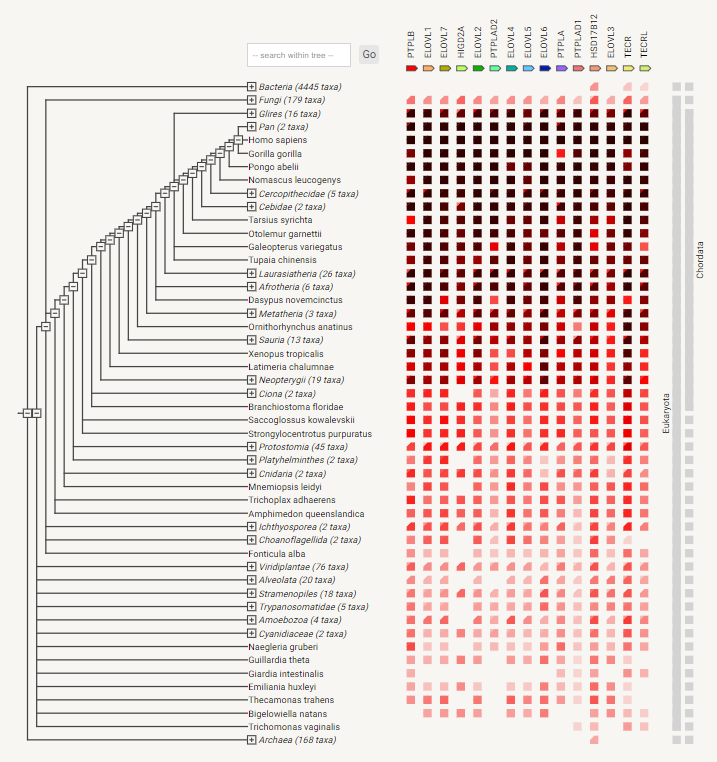
Анализ совместного присутствия генов у разных организмов показывает, что больше всего они встречаются у млекопитающих, при этом их встречаемость примерно одинакова. Среди необычных явлений можно отметить низкую представленность гена PTPLA у Gorilla gorilla, PTPLB у Tarsius syrichta, PTPLAD2, HSD17B12 и TECRL у Galeopterus variegatus. Можно отметить, что гены имеются в полном наборе у всех Хордовых, за исключением Оболочников (отсутствует ген HIGD2A). У остальных представителей Opisthokonta гены еще встречаются, хоть и реже, за пределами клады - редко и часто не в полном наборе. Но при этом ген HSD17B12 имеется даже у архей, а у бактерий, помимо него, имеются также TECR и TECRL. Что примечательно, ген HSD17B12 присутствует во всех таксонах, что позволяет предположить, что из данной группы он был самым древним и появился у общего предка клеточных организмов. Наиболее специфичным для позвоночных животных можно считать ген HIG2A, который чаще всего исчезает в более древних таксонах.
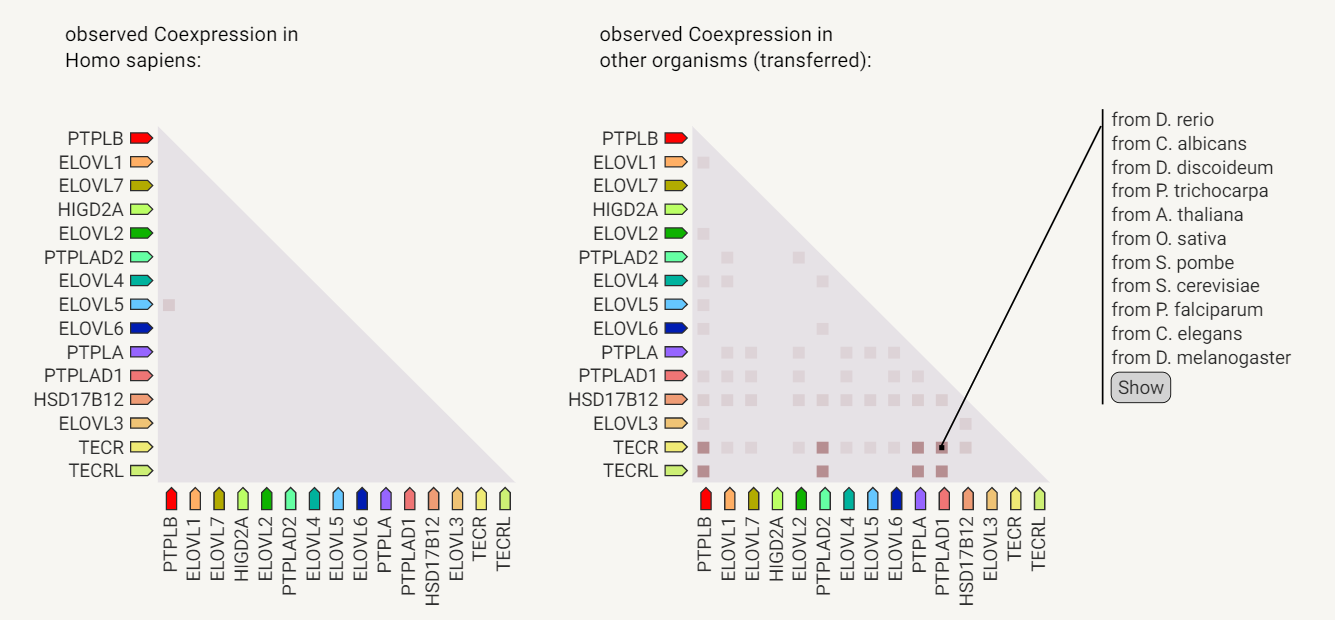
Анализ совместной экспрессии показывает, что у человека ассоциация всего одна - между генами ELOVL5 и PTPLB, при этом у других организмов наблюдаются довольно значительные корреляции, в частности генов PTPLAD2, PTPLA и PTPLAD1 с TECR и TECRL. На сноске можно видеть, у каких именно организмов наблюдается совместная экспрессия.