Трансмембранные белки
Задание 1. Знакомство с базой данных OPM
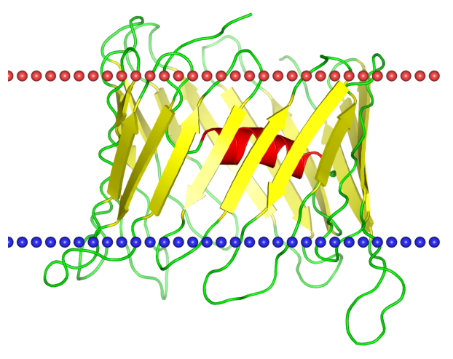
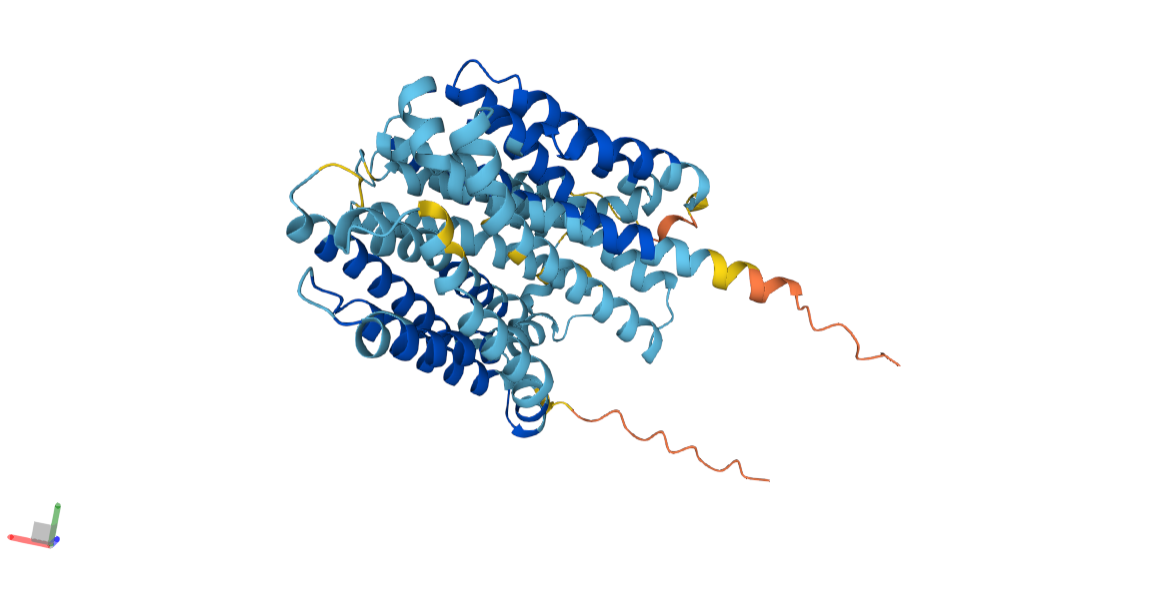
Для выполнения первого задания я выбрала в базе данных OPM трансмембранный белок, у которого погруженная в мембрану часть структуры представляет собой бета-бочонок (OPM -> Classes -> Beta-barrel transmembrane). Методом интуиции я выбрала канальный белок VDAC-1, который представляет собой потенциалзависимый канал, обеспечивающий транспорт анионов. Данный белок локализован во внешней мембране митохондрий человека. Справа представлено изображение вторичной структуры белка и его расположения в мембране, представленное на странице в базе данных.
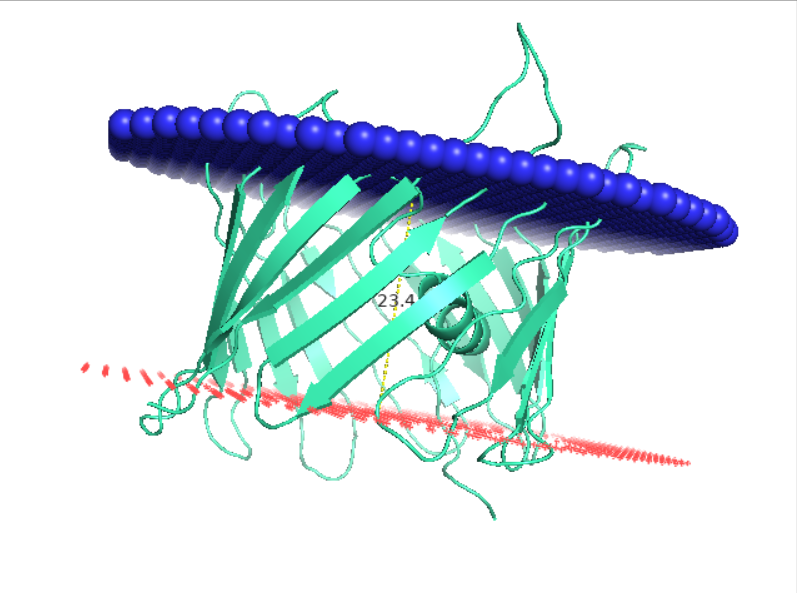
Толщина гидрофобной части белка составляет 23.4 ангстрем, согласно информации из базы данных. Аналогичный результат можно получить, измерив с помощью команды distance в Pymol расстояние между двумя слоями липидов мембраны.
Согласно базе данных, белок имеет следующие гидрофобные участки: 1(29-36), 2(42-51), 3(58-65), 4(72-79), 5(84-90), 6(98-105), 7(115-122), 8(126-134), 9(141-148), 10(152-160), 11(170-176), 12(182-187), 13(193-199), 14(206-213), 15(222-228), 16(235-241), 17(246-252), 18(258-266), 19(277-285). Всего 19 бета-листов, пронизывающих мембрану, средняя длина которых составляет 6-7 остатков.
| Характеристика | Данные |
|---|---|
| Тип белка | Трансмембранный белок, бета-бочонок |
| Суперсемейство | Митохондриальные и пластидные порины |
| Семейсво | Потенциалзависимый анионный канал |
| Организм | Homo sapiens |
| Локализация | Внешняя мембрана митохондрий |
| Uniprot ID | VDAC1_HUMAN |
| PDB | 2JK4 |
| Толщина гидрофобного слоя | 23.4 |
| Число трансмембранных структур | 19 |
| Средняя длина трансмембранных структур | 6-7 |
Задание 2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
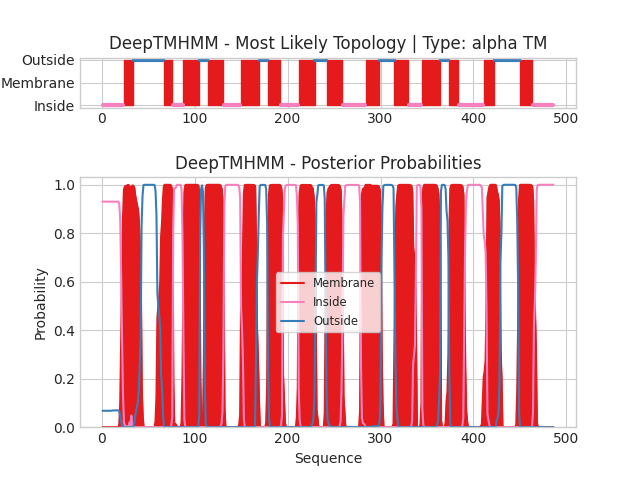
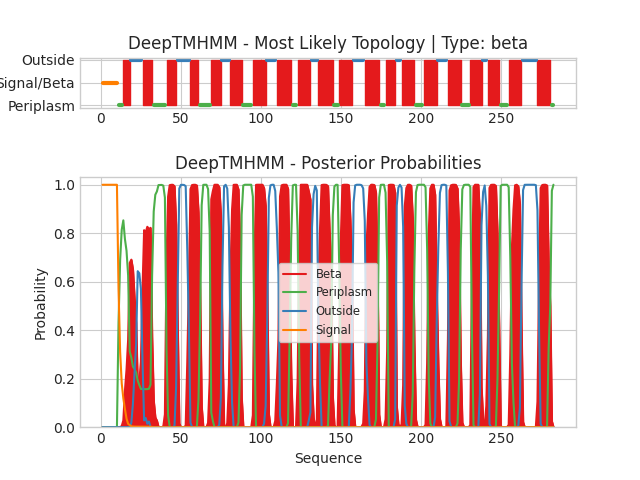
В качестве альфа-спирального белка был взят транспортер цефамицина CMCT_AMYLA, бета-листового - потенциалзависимый анион-селективный канал VDAC1_HUMAN. В качестве входных данных использовались fasta-файлы: CMCT_AMYLA, VDAC1_HUMAN. Полученные предсказания трансмембранных участков представлены на рисунках слева. Текстовую выдачу программы можно посмотреть по ссылкам: CMCT_AMYLA, VDAC1_HUMAN. На схеме для CMCT_AMYLA выделены участки структуры, расположенные со стороны цитоплазмы (inside), с внешней стороны клетки (outside) и внутри мембраны (membrane). В нижней части схемы для каждого аминокислотного остатка показана вероятность принадлежания его определенной части структуры. Можно отметить, что N- и C-концы погружены в цитоплазму. На схеме для VDAC1_HUMAN отмечены участки, расположенные с внешней стороны мембраны митохондрии (обращенные в цитоплазму), обращенные в межмембранное пространство (periplasm) и пронизывающие мембрану. Также имеется сигнальная структура в виде спирали. N- и С-концы обращены в межмембранное пространство.
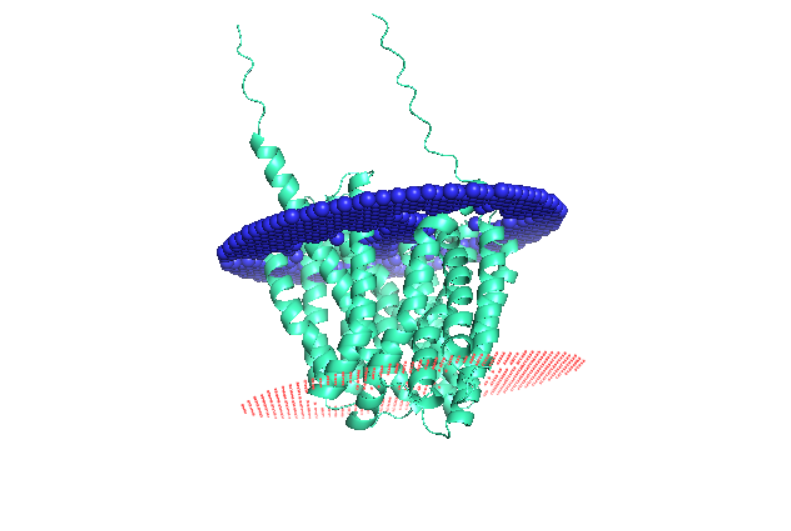
Задание 3. PPM: Предсказание положения выданного белка в мембране
Объектом исследования в следующем задании был выданный мне белок - экспортер цефамицина CMCT_AMYLA. Его структура достоверно неизвестна, но существует её модель, предсказанная с помощью алгоритма AlphaFold. Эта модель изображена на рисунке слева, а также её можно просмотреть, открыв следующий PDB-файл. Касательно самой модели можно отметить, что качество предсказания достаточно высокое, практически вся структура, помимо лишенных вторичной структуры концевых участков, имеет pLDDT > 70 (качество предсказания здесь убывает от темно-синего к красному). От качества модели должно напрямую зависеть качество предсказания топологии, ведь протяженность находящихся внутри мемраны участков белка зависит от длины альфа-спиралей - количества включенных в них аминокислот, в то время как число аминокислот, вовлеченных в формирование вторичной структуры, определяется в процессе предсказания структуры. Для предсказания его расположения в мембране была использована программа PPM, которая принимает на вход файл с координатами (представлен выше) и набор параметров. В данном случае я указала, что белок находится в мембране грам-положительной бактерии (информацию о таксономической принадлежности организма можно найти в Uniprot), N-конец обращен внутрь от мембраны (можно было увидеть на схеме, полученной в предыдущем задании), белок пересекает одну мембрану. Отдельно исключать небелковые молекулы в данном случае не требуется, так как на вход подается не структура, полученная рентгеноструктурным анализом кристалла, а только предсказание, и файл не содержит ничего, кроме белка. Полученная информация о расположении его в мембране представлена в следующей таблице:
| Характеристика | Данные |
|---|---|
| Тип белка | Трансмембранный альфа-спиральный |
| Название | Белок экспорта цефамицина |
| Организм | Amycolatopsis lactamdurans |
| Локализация | Мембрана бактериальной клетки |
| Uniprot ID | CMCT_AMYLA |
| PDB | модель |
| Толщина гидрофобного слоя | 30.9 ± 0.6 |
| Число трансмембранных структур | 14 |
| Средняя длина трансмембранных структур | 20-21 |
С помощью данной программы были предсказаны следующие участки, расположенные внутри мембраны: 1( 24- 44), 2( 57- 77), 3( 88- 105), 4( 111- 131), 5( 149- 170), 6( 175- 195), 7( 212- 232), 8( 242- 261), 9( 278- 305),10( 312- 337),11( 343- 364),12( 369- 393),13( 409- 431),14( 446- 466). Нижний рисунок справа отражает их положение относительно мембраны.
Задание 4. Сравнение алгоритмов предсказания трансмембранных спиралей
В рамках практической работы было получено несколько пресказаний внутримембранных участков. Для начала рассмотрим предсказания для альфа-спирального белка CMCT_AMYLA: имеем предсказание сервера PPM и DeepTMHMM. Приведем ниже списки найденных разными программами - PPM и DeepTMHMM соответственно - участков еще раз:
1( 24- 44), 2( 57- 77), 3( 88- 105), 4( 111- 131), 5( 149- 170), 6( 175- 195), 7( 212- 232), 8( 242- 261), 9( 278- 305),10( 312- 337),11( 343- 364),12( 369- 393),13( 409- 431),14( 446- 466)
1(22-42), 2(60-76), 3(88-105), 4(111-131), 5(150-166), 6(179-194), 7(212-230), 8(241-259), 9(279-300), 10(316-338), 11(345-364), 12(374-389), 13(415-428), 14(449-464)
Из сопоставления видно, что для некоторых участков предсказания совпали полностью, в основном есть расхождения на 2-5 остатков, но тем не менее все спирали найдены примерно в одних и тех же частях последовательности. Аналогично посмотрим VDAC1_HUMAN: база данных ОРМ и предсказание DeepTMHMM:
1(29-36),2(42-51),3(58-65),4(72-79),5(84-90),6(98-105),7(115-122),8(126-134),9(141-148),10(152-160),11(170-176),12(182-187),13(193-199),14(206-213),15(222-228),16(235-241),17(246-252),18(258-266),19(277-285)
1(41-47), 2(56-62), 3(69-75), 4(81-87), 5(96-104), 6(112-118), 7(124-131), 8(136-144), 9(150-157), 10(166-173), 11(180-184), 12(190-195), 13(205-209), 14(218-224), 15(232-237), 16(243-249), 17(257-262), 18(247-281)
Нетрудно заметить, что в этом случае DeepTMHMM справился хуже и предсказал на один трансмембранный элемент меньше. Согласно данным ОРМ, участок 29-36 также является трансмембранным, но в выдаче DeepTMHMM он входит в состав периплазматического.
Задание 5. База данных TCDB
Последнее задание заключалось в том, чтобы найти информацию о белках в базе данных TCDB. Поиск осуществлялся по идентификаторам Uniprot, при этом белок CMCT_AMYLA(Q04733) в базе данных найден, а VDAC1_HUMAN(P21796) - нет. Для CMCT_AMYLA в базе данных существует страница, где можно найти информацию о длине последовательности, молярной массе, принадлежности организму, топологии белка, субстрате и условиях его взаимодействия с субстратом, количестве трансмембранных элементов. По команде Predict TMSs можно получить список координат трансмембранных участков и визуализацию их в последовательности. По сравнению с предсказанием PPM, есть некоторые различия в количестве включенных в каждый элемент остатков, но их количество и расположение совпадает. Данный белок имеет ТС-код 2.А.1.3.8, что отражает его включение в определенные категории трансмембранных белков: 2 - электрохимический потенциалзависимый транспортер; А - транспортный белок, формирующий пору; 3 - семейство транспортеров аминокислотных органокатионов (АРС); 8 - семейство животных белков, которые для функционирования нуждаются в связывании с трансмембранным гликопротеидом I типа (LAT).
Для VDAC1_HUMAN по запросу не нашлось отдельной страницы, однако он упомянут на страницe семейства. Проходя по дереву белков в разделе ТС-system по цепочке Channels/Pores -> β-Barrel Porins -> The Mitochondrial and Plastid Porin (MPP) Family попадаем на нужную страницу и ищем упоминание VDAC1. Его ТС-код 1.В.8.-, о нем известно, что его оверэкспрессия у человека может приводить к развитию раковых опухолей, он вовлечен в процессы клеточного метаболизма и апоптоза.