Знакомство с базой данных OPM
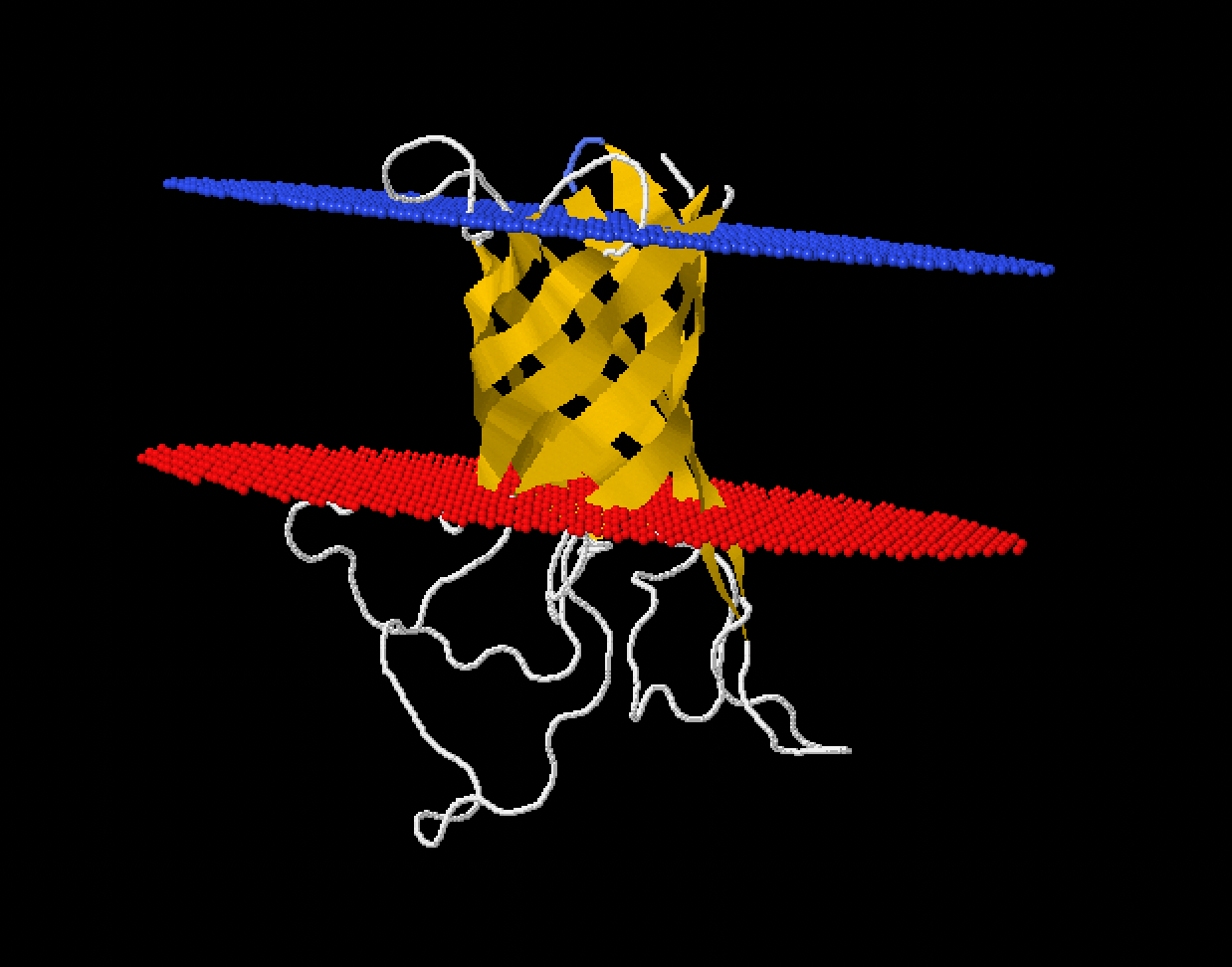
Для выполнения данного задания в базе данных OMP был выбран 2lhf - Белок внешней мембраны H
AC в UniProt: Q51486 ; идентификатор PDB 2lhf.
| Характеристика | Данные |
| Тип белка | Трансмембранный белок, бета-бочонок |
| Суперсемейство | Opacity porins |
| Семейсво | Opacity porins |
| Организм | Pseudomonas aeruginosa (Синегнойная палочка) |
| Локализация | Внешняя мембрана Грам-отрицательной бактерии |
| Uniprot ID | Q51486_PSEAI |
| Толщина гидрофобного слоя | 20.6 Å |
| Число трансмембранных структур | 8 |
| Средняя длина трансмембранных структур | 9 |
2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
Мне был выделен белок с AC в UniProt Q9ZDS0, выбранный мной для первого задания белок с AC в UniProt Q51486 при помощи сервиса DeepTMHMM был проведен анализ каждого из этих белков.
Этот сервис позволяет предсказывать трансмемранные участки белка по первичной структуре.
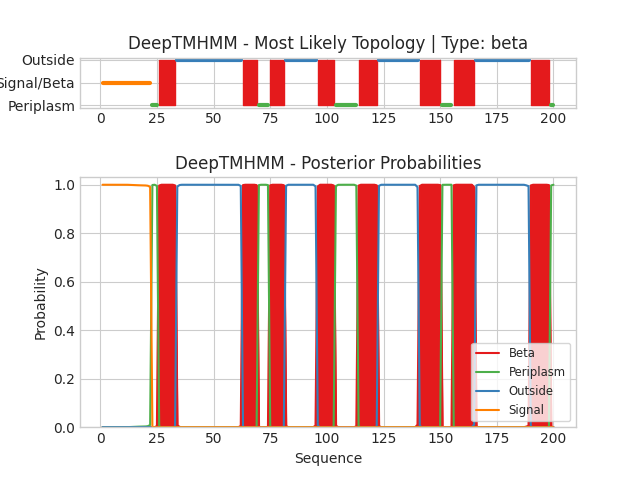
На схеме цвета обозначают где находится участок: Beta - бета-слой, рeriplasm - периплазматический слой, Outside - внеклеточный, Signal - сигнальный пептид. По горизонтали отмечены координаты остатков белка, по вертикали на верхней схеме рисунка - предсказание положения участков белка, а на нижней схеме - вероятность с которой остаток принадлежит той или иной топологии. Для этого белка предсказано 8 трансмембранных участков. N-конец оказался сигнальным, после него следует небольшой периплазматический участок. С-конец находится в периплазматическом слое.
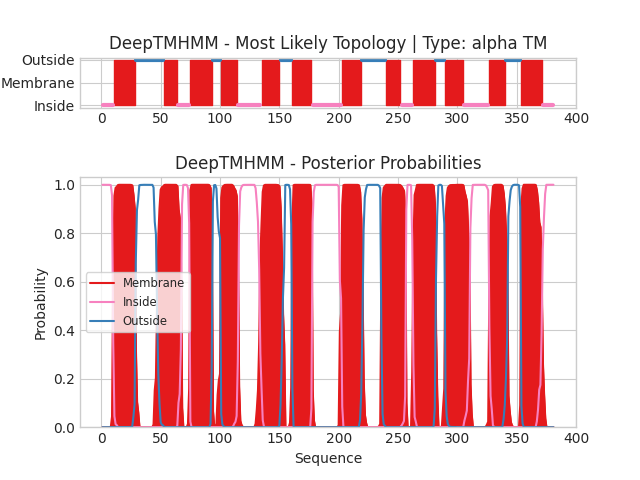
На схемах показано какие участки белка находятся внутри мембраны (membrae), с внутренней стороны мембраны (inside) и с внешней стороны мембраны (outside). По горизонтали отмечены координаты остатков белка, по вертикали на верхней схеме рисунка - предсказание положения участков белка, а на нижней схеме - вероятность с которой остаток принадлежит той или иной топологии. Программа предсказала 12 трансмембранных участков. N и C концы находятся с внутренней стороны мембраны.
3. PPM: Предсказание положения выданного белка в мембране
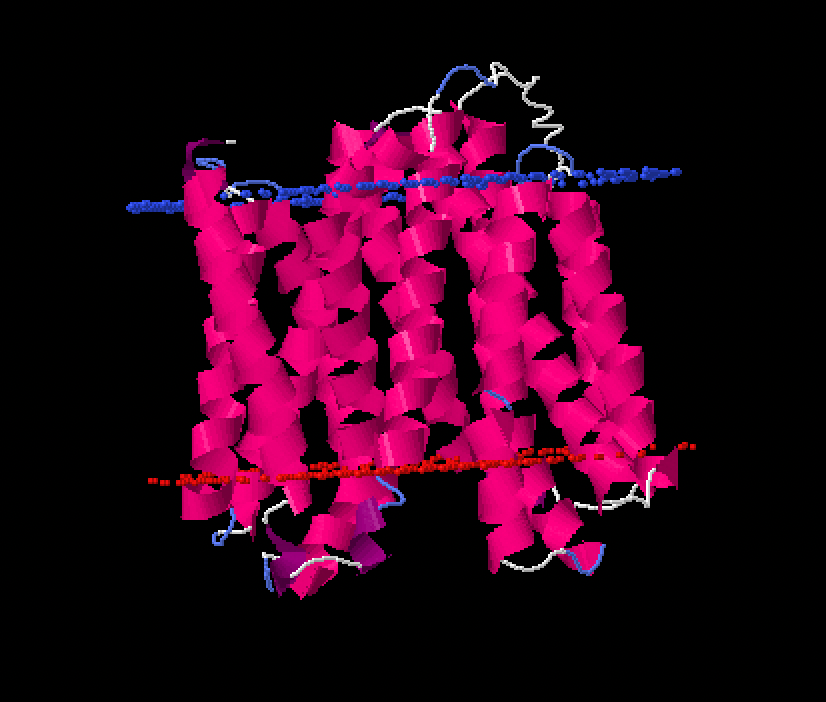
Для данного задания я использовал выданный мне белок с AC в UniProt Q52813. В соответствии с данными, полученными в предыдущем задании параметр Topology (N-ter) был установлен in.
| Характеристика | Данные |
| Толщина гидрофобного слоя | 27.8 ± 2.6 Å |
| ΔGtransfer | -74.5 kcal/mol |
| Угол изгиба структур | 4 ± 2° |
| Число трансмембранных структур | 12 |
| Трансмембранные сегменты | 1( 8- 28), 2( 48- 67), 3( 72- 90), 4( 98- 122), 5( 129- 147), 6( 161- 177), 7( 199- 218), 8( 237- 256), 9( 261- 279),10( 290- 311),11( 322- 342),12( 354- 374) |
| Средняя длина трансмембранного сегмента | 19 |
4. Сравнение алгоритмов предсказания трансмембранных спиралей
Оба предсказания для Y255_RICPR в целом похожи.Оба алгоритма одинаково оценили количество трансмембранных участков. Отличия в пределах 1-5 остатков, при этом спирали находятся в +- одних и тех же местах (DeepTMHMM и PPM соответственно):
1(10-28), 2(47-67), 3(75-93), 4(101-115), 5(134-154), 6(161-177), 7(203-220), 8(236-256), 9(263-281), 10(290-310), 11(327-342), 12(354-371)
1( 8- 28), 2( 48- 67), 3( 72- 90), 4( 98- 122), 5( 129- 147), 6( 161- 177), 7( 199- 218), 8( 237- 256), 9( 261- 279),10( 290- 311),11( 322- 342),12( 354- 374)
Судя по AlphaFold структура белка предсказана с высокой точностью,за исключением его концов, что не должно сильно влиять на результат работы РРМ
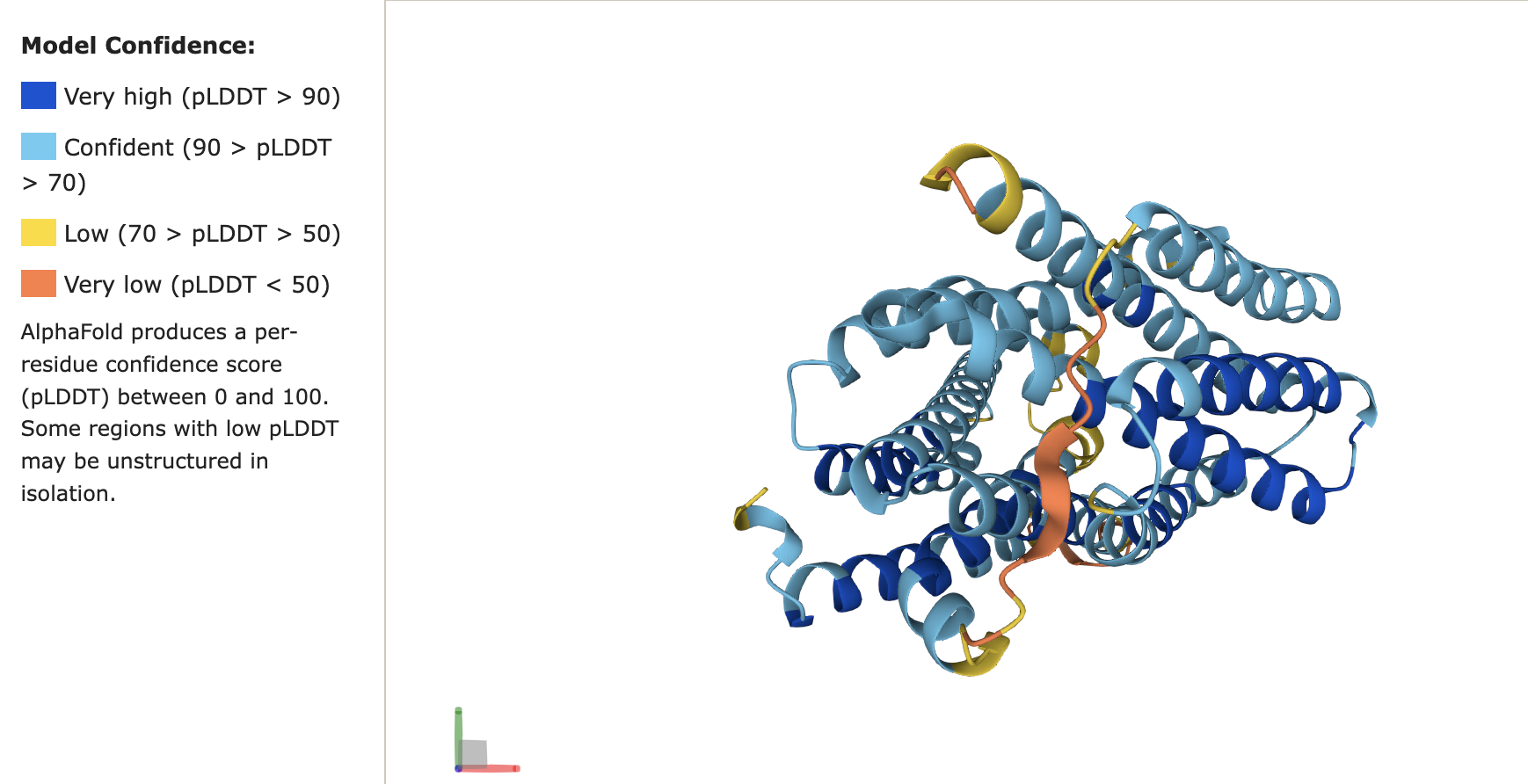
Для Q51486 все оказалось не так гладко. Ни один участок не был предсказан верно, возможно алгориитм плохо предсказывает положение B-листов в мембранах. (DeepTMHMM и PPM соответственно):
1(26 - 33), 2(63-69), 3(75-81) , 4(96-103), 5(114-122), 6(141-150), 7(156-165), 8(190-198)
1( 5- 11), 2( 41- 48), 3( 55- 60), 4( 74- 84), 5( 91- 101), 6( 119- 130), 7( 137- 144), 8( 170- 177)
База данных TCDB
В базе данных TCDB выбранного мной белка АС: Q51486 не оказалось.
Найденный Q9ZDS0 . Код: 2.A.1.64.2
2 - электрохимический потенциалзависимый транспортер
А - транспортный белок
1 - Фасилитаторы
64.2 - код белка