SECIS
Селеноцистеин распологается в белках с антиоксидантной активностью. В начале к специальной селенцистеиновой тРНК присоединяется цистеин. затем цистеин меняется на селеноцистеин с помощью селенцистеинсинтазы. Sec-тРНК не распознается фактором элонгации EF-Tu (eEF1). Ее кодон - это стоп кодон, но если в мРНК есть специальная шпилька SECIS-элемент, то этот кодон можно перекодировать а селенцистеин. В бактериях Sec-тРНК связывается с UGA кодоном и селенцистеин встраивается в пептидную цепь. У эукариот белок называетсяя eEFsec, a SECIS-элемент распологается в 3' нетранслируемой области, что позволяет вводить несколько селенцистеинов в белок, синтезируемый одной мРНК.
PWM последовательности Козак человека.
Для выполнения данного задания использовался скрипт Максима Смирнова, за что ему огромное Спасибо. Данный скрипт выбирает 100 случайных геннов человека и вырезает 7 нуклеотидов до старта трансляции ATG + ATG + 3 нук. после ATG. Результат: обучение (40), тест (60)
| N | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
| A | -2,29 | -0,37 | -0,65 | 0,11 | -0,82 | -0,50 | -0,82 | 1,22 | -4,08 | -4,08 | -0,37 | -0,50 | -1,31 |
| T | 0,37 | -0,50 | -1,31 | -0,37 | -1,31 | -0,82 | -1,68 | -4,08 | 1,22 | -4,08 | -1,04 | -1,04 | -0,82 |
| G | 0,31 | 0,31 | 0,74 | 0,11 | 0,85 | 0,54 | 0,11 | -3,72 | -3,72 | 1,58 | 0,99 | 0,39 | 0,94 |
| C | 0,31 | 0,54 | 0,54 | 0,21 | 0,47 | 0,54 | 1,08 | -3,72 | -3,72 | -3,72 | -0,28 | 0,74 | 0,31 |
Последовательности из тестового файла использовались для положительного тык контроля, а для отрицательного содержащие ATG. Также с помощью скрипта были получены positive control mean score: 5.242721611434907, negative control mean score: 1.8018855083930998, p-value: 6.763492270192289e-10 и ic:9.784051189548972. Их з этих данных можно сделать вывод о специфичности стартового кодона Козак, т.к. positive control больше чем negative control.
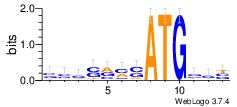
Logo
Для вычисления информационного содержания последовательности Козак была построена матрица информационного содержания.
| N | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
| A | -0.10 | -0.11 | -0.14 | 0.05 | -0.15 | -0.13 | -0.15 | 1.78 | 0 | 0 | -0.11 | -0.13 | -0.15 |
| T | 0.23 | -0.13 | -0.15 | -0.11 | -0.15 | -0.15 | -0.13 | 0 | 1.78 | 0 | -0.16 | -0.16 | -0.15 |
| G | 0.13 | 0.13 | 0.46 | 0.04 | 0.59 | 0.28 | 0.04 | 0 | 0 | 2.30 | 0.80 | 0.17 | 0.73 |
| C | 0.13 | 0.28 | 0.28 | 0.08 | 0.23 | 0.28 | 0.94 | 0 | 0 | 0 | -0.06 | 0.46 | 0.13 |
После чего ИС было визуализированно с помощью программы WebLOGO 3