Чтение последовательностей по Сэнгеру
Задание 1
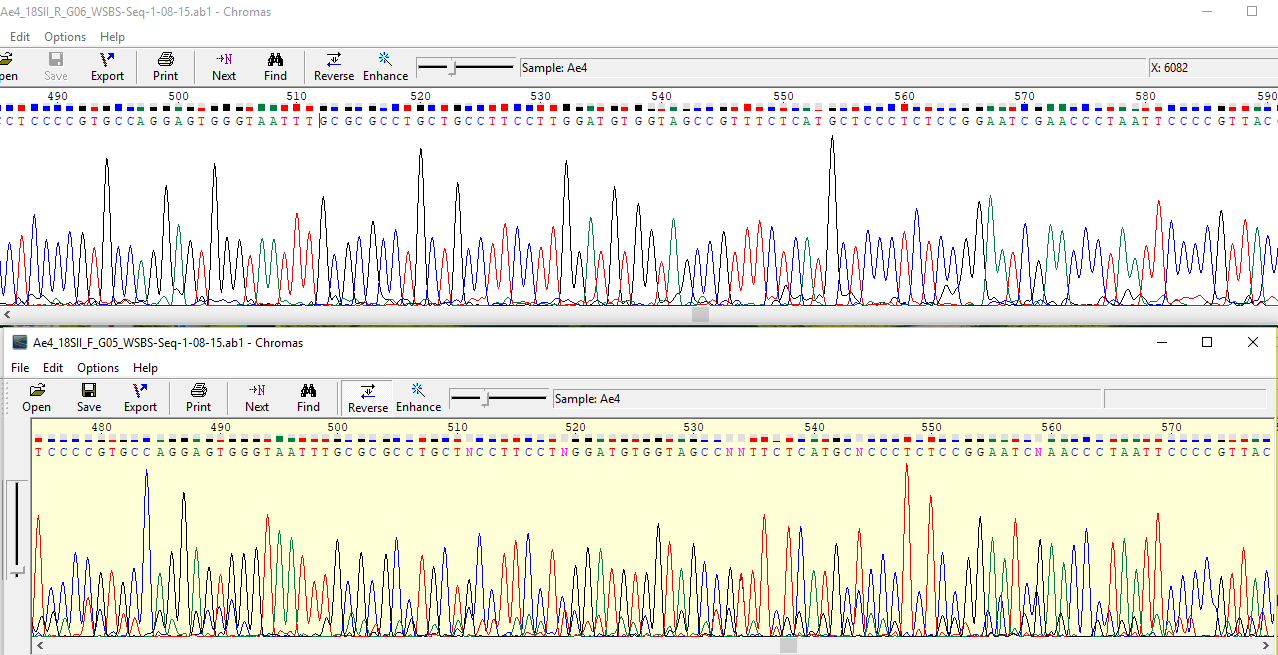
Ниже представлены два случайных участка двух последовательностей:
1. Ae6_18SII_R_H06_WSBS-Seq-1-08-15.ab1 ( ссылка)
2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 ( ссылка)
Для последней была применена комаенда "reverse complement", соответсвенно на картинке показана комплементарная цепочка.
Обе хроматограммы можно назвать хоришими, потому что по подавляющемубольшенству пиков однозначно определяется нуклеотид
в последовательности. Однако стоит отметить, что уровень шума для первой последовательности существенно ниже,
чем для второй. Если для первой отношенение сигнала к шуму отличается в среднем примерно в 20 раз, то для второй в 5.
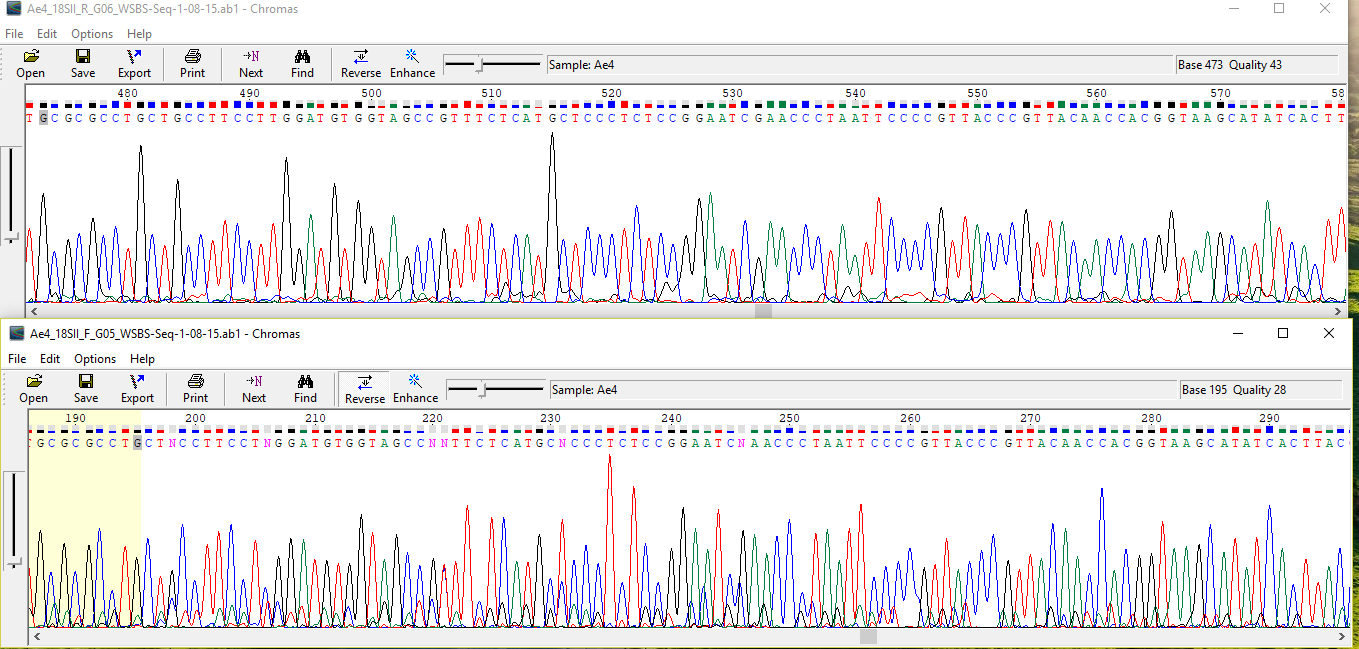
Границы нечитаемых 5'- и 3'-участков, которые были удалены:
1. Ae6_18SII_R_H06_WSBS-Seq-1-08-15.ab1:1-39 и 750-920
2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1:1-39 и 620-920
Проблемные участки:
Представлены диграммы после удаления нечитаемых участков.
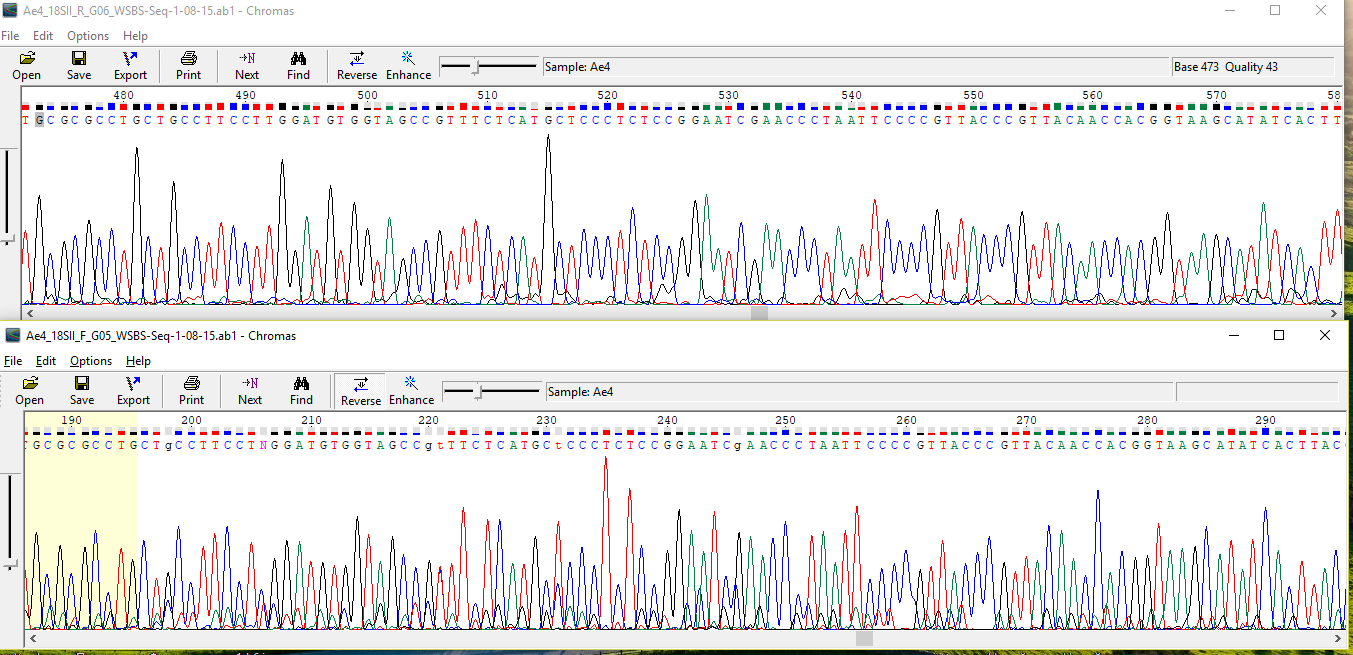
 На хроматограмме для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 видно 5 букв "N". Если сравнить нуклеотиды 2 последовательностье,
то видно, что все буквы, кроме "N" совпадают. Однако для данного участка можно однозначно определить каждую букву "N",
потому что для 1. Ae6_18SII_R_H06_WSBS-Seq-1-08-15.ab1 буквы в соответствующих местах однозначно определены и цвет максимальных
пиков для двух последовательностей совпадает, но для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 слишком большой шум.
Ниже приведена картинка с исправлеными нуклеотидами(написаны прописными буквами).
На хроматограмме для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 видно 5 букв "N". Если сравнить нуклеотиды 2 последовательностье,
то видно, что все буквы, кроме "N" совпадают. Однако для данного участка можно однозначно определить каждую букву "N",
потому что для 1. Ae6_18SII_R_H06_WSBS-Seq-1-08-15.ab1 буквы в соответствующих местах однозначно определены и цвет максимальных
пиков для двух последовательностей совпадает, но для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 слишком большой шум.
Ниже приведена картинка с исправлеными нуклеотидами(написаны прописными буквами).
 последовательности в Fasta-формате:
1. Ae4_18SII_R_G06_WSBS-Seq-1-08-15
2. Ae4_18SII_F_G05_WSBS-Seq-1-08-15
проект выравнивания:
ссылка
последовательности в Fasta-формате:
1. Ae4_18SII_R_G06_WSBS-Seq-1-08-15
2. Ae4_18SII_F_G05_WSBS-Seq-1-08-15
проект выравнивания:
ссылка
Задание 2
На картинке представлен нечитаемый учаток хроматограммы.
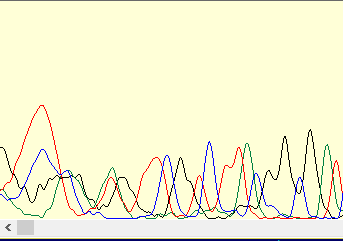 Хроматограмма является нечитаемой, так как пики размытые и непериодичные, сигнал шума на слишком высоком уровне, поэтому
невозможно установить нуклеотидную последовательность.
Хроматограмма является нечитаемой, так как пики размытые и непериодичные, сигнал шума на слишком высоком уровне, поэтому
невозможно установить нуклеотидную последовательность.
 На хроматограмме для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 видно 5 букв "N". Если сравнить нуклеотиды 2 последовательностье,
то видно, что все буквы, кроме "N" совпадают. Однако для данного участка можно однозначно определить каждую букву "N",
потому что для 1. Ae6_18SII_R_H06_WSBS-Seq-1-08-15.ab1 буквы в соответствующих местах однозначно определены и цвет максимальных
пиков для двух последовательностей совпадает, но для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 слишком большой шум.
Ниже приведена картинка с исправлеными нуклеотидами(написаны прописными буквами).
На хроматограмме для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 видно 5 букв "N". Если сравнить нуклеотиды 2 последовательностье,
то видно, что все буквы, кроме "N" совпадают. Однако для данного участка можно однозначно определить каждую букву "N",
потому что для 1. Ae6_18SII_R_H06_WSBS-Seq-1-08-15.ab1 буквы в соответствующих местах однозначно определены и цвет максимальных
пиков для двух последовательностей совпадает, но для 2. Ae6_18SII_F_H05_WSBS-Seq-1-08-15.ab1 слишком большой шум.
Ниже приведена картинка с исправлеными нуклеотидами(написаны прописными буквами).
 последовательности в Fasta-формате:
1. Ae4_18SII_R_G06_WSBS-Seq-1-08-15
2. Ae4_18SII_F_G05_WSBS-Seq-1-08-15
проект выравнивания:
ссылка
последовательности в Fasta-формате:
1. Ae4_18SII_R_G06_WSBS-Seq-1-08-15
2. Ae4_18SII_F_G05_WSBS-Seq-1-08-15
проект выравнивания:
ссылка

 Хроматограмма является нечитаемой, так как пики размытые и непериодичные, сигнал шума на слишком высоком уровне, поэтому
невозможно установить нуклеотидную последовательность.
Хроматограмма является нечитаемой, так как пики размытые и непериодичные, сигнал шума на слишком высоком уровне, поэтому
невозможно установить нуклеотидную последовательность.