6. Поиск по сходству (нуклеотидный blast)
Задание 1
Полученная в ходе исследования результатов капиллярного секвенирования ссылка последовательность была направлена на сравнение с другими последовательностями
в базе данных с помощью нуклеотидного BLAST для установления таксономии и определения функции гена.
На картинке ниже представлены находки программы.
 Из результатов видно, что для луxших находок E-value = 0, а процент идентичности 97%, из чего можно однозначен утверждать, что организм, к которому
относится последовательность имеет следующую таксономию до рода: Eukaryota; Metazoa; Lophotrochozoa; Annelida; Polychaeta;
Polychaeta incertae sedis; Saccocirridae;
При этом существует 2 рода (Pharyngocirrus и Saccocirrus), содержащий мою последовательность.
Последовательность кодирует 18S рибосамальную РНК.
Из результатов видно, что для луxших находок E-value = 0, а процент идентичности 97%, из чего можно однозначен утверждать, что организм, к которому
относится последовательность имеет следующую таксономию до рода: Eukaryota; Metazoa; Lophotrochozoa; Annelida; Polychaeta;
Polychaeta incertae sedis; Saccocirridae;
При этом существует 2 рода (Pharyngocirrus и Saccocirrus), содержащий мою последовательность.
Последовательность кодирует 18S рибосамальную РНК.
Задание 2
При поиске последовательностей, гомологичных последовательности из предыдущего задания.
Плодотворной идеей для снижения числа находок до обозримого оказалось ограничить поиск таксоном Bacteria. Поиск по базе nr (в Refseq ничего не ищется), галочки в полях "Exclude Uncultured/environmental sample sequences" и "Exclude Models (XM/XP)". Проделываем три поиска: Megablast со стандартными настройками, blastn co стандартными настройками, blastn с длиной слова 7 и Match/mismatch scores = 1;-1. На всякий случай везде меняем максимальное количество находок на 500. Получаем вот что:
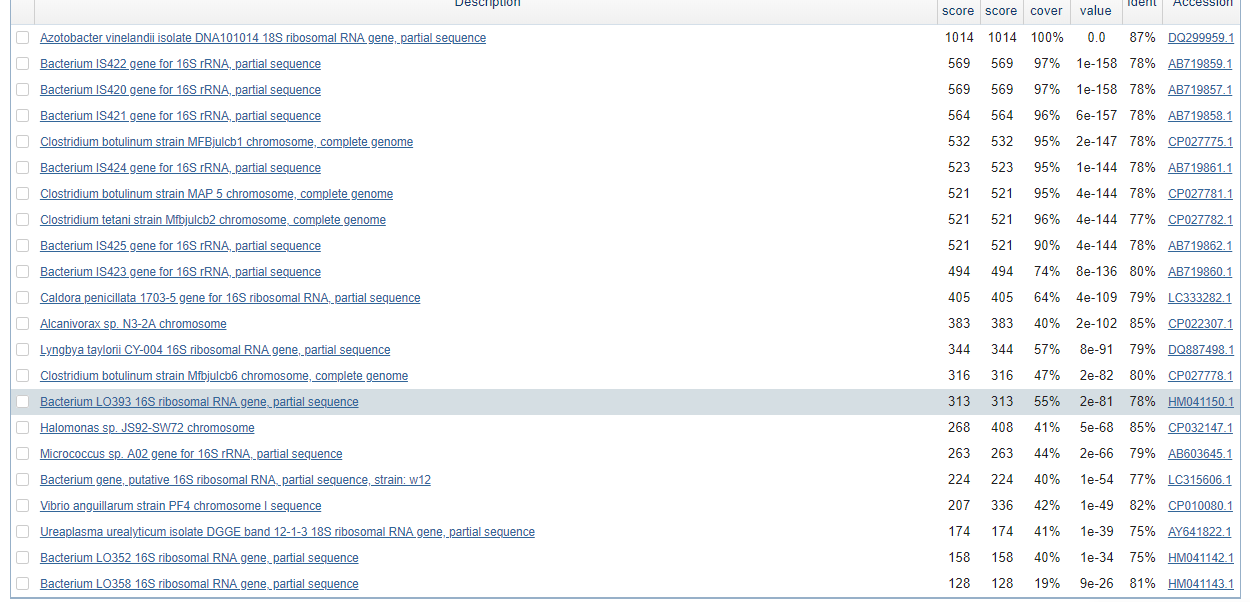
1.Blastn co стандартными настройками (64 находки)
 2.Megablast (22 находки)
2.Megablast (22 находки)
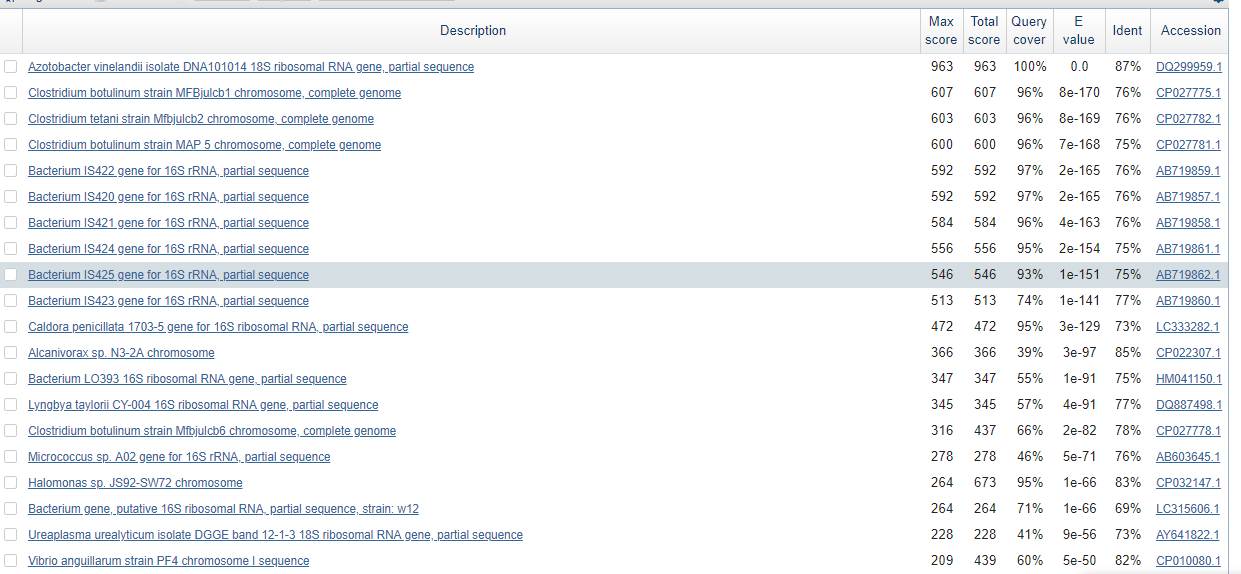
 3.Blastn c чувствительными параметрами(52 находки)
3.Blastn c чувствительными параметрами(52 находки)
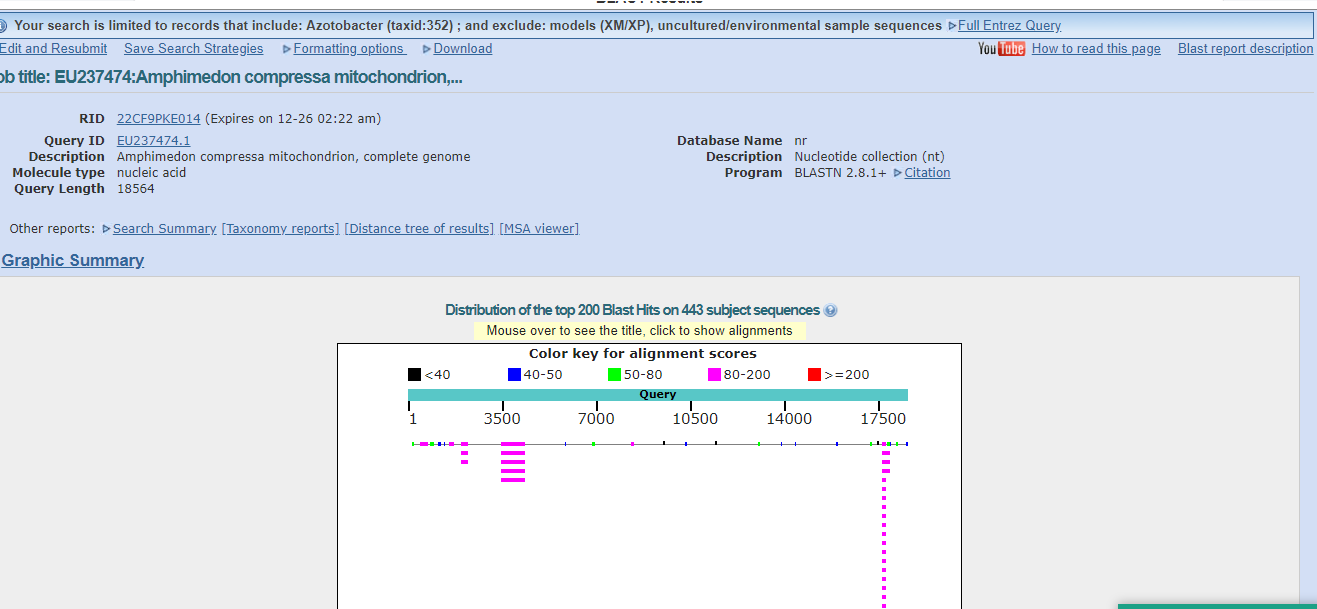
 Была взята последовательность из Sequence: EU237474.1 из tRNA 17141..17213 (/gene="trnF(gaa)",/product="tRNA-Phe",/anticodon=(pos:17174..17176,aa:Phe,seq:gaa))
Поиск по базе nr (в Refseq ничего не ищется), галочки в полях "Exclude Uncultured/environmental sample sequences" и "Exclude Models (XM/XP)", Azotobacter (taxid:352), Match/mismatch scores = 1;-1, изменялась только длина слова
Для неё BLASTN с длиной слова 7 дал результаты (443 находки):
Была взята последовательность из Sequence: EU237474.1 из tRNA 17141..17213 (/gene="trnF(gaa)",/product="tRNA-Phe",/anticodon=(pos:17174..17176,aa:Phe,seq:gaa))
Поиск по базе nr (в Refseq ничего не ищется), галочки в полях "Exclude Uncultured/environmental sample sequences" и "Exclude Models (XM/XP)", Azotobacter (taxid:352), Match/mismatch scores = 1;-1, изменялась только длина слова
Для неё BLASTN с длиной слова 7 дал результаты (443 находки):
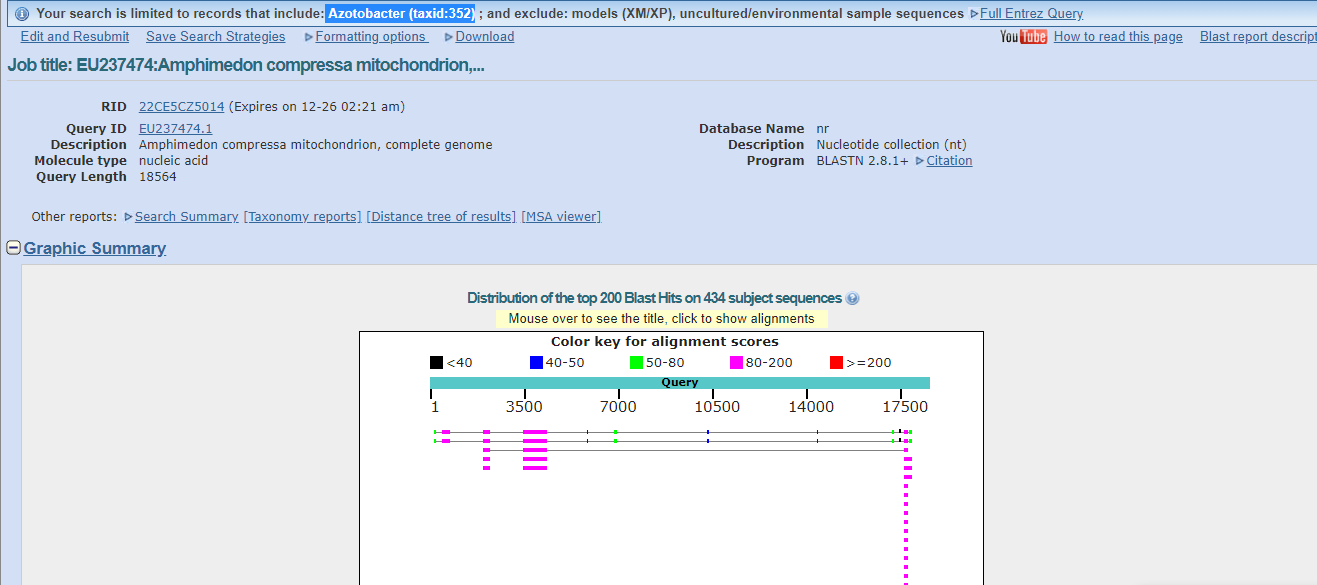
 с длиной слова 11 дал резульаты (434 находки):
с длиной слова 11 дал резульаты (434 находки):
 с длиной слова 15 дал резульаты (368 находки):
с длиной слова 15 дал резульаты (368 находки):
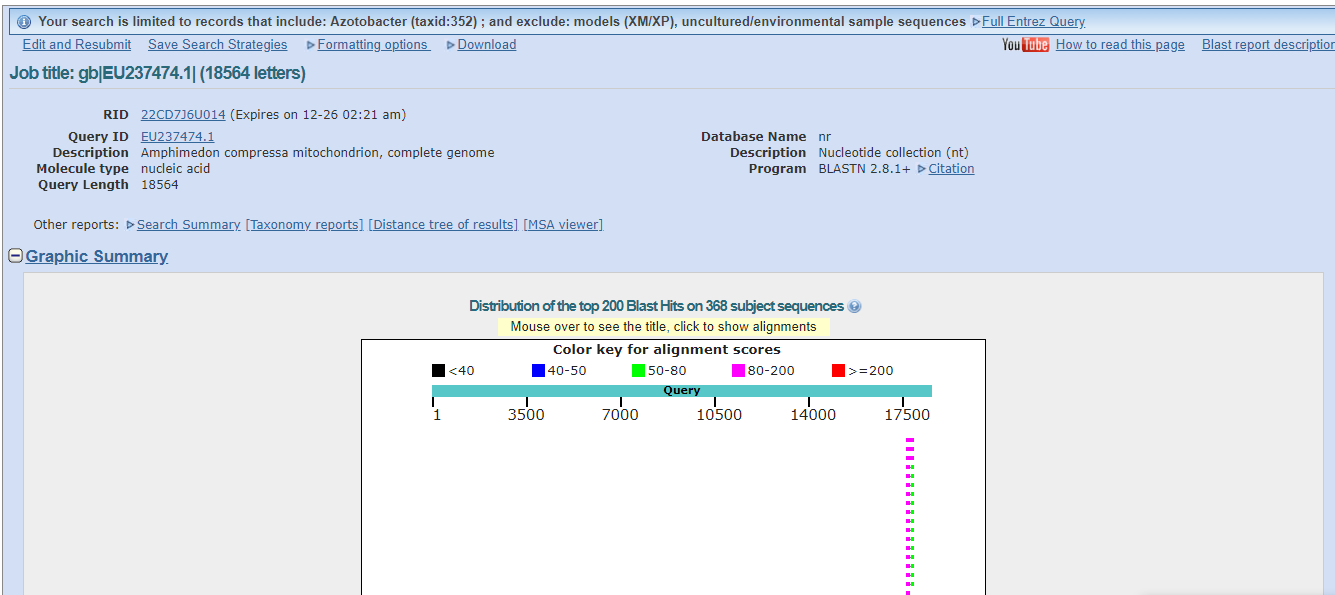 Сравнение алгоритмов демонстрирует, что чувствительность уменьшается при увеличении длины слова, а также чувствительность megablast меньше,чем
BLASTN, т.к. megablast даёт меньше находок.
Сравнение алгоритмов демонстрирует, что чувствительность уменьшается при увеличении длины слова, а также чувствительность megablast меньше,чем
BLASTN, т.к. megablast даёт меньше находок.
Задание 3
В этом задании была использована программа tblastn. Для белка HSP71_YEAST был файл найден гомолог scaffold-199
с параметрами сходства: Score = 920 bits (2377), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 481/609 (79%), Positives = 550/609 (90%), Gaps = 3/609 (0%). Вероятнее всего белок консервативен и выполняет ту же функцию, что
и HSP71_YEAST, который является белок теплового шока.
Для белка TERT_HUMAN был получен файл . Параметры для лучшего выравнивания: Score = 105 bits (263), Expect = 8e-23, Method: Compositional matrix adjust.
Identities = 151/568 (27%), Positives = 248/568 (44%), Gaps = 43/568 (8%)
Frame = +1.
Этот результат является условно положительным. BLAST выдал три находки, однако даже у самой лучшей из них параметры сходства слишком низкие, чтобы утверждать сохранение функций.
Гомология отдельных доменов также маловероятна, так как совпадения распределены по всей длине последовательности относительно равномерно.
Для белка CISY_HUMAN был получен файл . Параметры для лучшего выравнивания: Score = 565 bits (1457), Expect = 2e-180, Method: Compositional matrix adjust.
Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%)
Frame = +1
Скорее всего она действительно является гомологом интересующего нас белка с сохранением функций ввиду относительно хороших параметров сходства.
Задание 4
С помощью программы infoseq был получин континг unplaced-987, для которго был запущен blastx и получен ген белка XM_022029653.1.
Выравнивание имело было следующим:
Score Expect Method Identities Positives Gaps Frame
1055 bits(2727) 0.0 Compositional matrix adjust. 538/1009(53%) 707/1009(70%) 29/1009(2%) -3
Query 3162 ATMDQVNDLFSKLSVGKETAPRAQVASELVRLVKSSHDIHFLKSY*IIDKIKIEADNAKS 2983
AT DQ KE A + + + ++K+ HD + +K+ + +N K
Sbjct 14 ATQDQTTS-----EARKEQAEKVAEEVKNLGIIKAFHDGNLIKT------VTTLLENKKQ 62
Query 2982 APAREGAMLTFAALCKDV-PVCDPYLLPLLPIVLERMGDKA*EVRKAADEAGLAFIERVN 2806
AP RE A A++ K V +PYL+PL+P VL+ DK VR AADEA A + +
Sbjct 63 APFREAAYYILASVSKTVGQAGEPYLIPLVPKVLDGYADKVTSVRDAADEASKAIMALPS 122
Query 2805 PHAVKAVLPVLYEAIEHGIKWQTKFGGLALLRALTKKAP**IRLCLPDIIPHASAAMWDT 2626
+AVK +LPVL+ +IE+G +WQ++ G L LL L+K +P I CL +I+P SA+MW T
Sbjct 123 RYAVKLLLPVLFASIENG-RWQSQVGSLQLLAGLSKSSPKQISKCLSEIVPVLSASMWST 181
Query 2625 KSEVNKSAAEAMAELCKLVGNPDIEAFIPALIRTIANPSEVPECVYQLAATTFVTTVESP 2446
+ EV A + ++VGNPD+ + IP L+ I P EVPEC++QLA+TTFVTTVE+P
Sbjct 182 RPEVRAEATKTTTACFEVVGNPDLISSIPYLVGCINRPEEVPECIHQLASTTFVTTVEAP 241
Query 2445 ALAIMLPLLVRGLGESVTAVKRQTAVIIDNMCKLVLDPAQAEFFIPKLLPGLDRIIEVAA 2266
LAIM PLL+RGL E +++RQTAVIIDNMCKLV +PA A F+PKLLPGLDR+IE+ A
Sbjct 242 TLAIMTPLLIRGLAERTPSIQRQTAVIIDNMCKLVENPAHAHQFLPKLLPGLDRLIEIGA 301
Query 2265 DPDLRSVAERARKTMLKAGGFDHKLQASDIQVAVKWVSNVDNLMKVLKDIVSAQAPSHKD 2086
P+LRSVAERAR T+++ GG + + S + +A + N +++ LK + +
Sbjct 302 SPELRSVAERARATLIRVGGGEKAQEESILNIAYEIKPN--EVLETLKKTIGSSIKV--- 356
Query 2085 EMKD*FFVILL*FVCTLASELVEHKCFEEAEWVATIKPYLSAFLSADESANCAKEFLKVS 1906
D F L + TL SEL+ + FE W A+I PYL F+S DE+ A K
Sbjct 357 ---DDFVQTSLSYSATLCSELITSRDFESDAWDASITPYLLTFISKDEAKRIATSVHKFY 413
Query 1905 LENIQKeteeeeeeeeegED--LCDCEFSLAYGGMILLNSTKLHLKRGHRYGLCGANGVG 1732
++ K + E LCDCEFSLAYGGMILLN T+L+L+RG RYGLCG NGVG
Sbjct 414 VDYDAKNALSNAAVADVEEGELLCDCEFSLAYGGMILLNKTRLNLRRGQRYGLCGPNGVG 473
Query 1731 KSTLMRAIANGQLDGFPPQDVLKSVFVEHKL*ASDAEILIMDFVQNDDMTKDVEKDVILK 1552
KSTLMRAIA+GQL+GFPP D L++VFVEH L A +A++ +++F+ D D+ + ++
Sbjct 474 KSTLMRAIADGQLEGFPPADELRTVFVEHNLQAEEADLPVVEFMFADPKLSDIPHEEVVS 533
Query 1551 TLESVGFDEEMRAKKVGELSGGWKMKLELARAIMVNADILLLDEPTNHLDKRNVKWLTDY 1372
L SVGF M+ + VG LSGGWKMKLELARA+++NADILLLDEPTNHLD NV WL Y
Sbjct 534 RLSSVGFTPAMQQQAVGSLSGGWKMKLELARAMLMNADILLLDEPTNHLDVHNVAWLESY 593
Query 1371 LNSLSNVTSLIVSHDSTFLDNVCTDILHYENRKLKRYRGNLSEFVKIRPEAKAYYNLTQS 1192
L SL+NVTS+IVSHDS+FLDNVCT I+HYE+RKLK+YRGNLS+FV+ P+AK+YY L S
Sbjct 594 LTSLTNVTSMIVSHDSSFLDNVCTGIIHYESRKLKKYRGNLSKFVEQYPDAKSYYELKSS 653
Query 1191 QYNFKLPEPGFLDGVNTKDKAIIKLVRVGFRYPGSDKDQLSGVSVACSLSSRIAVLGPNG 1012
FKLPEPGFLDGV +KDKA++K + + F YPG+ + +S SL+SR+AV+GPNG
Sbjct 654 LITFKLPEPGFLDGVKSKDKALLKFINISFTYPGNTTPTIRNMSAQVSLNSRVAVVGPNG 713
Query 1011 AGKSTLIKVLTGETIPTAGDVIRHPNLRICYVAQHAFHHLDRHLDKTPNQYIQWRYQFGE 832
AGKSTLIKVLTGETIP G+V +HPNLR+ YVAQHAFHH+++HL KTPN+YI+WRYQFGE
Sbjct 714 AGKSTLIKVLTGETIPQVGEVTKHPNLRVAYVAQHAFHHVEQHLTKTPNEYIRWRYQFGE 773
Query 831 DKELLEKESRQMTDAD----KKVFEQ*VVFDG*KRKLESLMGRRKLKKSFEYEVKWVNMT 664
D+EL K SRQ++ + KKV + + K +++ L GRRK K+SFEYEV+WV T
Sbjct 774 DRELAAKASRQISPEEEAQMKKVIQWEINGKMEKLQIDDLYGRRKAKRSFEYEVQWVGRT 833
Query 663 HEENSWVPRDKLEEWGFNKIV*KFDDKEAAKAGAYTRPLTAANVQKHLEDLGLDPEFATH 484
+++N+W+ R+KLEEWGF K++ FDDKEAA+AGA+TR LTA V+KHL DLGL EFATH
Sbjct 834 YDDNAWISREKLEEWGFEKLLQSFDDKEAARAGAWTRSLTAVEVEKHLGDLGLPAEFATH 893
Query 483 SFIKGLSGGQKVKVVIGASTWNNPHMLVLDEPSNYLDRDSLAALSQALKEFGGGVIVISH 304
+ IKGLSGGQKVKVV+ A+ W NPH+LVLDEP+NYLDRDSL AL++AL+EFGGGV++ISH
Sbjct 894 NHIKGLSGGQKVKVVLAAAMWLNPHILVLDEPTNYLDRDSLGALTEALREFGGGVVIISH 953
Query 303 NQEFVDAVCTERWNMDNGLL*IEGQ--TTAvkekiv*keeeemVDAFGN 163
+++F +A+CTE W+++ G L + G T V+ KIV KE E +DAFGN
Sbjct 954 HRDFTEAICTETWSINAGELTVTGNNYTQRVESKIVQKEAETKIDAFGN 1002
Ген, кодирует P-петлю, содержащую нуклеозидтрифосфат гидролазный белок. В выравнивании присутствуют консервативные участки, на основании чего
можно предположить, что белок выполяняет ту же функцию и в исходном организме.
Задание 5
В этом задании было взято 2 генома NC_015408.1 Chlamydophila pecorum E58, complete genome (lcl|Query_55631)
и NC_017287.1 Chlamydia psittaci 6BC, complete genome (lcl|Query_55633) и при помощи blastn была получена карта локального сходства
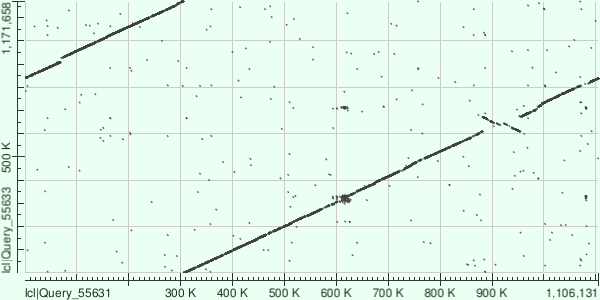 Можно наблюдать инверсию, которая соответствует нуклеотидам 600К -700К для Query_55633
и 850К-950К для Query_55631.
Можно наблюдать инверсию, которая соответствует нуклеотидам 600К -700К для Query_55633
и 850К-950К для Query_55631.
 Из результатов видно, что для луxших находок E-value = 0, а процент идентичности 97%, из чего можно однозначен утверждать, что организм, к которому
относится последовательность имеет следующую таксономию до рода: Eukaryota; Metazoa; Lophotrochozoa; Annelida; Polychaeta;
Polychaeta incertae sedis; Saccocirridae;
При этом существует 2 рода (Pharyngocirrus и Saccocirrus), содержащий мою последовательность.
Последовательность кодирует 18S рибосамальную РНК.
Из результатов видно, что для луxших находок E-value = 0, а процент идентичности 97%, из чего можно однозначен утверждать, что организм, к которому
относится последовательность имеет следующую таксономию до рода: Eukaryota; Metazoa; Lophotrochozoa; Annelida; Polychaeta;
Polychaeta incertae sedis; Saccocirridae;
При этом существует 2 рода (Pharyngocirrus и Saccocirrus), содержащий мою последовательность.
Последовательность кодирует 18S рибосамальную РНК.
 2.Megablast (22 находки)
2.Megablast (22 находки)
 3.Blastn c чувствительными параметрами(52 находки)
3.Blastn c чувствительными параметрами(52 находки)
 Была взята последовательность из Sequence: EU237474.1 из tRNA 17141..17213 (/gene="trnF(gaa)",/product="tRNA-Phe",/anticodon=(pos:17174..17176,aa:Phe,seq:gaa))
Поиск по базе nr (в Refseq ничего не ищется), галочки в полях "Exclude Uncultured/environmental sample sequences" и "Exclude Models (XM/XP)", Azotobacter (taxid:352), Match/mismatch scores = 1;-1, изменялась только длина слова
Для неё BLASTN с длиной слова 7 дал результаты (443 находки):
Была взята последовательность из Sequence: EU237474.1 из tRNA 17141..17213 (/gene="trnF(gaa)",/product="tRNA-Phe",/anticodon=(pos:17174..17176,aa:Phe,seq:gaa))
Поиск по базе nr (в Refseq ничего не ищется), галочки в полях "Exclude Uncultured/environmental sample sequences" и "Exclude Models (XM/XP)", Azotobacter (taxid:352), Match/mismatch scores = 1;-1, изменялась только длина слова
Для неё BLASTN с длиной слова 7 дал результаты (443 находки):
 с длиной слова 11 дал резульаты (434 находки):
с длиной слова 11 дал резульаты (434 находки):
 с длиной слова 15 дал резульаты (368 находки):
с длиной слова 15 дал резульаты (368 находки):
 Сравнение алгоритмов демонстрирует, что чувствительность уменьшается при увеличении длины слова, а также чувствительность megablast меньше,чем
BLASTN, т.к. megablast даёт меньше находок.
Сравнение алгоритмов демонстрирует, что чувствительность уменьшается при увеличении длины слова, а также чувствительность megablast меньше,чем
BLASTN, т.к. megablast даёт меньше находок.
 Можно наблюдать инверсию, которая соответствует нуклеотидам 600К -700К для Query_55633
и 850К-950К для Query_55631.
Можно наблюдать инверсию, которая соответствует нуклеотидам 600К -700К для Query_55633
и 850К-950К для Query_55631.