Комплексы ДНК-белок
Задание 1
В этом задании мы предсказали вторичную структуру тРНК (2FMT) тремя способами:
1) С помощью find_pair из предыдущего практикума
2) Программой einverted из пакета EMBOSS, наилучший из полученных результатов представлен ниже.
SEQUENCE: Score 116: 24/28 ( 85%) matches, 12 gaps
1 ggggtatc---g---ccaa--gcggtaaggcaccggattc 32
|||||| | | | || ||| || | ||||| ||
69 ccccat-gctcctaagcttggagcc-ttac--ggccttag 34
3) RNAfold из пакета Viena Rna Package, реализующей алгоритм Зукера
С основными результатами вы можете ознакомиться в приведенной ниже таблице
______________________________________________________________________________________________________________________________________________________________________
Участок структуры Позиции в структуре (find_pair) Результаты предсказания с помощью einverted Результаты предсказания по алгоритму Зукера
______________________________________________________________________________________________________________________________________________________________________
Акцепторный стебель (6/7) (6/7) (6/7)
5'- 2 - 7 - 3' 1 ggggta 6
5'- 66 - 71 - 3' ||||||
69 ccccat 64
______________________________________________________________________________________________________________________________________________________________________
D-стебель (3[4]/3) (0/3) (3/3)
5'- 10 - 12 - 3'
5'- 23 - 25 - 3'
_______________________________________________________________________________________________________________________________________________________________________
T-стебель (5[4]/5) (0/5) (5/5)
5'- 49 - 53 - 3'
5'- 61 - 65 - 3'
_______________________________________________________________________________________________________________________________________________________________________
Антикодоновый стебель (5/5) (5/5) (5/5)
5'- 39 - 43 - 3' 24 ccgga 29
5'- 27 - 31 - 3' |||||
35 ggcct 30
_______________________________________________________________________________________________________________________________________________________________________
Общее число канонических пар нуклеотидов 20 11 20
_______________________________________________________________________________________________________________________________________________________________________
Мы можем заметить, что наиболее эффективными в предсказывнии вторичной РНК-структуры были программы
find_pair и RNAfold; но даже здесь есть некоторые неточности.
Программа einverted "смогла" найти только акцепторный стебель и антикодонный стебель.
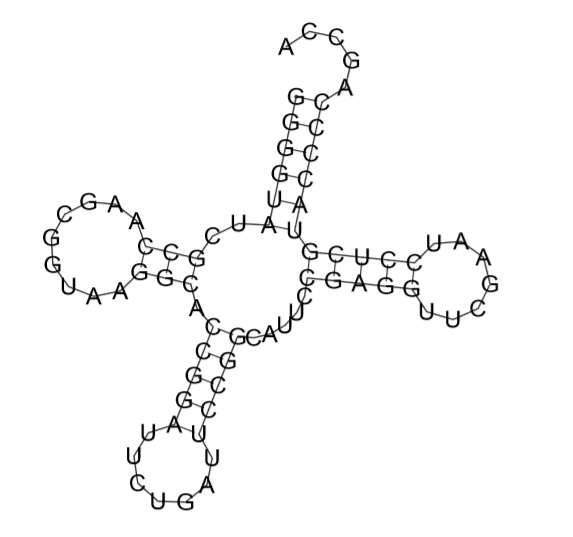 Результаты предсказания по алгоритму Зукера
Результаты предсказания по алгоритму Зукера
Задание 2
Упражениние 1.
Скрипт из пункта 4 ссылка
Скрипт из пункта 5 ссылка
Упражениние 2.
Со скриптом можно ознакомиться, перейдя по ссылке
Таблица. Контакты разного типа в комплексе 1RH6.pdb
______________________________________________________________________________________________
Контакты атомов белка с Полярные Неполярные Всего
______________________________________________________________________________________________
остатками 2'-дезоксирибозы 4 22 26
______________________________________________________________________________________________
остатками фосфорной кислоты 11 17 28
______________________________________________________________________________________________
статками азотистых оснований
со стороны большой бороздки 3 3 6
______________________________________________________________________________________________
остатками азотистых оснований
со стороны малой бороздки 1 0 1
______________________________________________________________________________________________
Из таблицы видно, что неполярных контактов наблюдается более, чем в 2 раза, больше полярных. Соответсвенно,
гидрофобных взаимодействий колическтвенно больше. Также на основании результатов можно сделать вывод, что
остатки азотистых оснований вносят наименьший вклад в взаимодействие ДНК и белка, что логично, так как азотистые
основания находятся с внутренней стороны спирали ДНК. Больше всего полярных взаимодействий с атомами остатков
фосфорной кислоты, что тоже логично, так как они находятся с внешней стороны спирали. В целом, с учётом
используемых приближений, были получены вполне закономерные результаты, поэтому данных подход изучения взимодействия
ДНК и белка может быть применим для грубых оценок.
Упражениние 3.
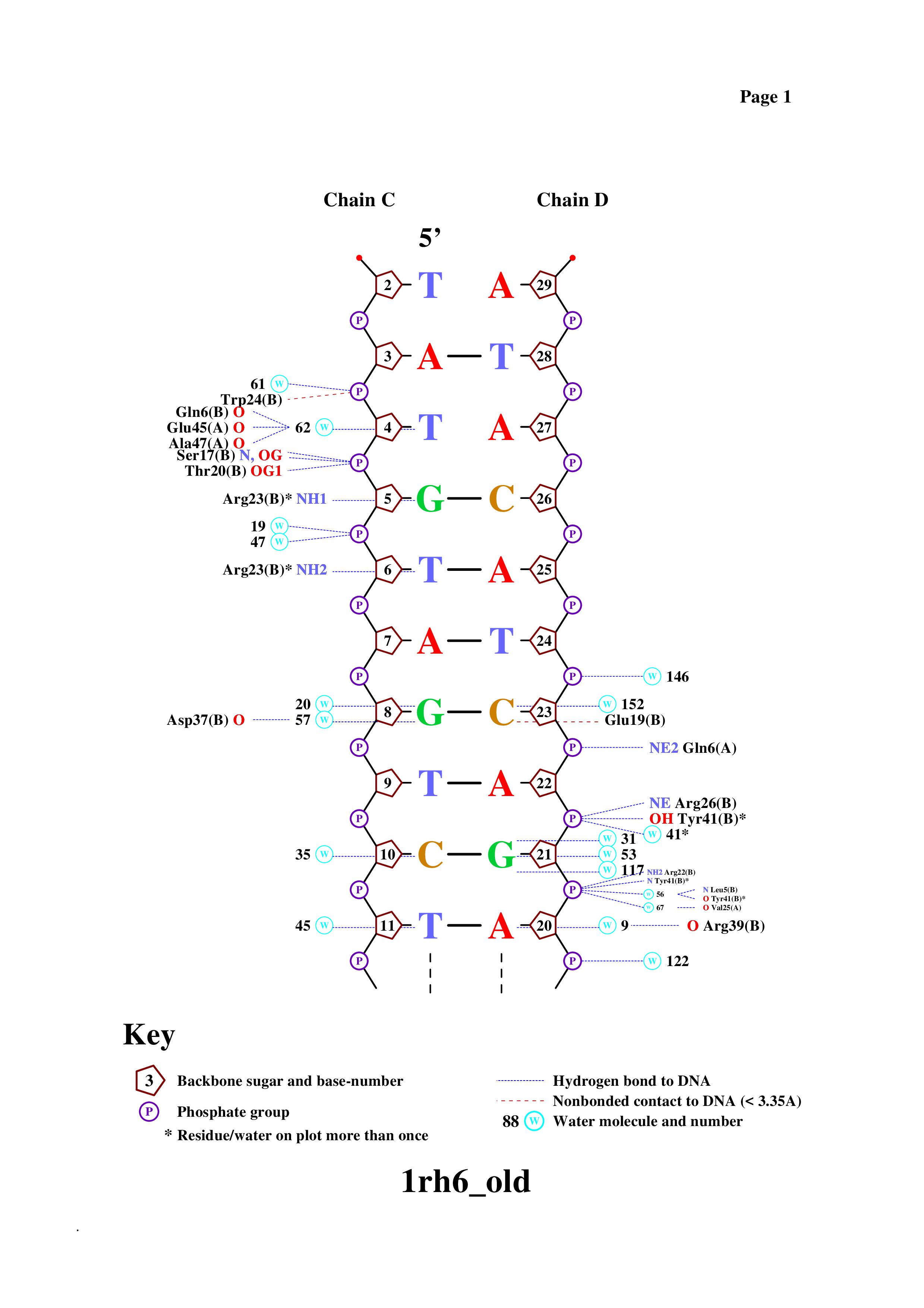
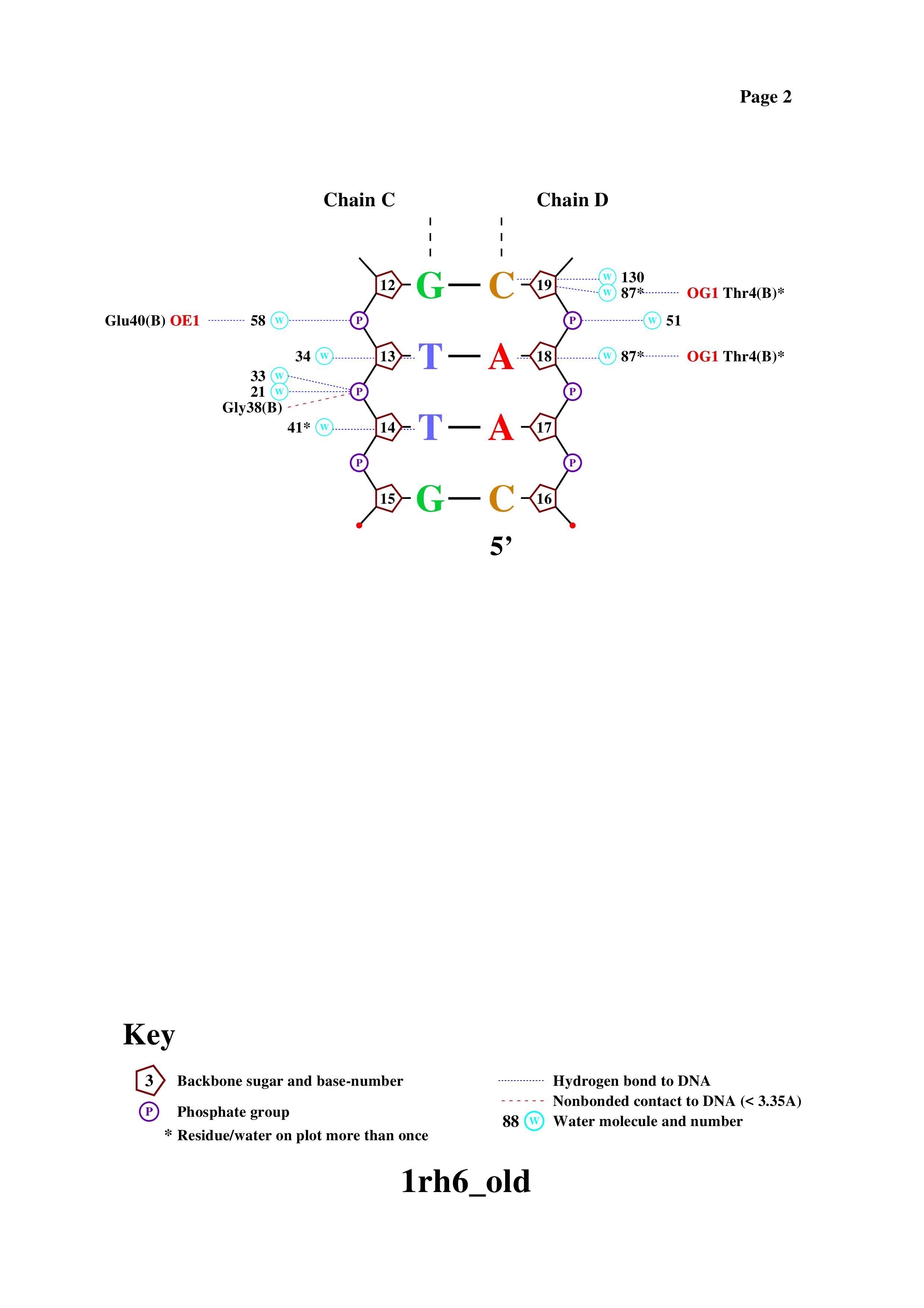
Упражениние 4.
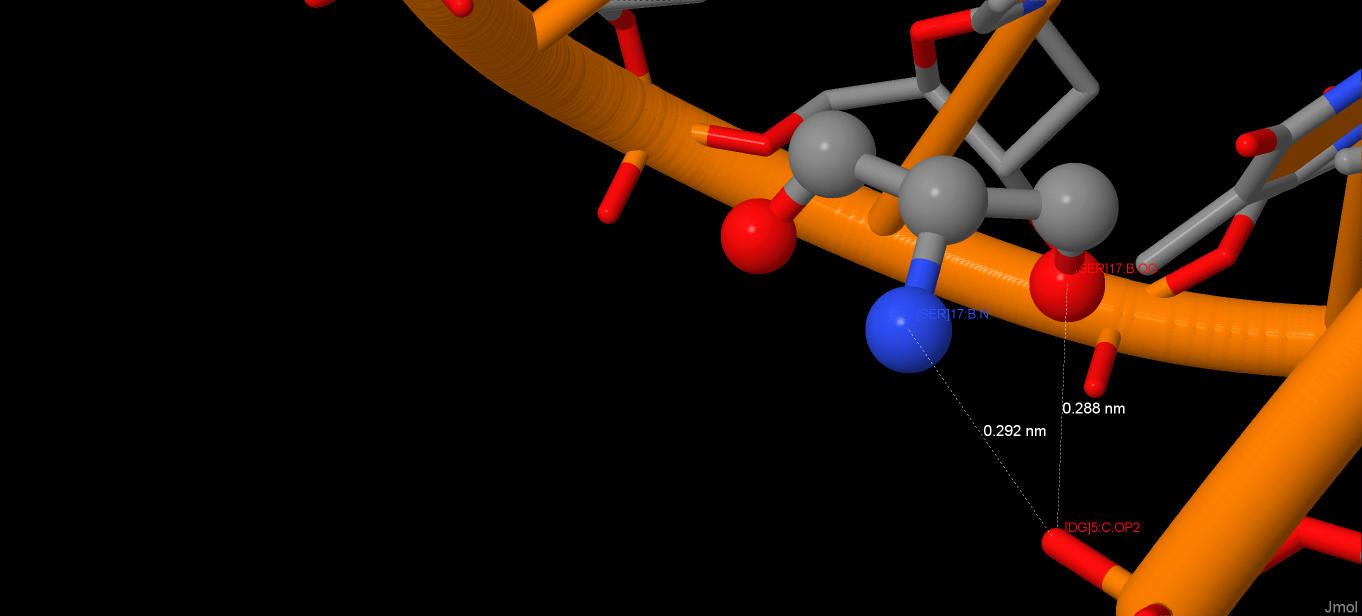
Было получено 3 аминокислотных остатка, которые имеют по 2 водородные связи с ДНК:
1. SER17
Donor Acceptor Distance
SER B 17 N G C 5 O2P 2.92
SER B 17 OG G C 5 O2P 2.88
на картинке ниже приведено его взаимодействие с ДНК:
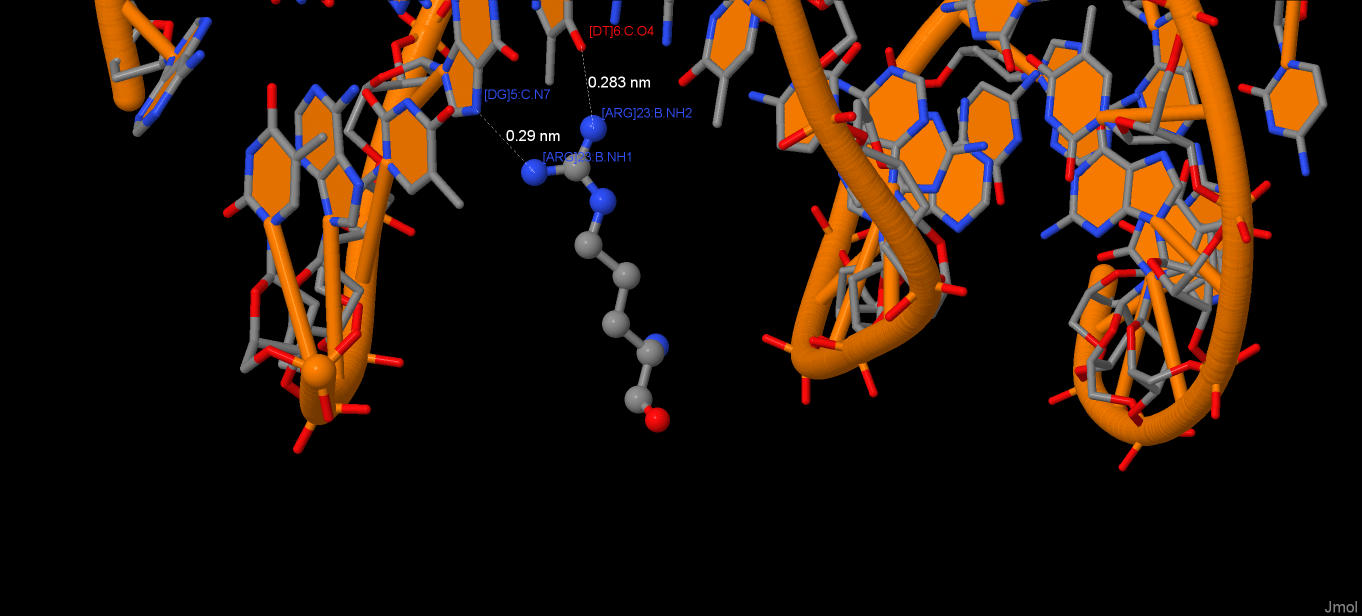
 2. ARG23
Donor Acceptor Distance
ARG B 23 NH1 G C 5 N7 2.90
ARG B 23 NH2 T C 6 O4 2.83
на картинке ниже приведено его взаимодействие с ДНК:
2. ARG23
Donor Acceptor Distance
ARG B 23 NH1 G C 5 N7 2.90
ARG B 23 NH2 T C 6 O4 2.83
на картинке ниже приведено его взаимодействие с ДНК:
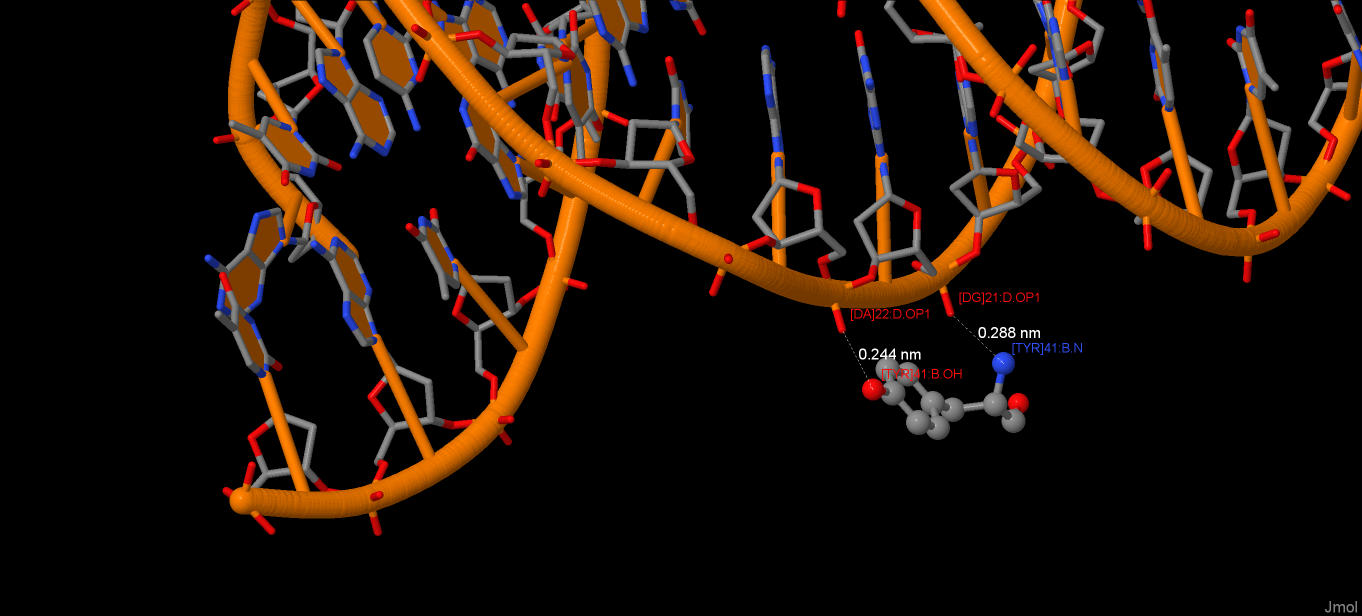
 3. TYR41
Donor Acceptor Distance
TYR B 41 N G D 21 O1P 2.88
TYR B 41 OH A D 22 O1P 2.44
на картинке ниже приведено его взаимодействие с ДНК:
3. TYR41
Donor Acceptor Distance
TYR B 41 N G D 21 O1P 2.88
TYR B 41 OH A D 22 O1P 2.44
на картинке ниже приведено его взаимодействие с ДНК:
 На мой взгляд, наиболее важным для распознования последовательности ДНК является ARG23, так как он связан водородными взаимодействиями
сразу с двумя нуклеотидами, что гарантирует высокую специфичность распознования и дальнейшего связывания.
На мой взгляд, наиболее важным для распознования последовательности ДНК является ARG23, так как он связан водородными взаимодействиями
сразу с двумя нуклеотидами, что гарантирует высокую специфичность распознования и дальнейшего связывания.
 Результаты предсказания по алгоритму Зукера
Результаты предсказания по алгоритму Зукера
 Результаты предсказания по алгоритму Зукера
Результаты предсказания по алгоритму Зукера


 2. ARG23
Donor Acceptor Distance
ARG B 23 NH1 G C 5 N7 2.90
ARG B 23 NH2 T C 6 O4 2.83
на картинке ниже приведено его взаимодействие с ДНК:
2. ARG23
Donor Acceptor Distance
ARG B 23 NH1 G C 5 N7 2.90
ARG B 23 NH2 T C 6 O4 2.83
на картинке ниже приведено его взаимодействие с ДНК:
 3. TYR41
Donor Acceptor Distance
TYR B 41 N G D 21 O1P 2.88
TYR B 41 OH A D 22 O1P 2.44
на картинке ниже приведено его взаимодействие с ДНК:
3. TYR41
Donor Acceptor Distance
TYR B 41 N G D 21 O1P 2.88
TYR B 41 OH A D 22 O1P 2.44
на картинке ниже приведено его взаимодействие с ДНК:
 На мой взгляд, наиболее важным для распознования последовательности ДНК является ARG23, так как он связан водородными взаимодействиями
сразу с двумя нуклеотидами, что гарантирует высокую специфичность распознования и дальнейшего связывания.
На мой взгляд, наиболее важным для распознования последовательности ДНК является ARG23, так как он связан водородными взаимодействиями
сразу с двумя нуклеотидами, что гарантирует высокую специфичность распознования и дальнейшего связывания.