Секвенирование по Сэнгеру
1. Получить последовательность ДНК на основании данных, полученных из капиллярного секвенатора. Составить отчёт о проблемах при чтении хроматограмм
1. Получить последовательность ДНК на основании данных, полученных из капиллярного секвенатора. Составить отчёт о проблемах при чтении хроматограмм
Прямое прочтение после изменения
Обратное прочтение после изменения
Последовательность без редактирования
Последовательность c редактированием
Сборка (до редактирования ошибок)
Выравнивание после редактирования
Для практикума я использовала программу UGENE. В ней в разделе "секвенирование по Сенгру", мои последовательности не выравнивались, если я выбирала референсом прямое прочтение или консенсусную последовательность из выравнивания прямого и обратного. Такие же проблемы были в Pearl. Поэтому я решила через blast найти
и оценить все вокруг него. Получилось. Я продолжила работу в UGENE.
Изменение 1. В чтении обратной последовательности нуклеотид в позиции 73 не был определен, поэтому я заменила его на нуклеотид из позиции прямого прочтения, к тому же пик G на обратном чтении также значим.
Изменение 2.Далее рядом наблюдалась проблемная зона. Здесь очевидно произошли ошибки при чтении прямой последовательностии - размазанные и перекрывающиеся пики. Поэтому, чтобы устранить неопредленности я заменила по обратной цепи.
Изменение 3. В этой позиции на прямом чтении пики были довольно сближены, поэтому определить нуклеотид не удалось. Но на обратном чтении - однозачный пик G, поэтому и в прямой заменяю на G.
Изменение 4. Похожая на 3 ситуацию и здесь. Заменяю на С.
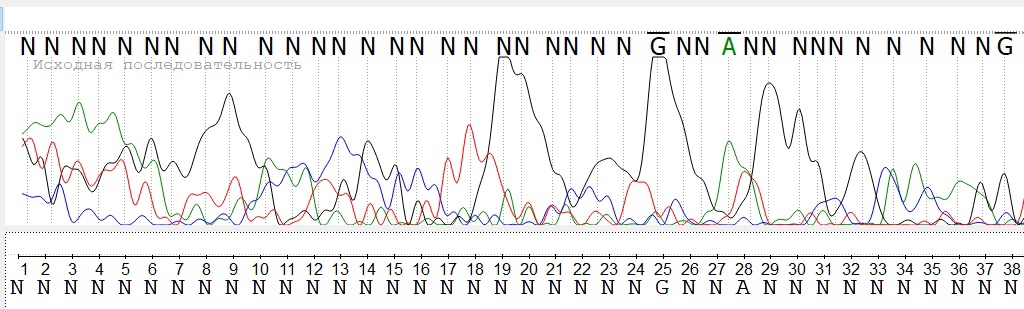
Длины начального и конечного трудно читаемых участков для прямого и обратного прочтений:
Начального: прямая цепь: 1-51, обратная цепь: 1-38
Конечного: прямая цепь: 378-383, обратная цепь: 382-383
Шум в среднем несильный, но на концах хроматограммы сложно определить последовательность. В середине последовательности 2-3 участка разрешимых неявных пиков.
2. Приведите пример нечитаемого фрагмента хроматограммы
Пример взят из обратного чтения моих последовательностей. Здесь смазаны пики и сложно четко выделить нуклеотиды