1. Определение функции и таксономии нуклеотидной последовательности
Анализируемая последовательность - последовательность, полученная секвенированием (практикум 6). Я использовала blastn, чтобы выяснить откуда эти последовательности. Blastn, потому что я анализирую нуклеотидную последовательнось, не знаю откуда она, поэтому пока не ввожу строгий критерий на идентичность. Я запускала со всеми стандартными настройками. Последовательность.
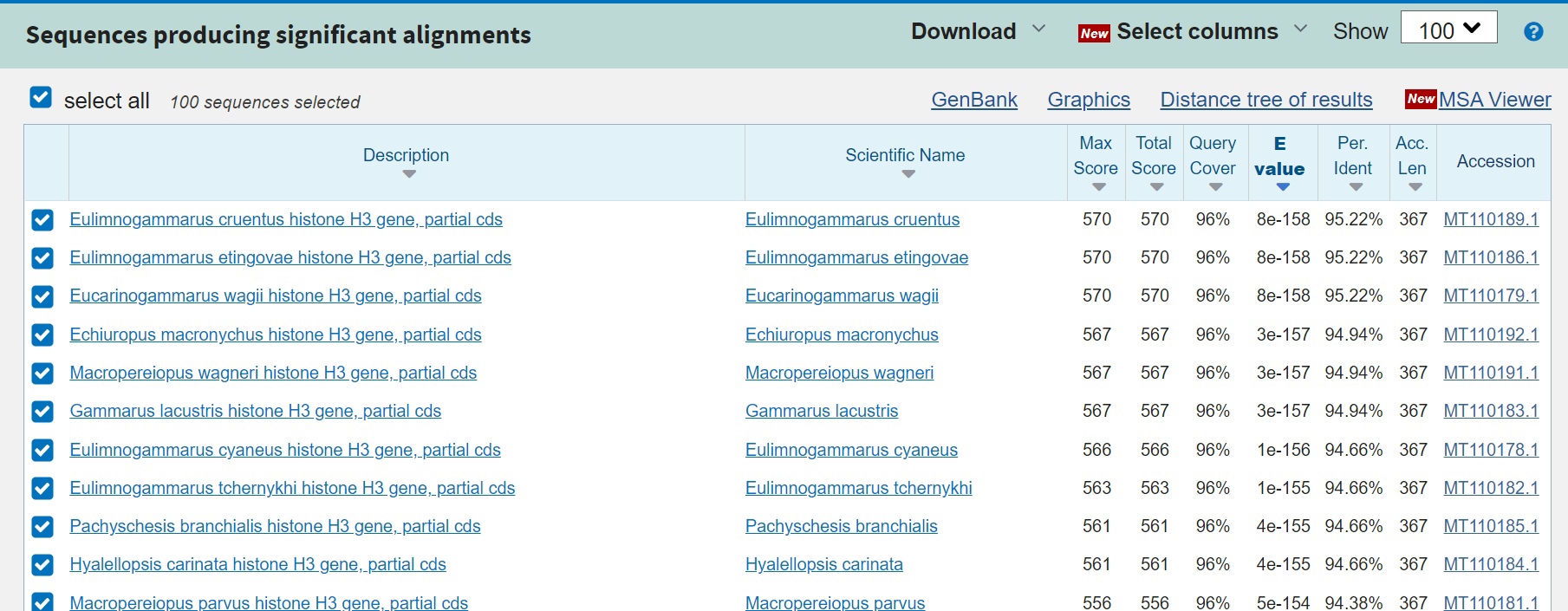
Оказалось, что это лучшее совпадение произошло с геном гистона H3 Eulimnogammarus cruentus, Eulimnogammarus etingovae, Eucarinogammarus wagii (per. ident = 95.22%, e-value = 5e-158). Значит, вероятно и наша отсеквенированная последовательность - кодирует гистон H3.
Размышления о таксономическом положении.
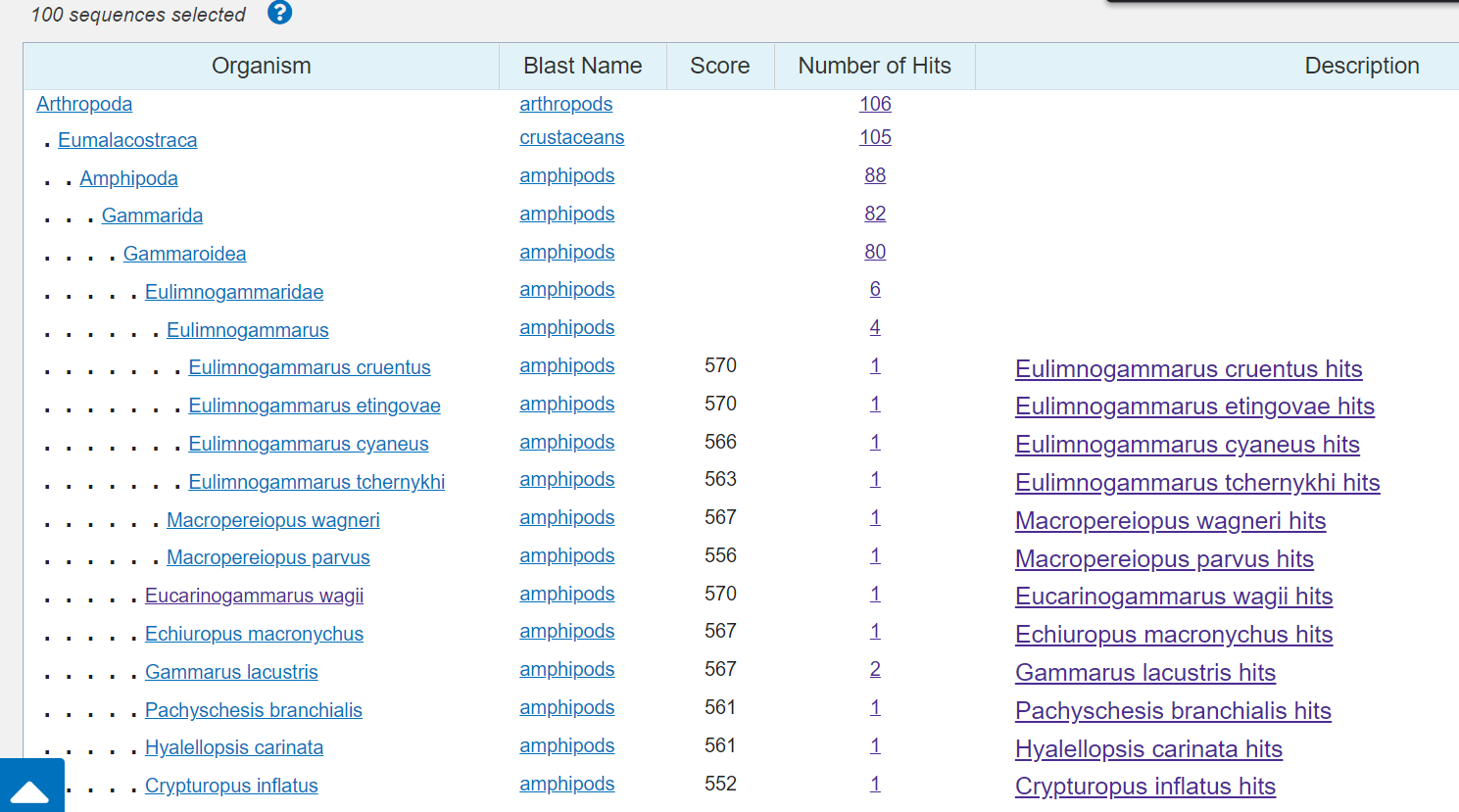
Посмотрим в раздел Taxonomy. В нем показано сколько находок приходится на определенную кладу. Из этого мы делаем вывод, что наша исследуемая последовательность скорее всего принадлежит к типу Arthropoda, подклассу Eumalacostraca, отряду Gammarida, семейству Gammaroidea. Дальше возможно принадлежит роду Eulimnogammarus. Если так, то с видом опредлиться тоже непросто, так как одинаковый score приходится на 2 разных вида - Eulimnogammarus cruentus и Eulimnogammarus etingovae. Такой же score показала находка из вида Eucarinogammarus wagii рода Eucarinogammarus wagii. Поэтому сложно сказать, какому конкретно из этих трех близкородственных организмов принадлежит находка.
Выдача Taxonomy.
2. Поиск генов белков в неаннотированной нуклеотидной последовательности
Я использовала контиг организма Pan troglodytes
Искала с помощью blastx, так как задача была найти гомологичные белки. Исключила из поиска организм Pan troglodytes (taxid:9598) и искала только в Mammals (taxid:40674).
В лучших 100 находках выдачи присутствуют в основном неохарактеризованные белки. Например, белки с AC EHH55556.1 (из Макаки),hCG1814203 (из человека). Из описанных - белок SPDYE1 (Speedy protein E1).У этой находки также самый низкий e-value (2e-37). У человека ген этого белка находится в критической области, приводящей к синдрому Вильямса-Бёрена (WBS).
Выдача первых 33 результатов blastx .
3. Интепретация карты локального сходства гомологичных хромосом двух бактерий
Для исследования я взяла последовательности хромосом двух видов рода Actinobacillus: Actinobacillus suis и Actinobacillus equuli.
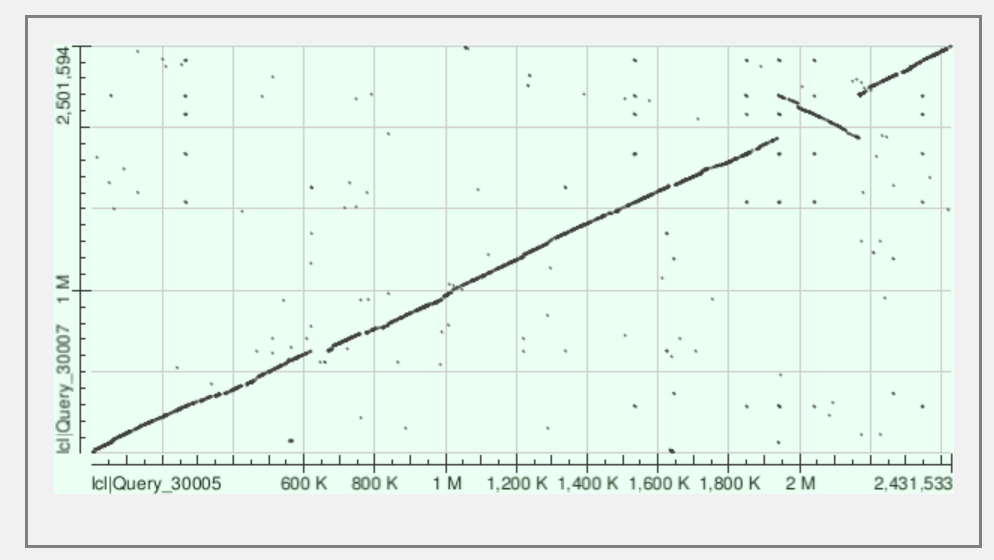
Видно, что геномы очень схожи. Из крупных перестроек - инверсия в области 1950К-2000К. Также, небольшие есть индели.