- Назад
- Страница первого семестра
Деноминация!
Стафилококк.
Кобыла и Трупоглазые жабы, "Войшвилло"
Обзор протеома бактерии Cronobacter dublinensis
Резюме
Бактерия Cronobacter dublinensis — распространнённая бактерия, обитающая в том числе и на пищевых продуктах. В данной работе используется функционал электронных таблиц для описания генома бактерии.
Ключевые слова: бактерии, геном, электронные таблицы
1. ВВЕДЕНИЕ
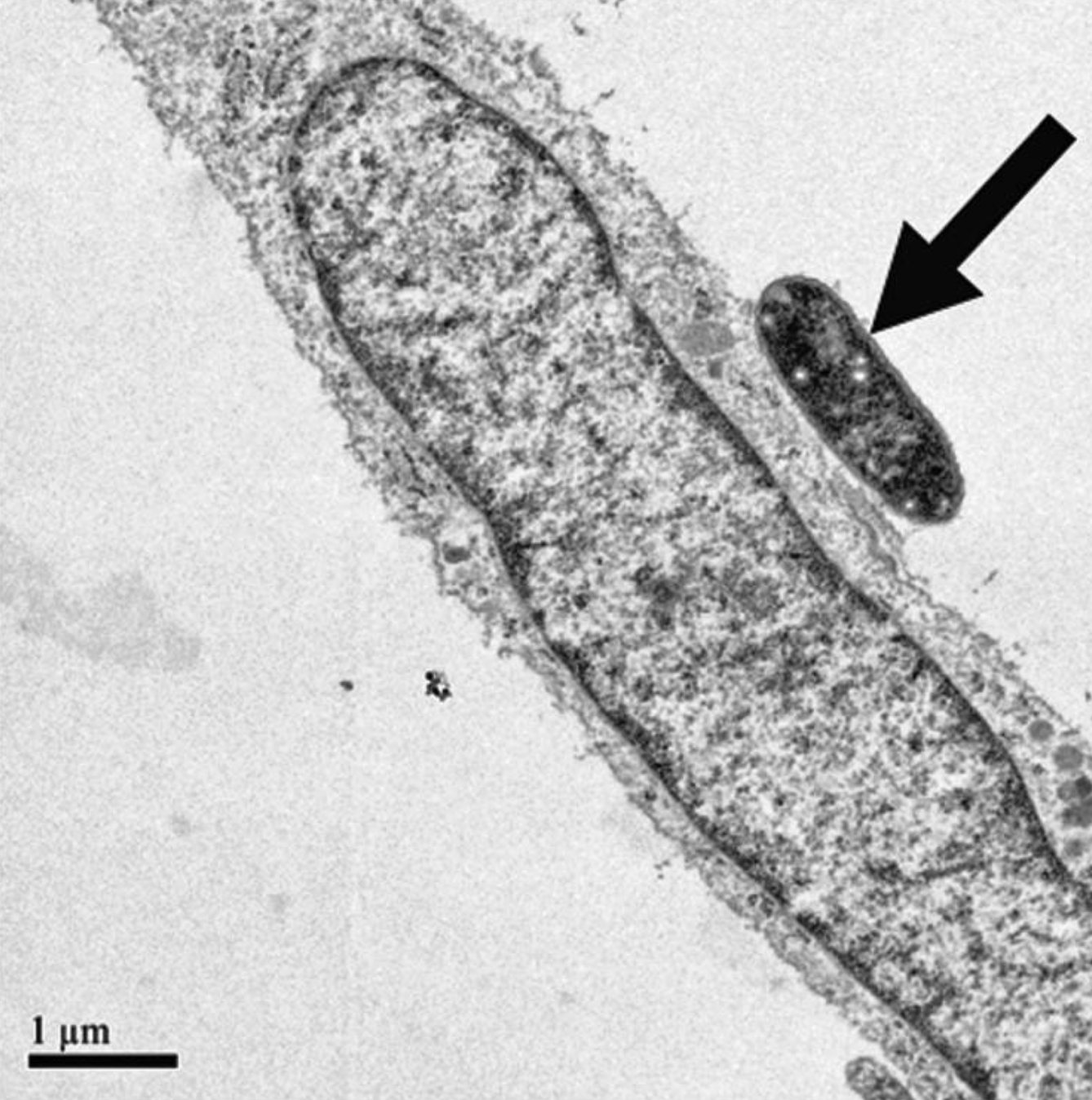
Бактерия Cronobacter dublinensis subsp. dublinensis LMG 23823 ранее относилась к виду Enterobacter sakazakii с большим количеством подвидов. В 2007 году этому виду дали ранг рода, а подвидам — вида в этом роде[1]. Бактерии этого рода патогенны и, предположительно, вызывают менингит и другие болезни у новорожденных детей[2], за что род и назвали Cronobacter, в честь Кроноса, титана из греческой мифологии, который съедал своих детей[2]. Видовое название dublinensis отсылается к Дублину, городу в котором этот штамм впервые был выделен[2].
Бактерии этого рода широко распространены, были найдены практически во всех пищевых продуктах[2].
Эти бактерии способны к адгезии и образованию биопленок на поверхности пищевых продуктов. Внутри организма-хозяина они способны прикрепляться к клеткам, внедряться внутрь и вызывать апоптоз[2].
В данной работе с помощью электронных таблиц был проведен анализ протеома C.dublinensis.
2. МАТЕРИАЛЫ И МЕТОДЫ
Информация о протеоме бактерии была взята из базы данных NCBI Genome, в работе использовался функционал Google Sheets.
1. Для общего подсчета генов разных типов применялась функция СЧЁТЕСЛИМН по параметрам из столбцов «class», «chromosome», «strand».
2. Для построения гистограммы длин белков применялась функция СЧЁТЕСЛИМН по столбцу «product_length».
3. Для подсчёта генов, расположенных на разных цепях применялась функция СЧЁТЕСЛИ по столбцу «strand». Для подсчета вероятности использовалась функция BINOM.DIST.
4. Для построения гистограммы длин межгенных промежутков была взята попарная разница между двумя соседними генами на любых цепях, а затем при помощи СЧЁТЕСЛИМН было посчитано, сколько в каждый «карман» попадает промежутков.
3. РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
3.1. Общая информация о геноме
Геном бактерии содержит 4377 генов, из которых 4113 на хромосоме и 264 на плазмиде. На прямой цепи хромосомы 1962 гена, на обратной — 2151, на плазмиде это соотношение составляет 168:96. Остальная информация содержится в таблицах 1, 2. В сопроводительных материалах ссылка на список названий всех рибосомальных белков и РНК.
| прямая цепь | обратная цепь | всего | % от всех генов | |
| белки | 1886 | 2076 | 3962 | 96.33% |
| рРНК | 13 | 19 | 22 | 0.53% |
| тРНК | 46 | 36 | 82 | 1.99% |
| псевдогены | 30 | 16 | 46 | 1.12% |
| гипотетические белки | 440 | 435 | 875 | 21.27% |
| рибосомальные белки | 21 | 41 | 62 | 1.51% |
| прямая цепь | обратная цепь | всего | % от всех генов | |
| белки | 154 | 94 | 248 | 93.94% |
| псевдогены | 30 | 16 | 46 | 17.42% |
| гипотетические белки | 49 | 22 | 71 | 26.89% |
| рибосомальные белки | 0 | 2 | 2 | 0.76% |
3.2. Гистограмма длин белков
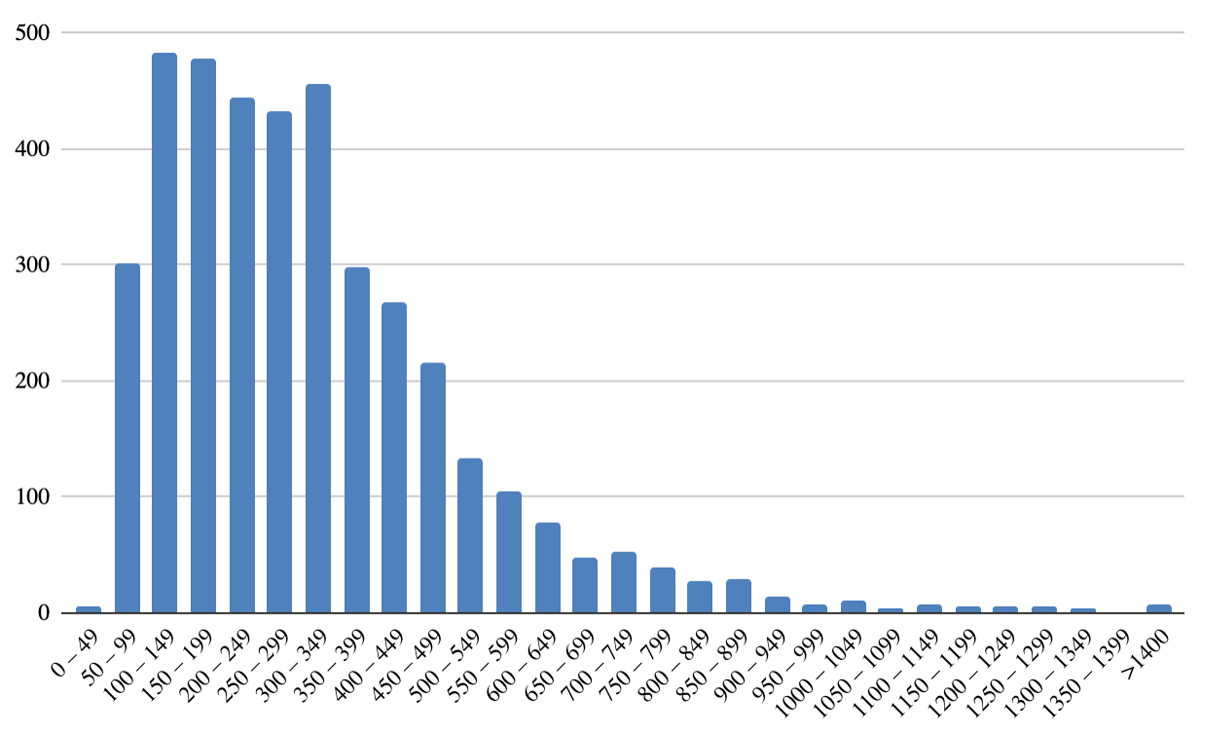
Гистограмма длин белков приведена на рисунке 2.
Из гистограммы видно, что наибольшее количество белков имеет длину 100-149 аминокислотных остатков.
После 150 с ростом длины белка количество белков тоже убывает, но есть три скачка: 1) 300–349, в этот промежуток попало больше белков, чем в промежутки 200–249 и 250–299; 2) 700–749, немного больше, чем в 650–699; 3) 850–899, небольшой скачок относительно предыдущего промежутка.
В протеоме этой бактерии классическое «двугорбое» распределение белков по длинам не наблюдается.
3.3. Случайное распределение генов по цепям
На прямой цепи хромосомы находится 1962 гена из 4113. Вероятность такого распределения или распределения с большей разницей составляет 0.16%, что не противоречит предположению о случайности распределения.
На обратной цепи плазмиды 96 генов из 264. Вероятность этого составляет 0.0000055, из чего можно сделать вывод, что на плазмиде гены распределены не случайно. Возможно, это связано с тем, что плазмида маленькая и гены в ней еще не успели «перераспределиться» в процессе эволюции C. dublinensis.
3.4. Гистограмма длин межгенных промежутков
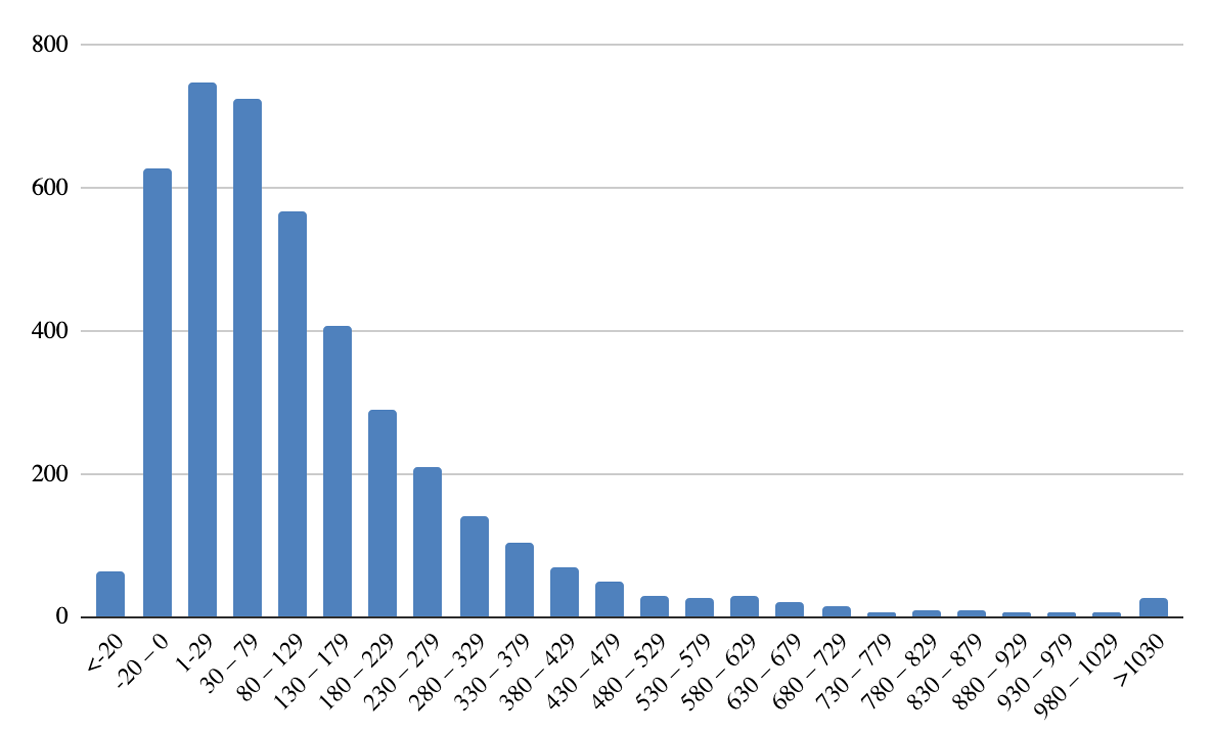
Гистограмма длин межгенных промежутков представлена на рисунке 3.
Существуют отрицательные межгенные промежутки, это означает, что гены перекрываются, и таких перекрывающихся генов довольно много.
В целом, из гистограммы видно, что большинство генов находятся на небольшом расстоянии друг от друга, пик приходится на промежутки длины от 1 до 29. После пика количество расстояний данной длины монотонно убывает, за исключением небольшого скачка на промежутке 780–829.
4. ЗАКЛЮЧЕНИЕ
Таким образом, Cronobacter dublinensis subsp. dublinensis LMG 23823 — это бактерия, которая представляет опасность практически только для младенцев. В основном синтезирует белки маленькой и средней длины, «двугорбого» распределения длин белков не наблюдается. На хромосоме гены распределены по цепям случайным образом. Некоторые гены накладываются друг на друга.
5. СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
6. БЛАГОДАРНОСТИ
Автор обзора выражает благодарность преподавателям информатики факультета биоинженерии и биоинформатики за интересный и понятный курс практической информатики.
7. ИСТОЧНИКИ
- Iversen C. et al., « The taxonomy of Enterobacter sakazakii: proposal of a new genus Cronobacter gen. nov. and descriptions of Cronobacter sakazakii comb. nov. Cronobacter sakazakii subsp. sakazakii, comb. nov., Cronobacter sakazakii subsp. malonaticus subsp. nov., Cronobacter turicensis sp. nov., Cronobacter muytjensii sp. nov., Cronobacter dublinensis sp. nov. and Cronobacter genomospecies 1.» BMC Evol Biol 7, 64 (2007), doi:10.1186/1471-2148-7-64
- Catherine J. Hunter et al., «Enterobacter sakazakii: An Emerging Pathogen in Infants and Neonates», Surg Infect (Larchmt) 2008 Oct; 9(5): 533–539. doi: 10.1089/sur.2008.006