Эволюционные домены. Pfam и InterPro.
1. Описание доменной архитектуры белка в соответствии с банком Pfam.
С главной страницы Pfam доступны разные виды поиска. В частности, "JUMP TO" позволяет искать по ID белка. По полученным
данным заполняем таблицу:
Доменная структура белка YSDC_BACSU по данным Pfam
Cхема из Pfam:
 |
Пояснения к схеме
|
| № |
Pfam AC |
Pfam ID |
Полное название семейства доменов |
Положение в последовательности белка XXXX_BACSU |
Клан |
| 1. |
PF05343 |
Peptidase_M42 |
Семейство глутамиловых аминопептидаз М42. Оно включает в себя металлопептидазы, некоторые из которых
также имеют ациламиноацилпептидазную активность... Обычно связывают 2 атома цинка или кобальта.
|
48-342 |
Клан Peptidase_MH (CL0035), содержит 12 семейств, из которых у двух неизвестна функция (PFAM ID начинается с DUF). |
Нашелся также еще какой-то домен (координаты 333-353, на схеме он виден), но т.к. он совсем неизвестный, сказать о нем нечего,
и в таблице его нет.
2. Привожу данные о домене PF05343.
1) Домен входит в 4 разные архитектуры: Peptidase_M42; Peptidase_M42 x 2; Peptidase_M42, M20_dimer; Peptidase_M42, Lactamase_B, Flavodoxin_1.
2)Последовательность известна для 2607 белков, содержащих домен.
3)Пространственная структура определена для 5 разных белков, содержащих домен.
4)Получаем выравнивание "seed" фрагментов белков, соответствующих домену: PF05343_seed.msf
3. Описание архитектуры с двумя или более доменами.
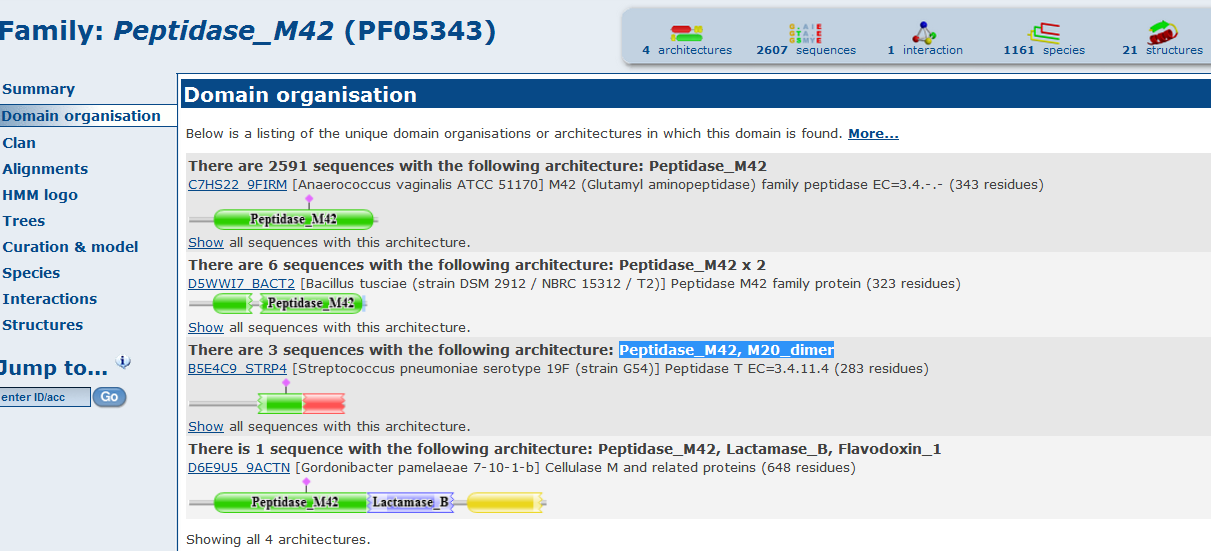
Выбираем архитектуру Peptidase_M42, M20_dimer. Схема: 
Открываем страничку домена. Переходим по ссыке "Species", далее – "Tree". Выбираем "Expand to depth" = 2. Получаем дерево!
Первая цифра возле названия таксона означает кол-во видов, вторая – кол-во последовательностей белков с данным доменом,
третья – общую представленность домена в последовательностях.
Представленность домена PF05343 в организмах разных таксонов
|
Таксон
|
Количество белков с доменом PF05343.
|
| Эукариоты |
Зеленые растения (Viridiplantae) |
- |
| Грибы (Fungi) |
- |
| Животные (Metazoa) |
- |
| Остальные эукариоты (Heterolobosea, Гетеролобозовые) |
1 |
| Археи (Archaea) |
133 |
| Бактерии (Bacteria) |
2466 |
| Вирусы (Viruses) |
- |
| Неклассифицированные (uncultured marine microorganism HF4000_009G21) |
1 |
Исходя из данных, делаем вывод о небольшой распространенности домена. Чаще всего его можно встретить
в последовательностях белков бактерий.
Представленность домена PF07687 в организмах разных таксонов
|
Таксон
|
Количество белков с доменом PF07687.
|
| Эукариоты |
Зеленые растения (Viridiplantae) |
230 |
| Грибы (Fungi) |
631 |
| Животные(Metazoa) |
326 |
| Эвгленовые (Euglenozoa) |
87 |
Остальные эукариоты |
110 |
| Археи (Archaea) |
359 |
| Бактерии (Bacteria) |
15224 |
| Вирусы (Viruses) |
- |
| Неклассифицированные |
31 |
Из-за большого числа разнообразных видов (2890), дерево стало доступно только после скачивания, в текстовом формате.
В отличие от PF05343, этот домен представлен в разнообразных таксонах очень широко. Наиболее часто встречается у бактерий;
довольно много представителей из эукариот (преимущество у грибов).
4. Сравнивание описания мотивов в разных банках семейств, по данным InterPro.
Открываем главную страничку InterPro. По идентификатору UniProt белка ищем описание всех подписей (signatures),
интегрированных в InterPro, т.е. имеющих InterPro ID. Получаем:
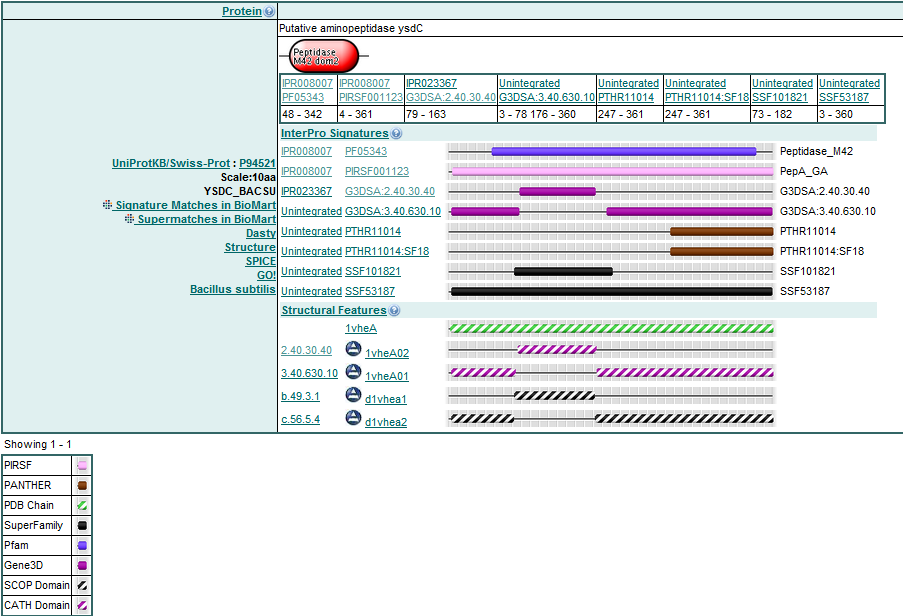
Самый короткий мотив – G3DSA:2.40.30.40, InterPro ID: IPR023367 (домен). Описан в банке Gene3D.
Самый длинный мотив – PepA_GA, InterPro ID: IPR008007 (домен). Описан в банке PIRSF.
Интегрированные структурные подписи: 1vheA, 1vheA01(2.40.30.40), d1vhea1(b.49.3.1), d1vhea2(c.56.5.4).
Границы структурных доменов от границ доменов Pfam довольно сильно отличаются. Первые оказываются шире:
2-361 (48-342 и 333-353 в Pfam), или уже: 77-164, или вообще имеют разрывные границы.



