Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
1. Запустила BLAST по Swiss-Prot,
ограничив выдачу таксоном Bacteria и поставив порог на E-value = 0.001.
Получив и отсортировав результаты, в рабочей директории создала файл myproteins.list:
Далее командой seqret @myproteins.list myproteins.fasta получила файл с последовательностями
белков в fasta-формате.
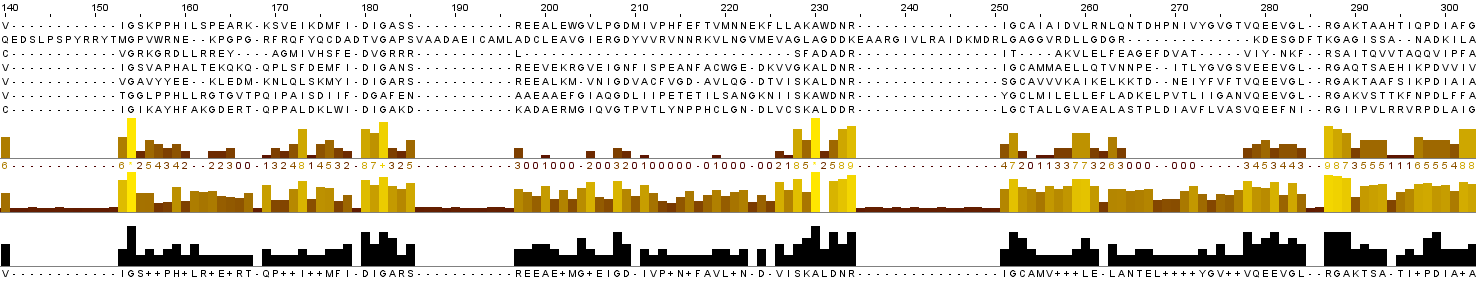
Открыла полученный файл с последовательностями в JalView, с помощью программы TcoffeeWS получила
множественное выравнивание:
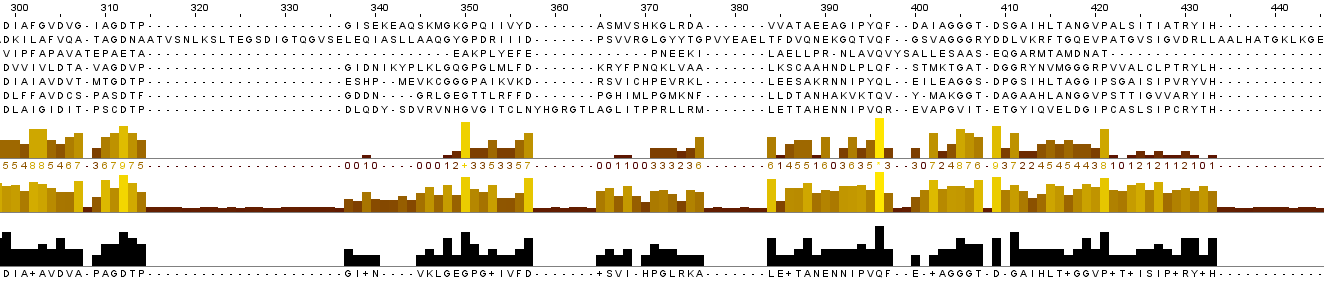
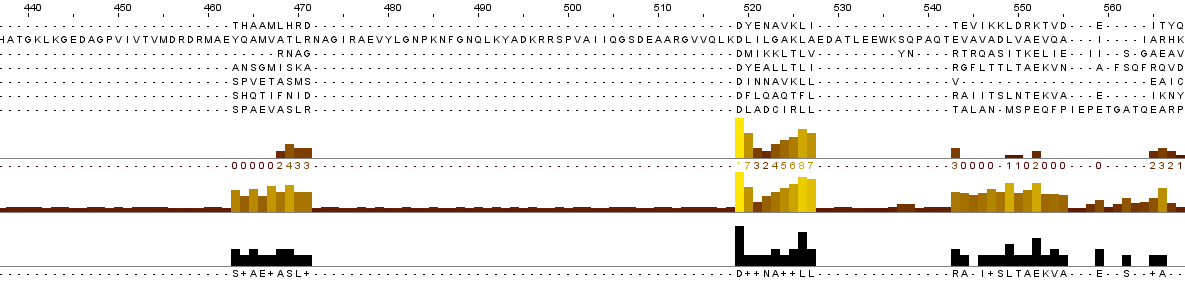
Участки с повышенной долей консервативных позиций, в координатах по столбцам: 59-60,
81-86, 106-107, 228-230, 232-234, 287-289 (по координатам белка YSDC_BACSU: 68-73, 91-92, 178-180, 182-184, 219-221).
Можно выделить также одиночные консервативные столбцы: 1, 28, 30, 53, 59, 98, 117, 154, 180, 299, 312, 396, 519
(в координатах по столбцам выравнивания). Могу предположить, судя по гэпам, что выравнивание не имеет биологического смысла на участках
18-24, 55-58, 127-134, 141-152, 186-196, 236-250, 315-336, 358-364, 434-462, 472-518, 528-542, 556-564 (в координатах по столбцам выравнивания).
Теперь определим группы сходных аминокислот, образующих в выравнивании функционально консервативные позиции.
Используем раскраску BLOSUM62. Подкрашены те остатки, которые совпадают с консенсусом в данной колонке
или имеют положительные вес в BLOSUM62 с ним; интенсивность цвета зависит от веса в матрице. 2. Используя SRS, получим файл с последовательностями малых дельта-антигенов в формате fasta.
sw:YSDC_BACSU
sw:SYH_JANSC
sw:ATPG_RHOCS
sw:FRVX_ECOLI
sw:AMP_CLOTM
sw:PEPA_LACLM
sw:SGCX_SALTY

Получим картинку с выравниванием (минимальная степень консервативности установлена на значении 30).
Вот некоторые группы: IL, VL, DE, RK, MLI. Из них группа DE встречается 3 раза, группа RK – 2 раза.
Строка запроса: ([swissprot-Taxonomy:Deltavirus*] & ([swissprot-Description:delta*] & [swissprot-Description:small*])).
Результатов: 17.
Сохраняем список, получаем файл delta.fasta.
Далее выравниваем несколько последовательностей, находящихся в файле "delta.fasta", программой muscle. Для этого
соединияемся с kodomo через putty, делаем активной рабочую директорию, а затем выполняем команду:
muscle -in delta.fasta -out smdelta_aligned_seq.fasta
Получаем файл с выровненными последовательностями в формате fasta: smdelta_aligned_seq.fasta.
Открываем этот файл в Jalview. Действительно, все уже выровнено, "на глаз" ничего подбирать не нужно.
Делаем раскраску Hydrophobicity. Самые гидрофобные остатки покрашены красным, а самые гидрофильные – синим.
Минимальная степень консервативности установлена на 60, чтобы лучше было видно полностью консервативные столбцы выравнивания.
Оказалось, их достаточно много.
Получим картинку с выравниванием.
Сохраняем выравнивание: delta.msf.