Поисковые системы.
1. Задание: с помощью SRS найти свой белок в банке SwissProt
и описать не менее трех "особенностей" (features) в его аминокислотной последовательности.
Заходим на Library page, отмечаем UniPRotKB/Swiss-Prot.
Выбираем Standard Query Form.
В одном из полей выбераем ID, вносим в окошко идентификатор белка.
Нажимаем Search. Переходим по гиперссылке в левом столбце.
Находим Features:
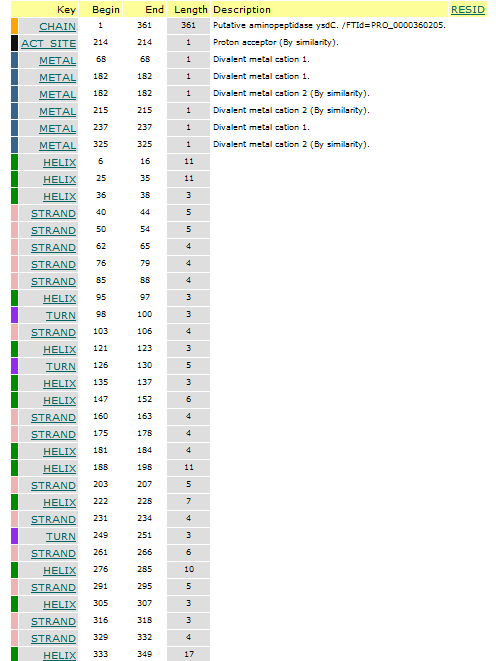
Выберем какие-нибудь три особенности.
Первая. Альфа-спираль длиной в 11 остатков: ETLTMLKDLTD.
ID YSDC_BACSU_9; parent: YSDC_BACSU
FT HELIX 6 16
SQ Sequence 11 AA;
ETLTMLKDLT D
//
Вторая. Бета-поворот, образован тремя остатками: MLA.
ID YSDC_BACSU_18; parent: YSDC_BACSU
FT TURN 98 100
SQ Sequence 3 AA;
MLA
//
Третья. Бета-тяж, образован шестью остатками: QIIVYD.
ID YSDC_BACSU_32; parent: YSDC_BACSU
FT STRAND 261 266
SQ Sequence 6 AA;
QIIVYD
//
2. В задании требуется получить и сохранить описание всех записей банка трехмерных структур PDB,
относящихся к нашему белку.
Возвращаемся на страницу находок; в окошке Apply options to указано,
к каким находкам будут применяться последующие действия.
Переходим по ссылке Link to related information. Выберираем банк PDB. Далее сохраняем информацию по ссылке Save.
В окошке должно быть указано: File(text). Число Number of entries to download должно быть больше
числа сохраняемых записей. Результат сохраняется в нужном нам виде; в данном случае, Save with view
установить на PDBShortView.
Получаем файл с описанием единственной структуры, PDB:1VHE: ysdc_bacsu_pdb.txt.
3. Находим полноразмерные белки из Firmicutes, выполняющие функцию, сходную с функцией ysdc_bacsu.
Для этого, пользуясь SRS, составляем несколько запросов к банку Swissprot.
Строка запроса содержится в верхней части страницы с результатами, в окошке Query.
Имеет смысл вводить слова по одному, соединяя их знаком &.
Составляем запросы и на все последовательности, и на полноразмерные (не фрагменты) только.
Таблица встеречаемости в SwissProt белков из Firmicutes , имеющих функцию, сходную с
функцией белка YSDC_BACSU из Bacillus subtilis:
| Формулировка функции белка | Строка запроса | Количество найденных документов
|
|---|
| предполагаемая аминопептидаза | ([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:putative*] & [swissprot-Comment:aminopeptidase*]) > parent )) | 1
|
| аминопептидаза | ([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:putative*] & [swissprot-Comment:aminopeptidase*]) > parent )) | 16
|
| :( предполагаемая аминопептидаза, только полноразмерные белки | (([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:putative*] & [swissprot-Comment:aminopeptidase*]) > parent )) & ([swissprot-Description:*] ! [swissprot-Description:fragment*])) | 1
|
| аминопептидаза... только полноразмерные | (([swissprot-Taxonomy:Firmicutes*] & ([swissprot-Comment:aminopeptidase*] > parent )) & ([swissprot-Description:*] ! [swissprot-Description:fragment*])) | 16
|
4. Требуется сохранить полноразмерные последовательности найденных белков в fasta-формате.
Для этого возвращаемся к наиболее подходящему запросу из задания 3: переходим по ссылке Results из верхнего меню,
отмечаем галочкой нужный запрос из списка всех запросов и Return query. Выберем второй из таблицы.
Отмечаем галочками 10 находок. Теперь сохраняем полноразмерные последовательности найденных белков в fasta-формате.
Получаем файл ysdc_bacsu_all_seq.fasta.
5. Нужно сохранить список последовательностей из задания 4,
в котором указаны ID, AC, организм, название белка и длина последовательности.
Для этого возвращаемся к стандартной форме запроса, вводим тот же запрос.
Ниже запроса в разделе Create view отмечаем поля ID, AccessionNumber, Description,
Species, Sequence Length. Ищем.
Далее отмечаем те последовательности, которые были сохранены в fasta-формате. Сохраняем.
Получаем плоскую таблицу ysdc_bacsu_fields.xls
