Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
Задание №0
Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков определены параметры, с помощью базы данных OPM.
| Таблица 1. Описание трансмембранных белков с известной 3D структурой | ||||||||||||||||||||||||||||||||||
| PDB код | Тип
(спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 2zzl | спираль | Мембрана архебактерий | 29.8 ± 0.6 A | 21 |
| 1xio | спираль | Грам-отрицательная внутренняя мембрана Anabaena sp. | 31.9 ± 1.5 A | 22 |
| 1h68 | спираль | Мембрана архебактерий | 30.3 ± 1.5 A | 19 |
| 1bxw | баррель | Грам-отрицательная внешняя мембрана E. coli | 25.4 ± 1.9 A | 9 |
| 2k0l | баррель | Грам-отрицательная внешняя мембрана K. pneumoniae | 24.3 ± 2.3 A | 9 |
| 3qra | баррель | Грам-отрицательная внешняя мембрана Yersinia pestis | 25.2 ± 1.4 A | 8 |
На основе полученных данных можно сделать некоторые выводы. Наружная мембрана грамотрицательных бактерий тоньше внутренней; одна трансмембранная альфа-спираль содержит, как правило, больше а.о., чем один бета-тяж.
Задание №1: отбор гомологов
Данный мне белок (его цепь А) оказался подозрительным, и гомологи у него искались плохо, поэтому я взяла резервный белок 3RKO (цепь М). С помощью PSI-BLAST проведен поиск гомологов. После 5 итераций с порогом e-value 1e-10 отобрано 10 гомолгов из разных таксонов. Названия последовательностей отредактированы, получен fasta-файл.
Задание №2: анализ структуры выданного белка
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей к нормали | Количество трансмембранных спиралей | Название белка |
| 3RKO | E. coli | Внутренняя мембрана (грамотрицательная) | 3.D.1.7.1 | 10° | 19 | H+/Na+-переносящая NADH дегидрогеназа |
3.* – первичные активные транспортёры
3.D.* – транспортёры, работающие за счёт окислительно-восстановительных реакций
3.D.1.* - NADH дегидрогеназы, осуществляющие транслокацию H+ или Nа+
3.D.1.7.* - оксидоредуктазы убихинона
Схема реакции:
NADH + ubiquinone + 4H+ (in) > NAD+ + ubiquinol + 4H+ (out)
Задание №3: анализ множественного выравнивания трансмембранных белков
Было построено множественное выравнивание с помощью Muscle и импортировано в Jalview. К выравниванию добавлена дополнительная аннотация положения трансмембранных спиралей. Установив связь между последовательностью и структурой мего белка, пометила участки выравнивания, отвечающие трансмембранным спиралям в строке-аннотации TM_REAL буквой "М". Добавила затем предсказание трансмембранных спиралей, выдаваемых программой TMHMM, для гомолога NU5M_HUMAN. Эти участки помечены в строке TM_PREDICTED буквой "P". Получила изображение структуры:
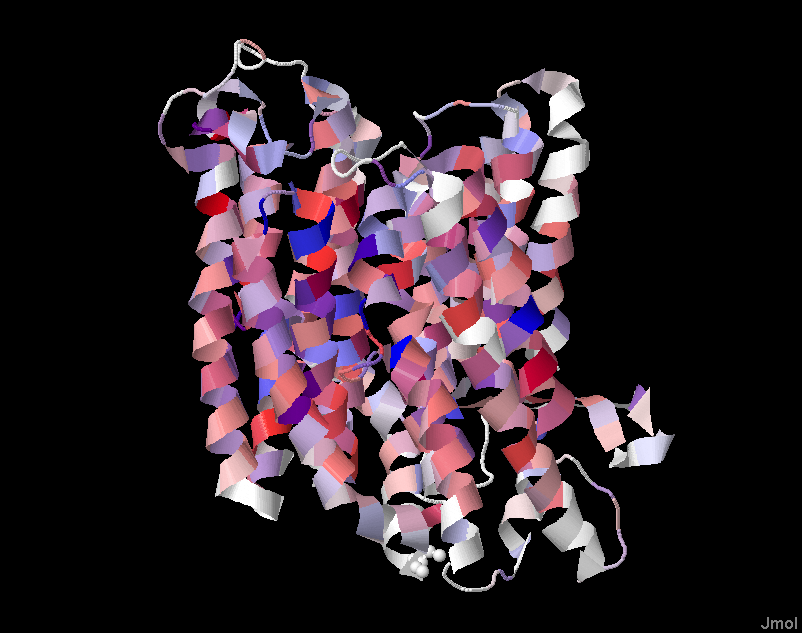
Применена окраска Hydrophobicity, c порогом консервативности 20%. Самые гидрофобные остатки - красные, а самые гидрофильные - синие. Цвета на структуре белка отвечают выбранной цветовой схеме. Часть белка, ориентированная в n-сторону мембраны, расположена сверху, а ориентированная в p-сторону - снизу. Попробуем проанализировать полученное. Действительно, можно заметить и по изображению, что консервативность на участках трансмембранных спиралей в целом выше. В трансмембранных спиралях встречаются высококонсервативные заряженные и полярные остатки, отвечающие, возможно, за функциональную роль белка. Между спиралями встерчаются как консервативные, так и неконсервативные участки. Спиралей в этой структуре много, и довольно сложно увидеть какую-либо закономерности между зарядом остатков и их расположением относительно мембраны.
Результаты TMHMM довольно плохо совпадают с реальной структурой, кое-где они все же перекрываются с небольшим смещением. Предполагаю, что возможно, программа работает хорошо, но у меня получилась не самая удачная выборка гомологов (следственно, плохое выравнивание), или структура NU5M_HUMAN отличается от структуры моего белка. Есть и совпадения (с небольшими отклонениями) результатов программы с реальной структурой.
