Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
1. Пользуясь таксономическим сервисом NCBI, определяем, к каким таксонам относятся отобранные бактерии. Находим на нашем дереве ветви, выделяющие какие-нибудь из таксонов.
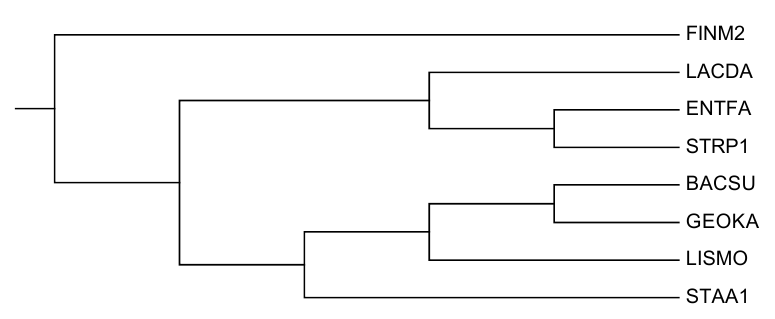
Семейство Bacillaceae выделяется ветвью {BACSU, GEOKA} vs {FINM2, LACDA, ENTFA, STRP1, LISMO, STAA1}.
Порядок Bacillales выделяется ветвью {BACSU, GEOKA, LISMO, STAA1} vs {FINM2, LACDA, ENTFA, STRP1}.
Порядок Lactobacillales выделяется ветвью {LACDA, ENTFA, STRP1} vs {FINM2, BACSU, GEOKA, LISMO, STAA1}.
Класс Bacilli выделяется ветвью {FINM2} vs {LACDA, ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1}.
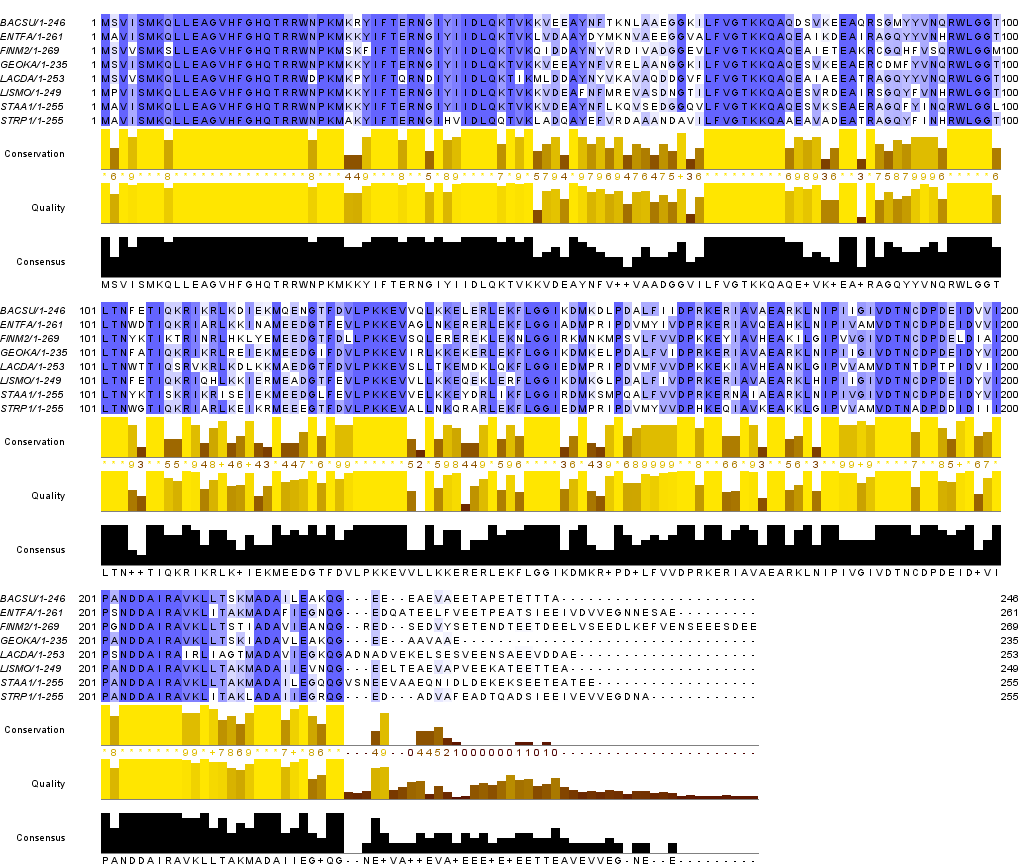
2. Дерево буду реконструировать по белкам семейства S2.
Командами seqret sw:****_**** получаем последовательности белков, помещаем их в один файл, редактируем названия.
В Jalview получаем выравнивание с помощью алгоритма Muscle, раскраска по идентичности:
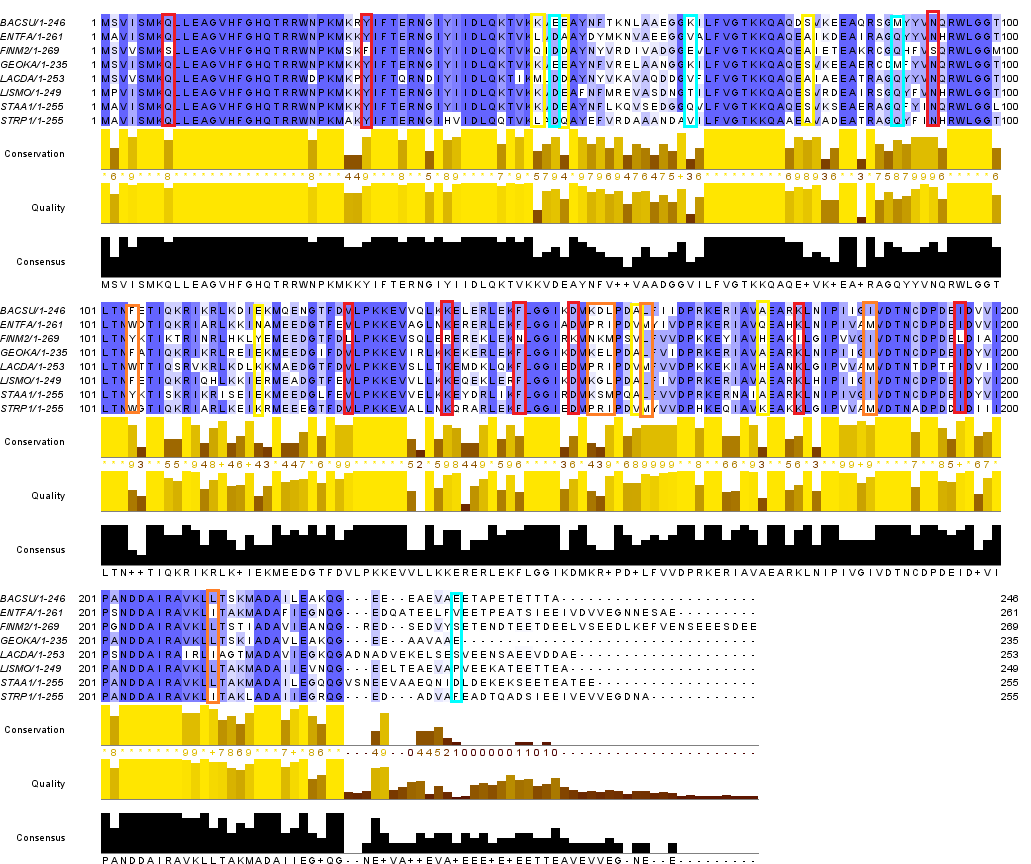
3. Поищем диагностические позиции выравнивания (т.е. те, по которым можно судить о том, относится ли та или иная последовательность выравнивания к некоторому таксону). Красным выделены позиции, диагностические для класса Bacilli; желтым - для порядка Bacillales; оранжевым - для порядка Lactobacillales; голубым - для семейства Bacillaceae.
4. Реконструируем филогенетическое дерево четырьмя методами, доступными из JalView.
1) Average Distance Using % Identity
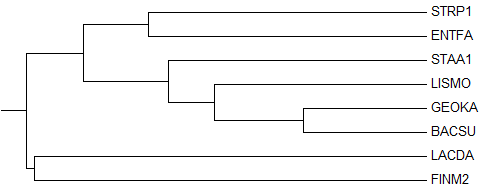
Ветви, отсутствующие в правильном дереве: {LACDA, FINM2} vs {ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1}
Ветви, отсутствующие в новом дереве: {LACDA, ENTFA, STRP1} vs {FINM2, BACSU, GEOKA, LISMO, STAA1}.
2) Neighbour Joining Using % Identity
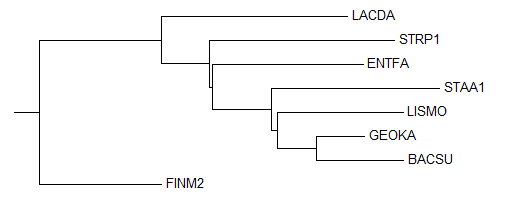
Ветви, отсутствующие в правильном дереве: {LACDA, FINM2} vs {ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1};
{FINM2, LACDA, STRP1} vs {ENTFA, BACSU, GEOKA, LISMO, STAA1}.
Ветви, отсутствующие в новом дереве: {LACDA, ENTFA, STRP1} vs {FINM2, BACSU, GEOKA, LISMO, STAA1};
{ENTFA, STRP1} vs {FINM2, LACDA, BACSU, GEOKA, LISMO, STAA1}.
3) Average Distance Using BLOSUM62
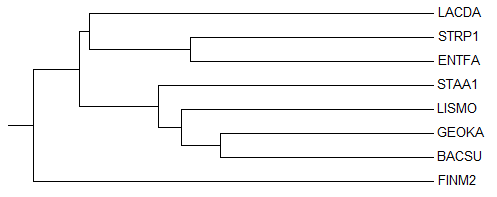
Топология этого дерева идентична топологии правильного.
4) Neighbour Joining Using BLOSUM62
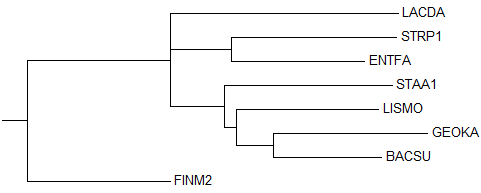
Топология также совпадает с правильным.
5. Импортируем выравнивание в программу Mega. Реконструируируем дерево методом "Maximum Parsimony", укореняем:
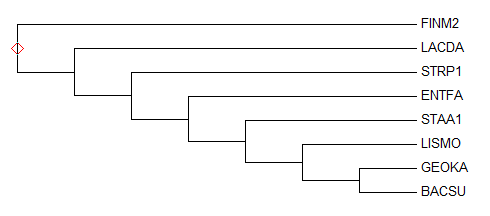
В топологии есть отличия. В данном дереве нет ветвей {ENTFA, STRP1} vs {FINM2, LACDA, BACSU, GEOKA, LISMO, STAA1};
{LACDA, ENTFA, STRP1} vs {FINM2, BACSU, GEOKA, LISMO, STAA1} и появились ветви
{LACDA, FINM2} vs {ENTFA, STRP1, BACSU, GEOKA, LISMO, STAA1};
{FINM2, LACDA, STRP1} vs {ENTFA, BACSU, GEOKA, LISMO, STAA1}.
По топологии это дерево совпадает со вторым деревом, построенным Jalview.