Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
1. Возможности Sculpting
При переходе в меню Wizard->Demo->Sculpting открывается структура в стержневой модели, с полупрозрачными сферами, обозначающими радиусы атомов. Ctrl+LeftClick позволяет деформировать структуру, "тянуть" за выбранные атомы или перемещать их, незвисимо от остальной части молекулы (переключение Toggle Sculpting). Ctrl+RightClick позволяет вращать молекулу вокруг выбранной связи.
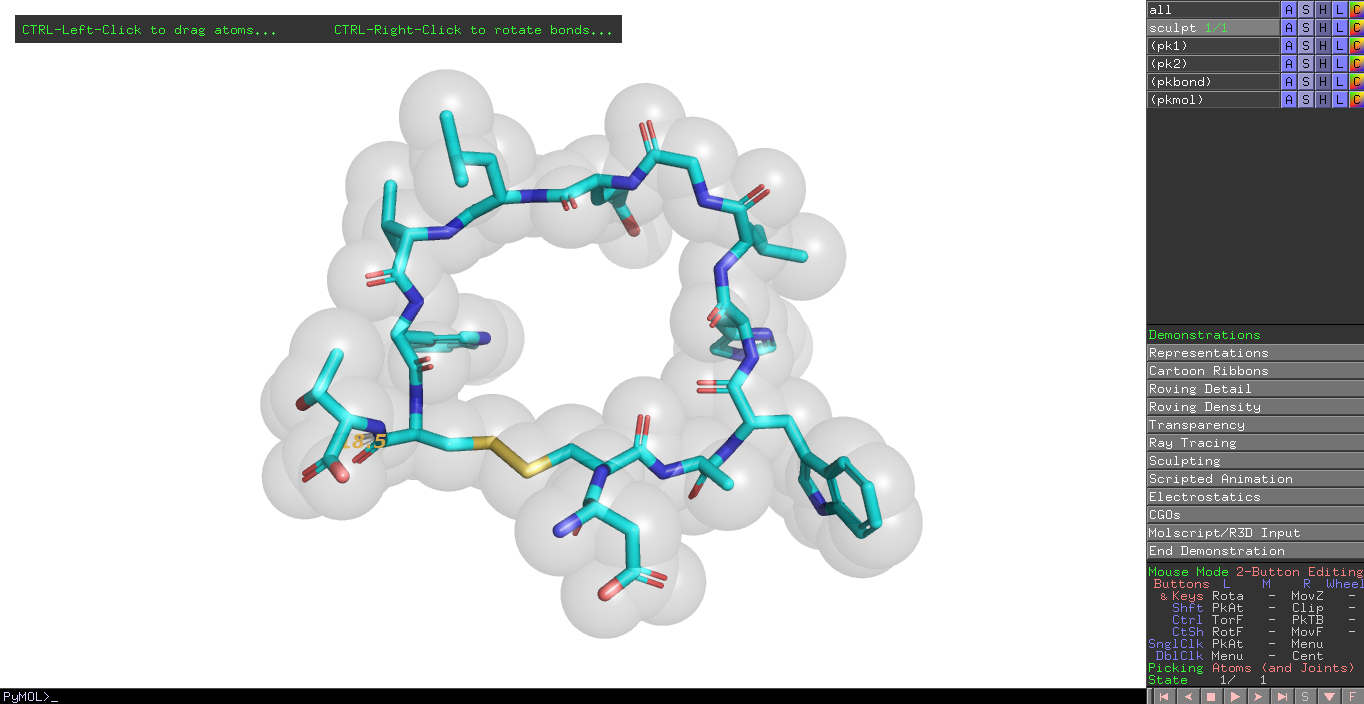
Рис. 1 Окно PyMol, в процессе Sculpting.
Можно проделать такие манипуляции и с нашей структурой 1LMP. Напрмер, попробуем переместить лиганд (Рис. 2).
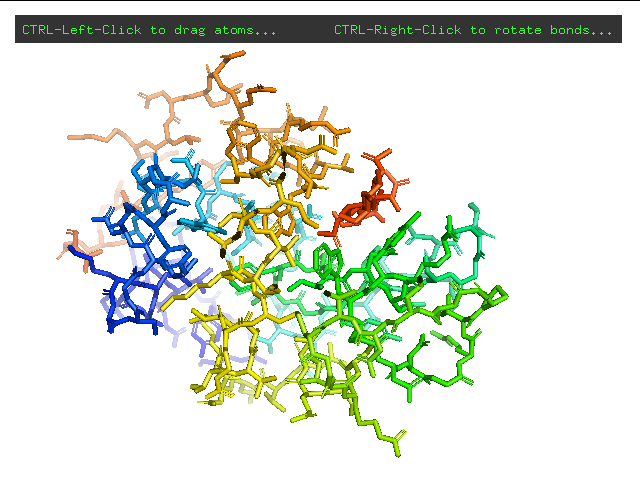 | 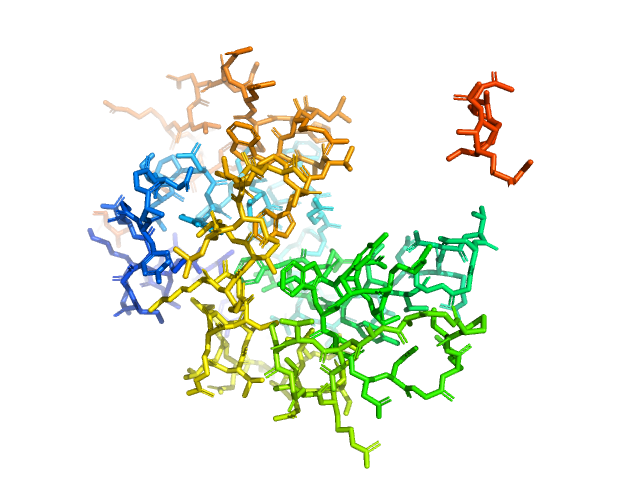 |
Рис. 2 1LMP до и после редактирования.
2. Мутация в белке
С помощью byres сначала определяем остатки, взаимодействующие с лигандом.
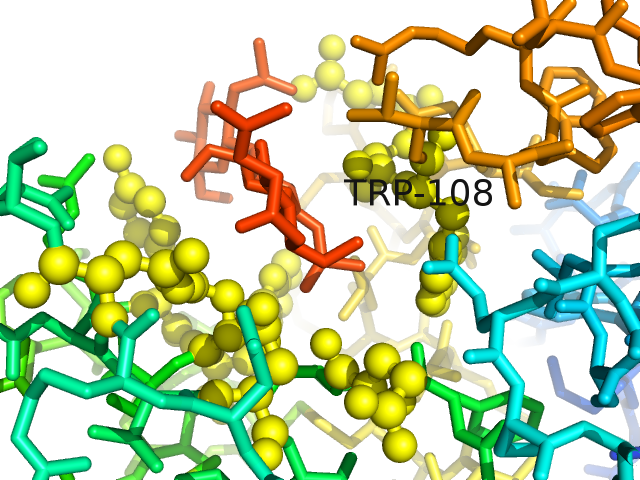 Рис. 3 Область контакта белка с лигандом. Атомы взаимодействующих остатков показаны в виде желтых шаров.
Рис. 3 Область контакта белка с лигандом. Атомы взаимодействующих остатков показаны в виде желтых шаров.
Теперь опробуем заменить Trp108 на непохожую аминокислоту, например, Ala; ожидаем, что связь с лигандом нарушится. Для этого в меню нужно перейти: Wizard->Mutagenesis... На самом деле, при проверке оказалось, что этот остаток с лигандом взаимодействовать больше не может.
3. Создание ролика
Создаем анимационный ролик, где происходит совмещение белков и показывается место мутации. Исходный остаток Trp108 выделен желтым цветом, полученный в результате мутации Ala выделен синим цветом, лиганд - красным.
Скрипт: script.pml
4. Присоединение метки TAMRA к белку
Присоединяем флуоресцентную метку TAMRA к белку через сложноэфирную связь, к кислороду Asp86. Используем fuse для сшивания молекул, а многократный torsion 15 - для оптимизации взаимного расположения.
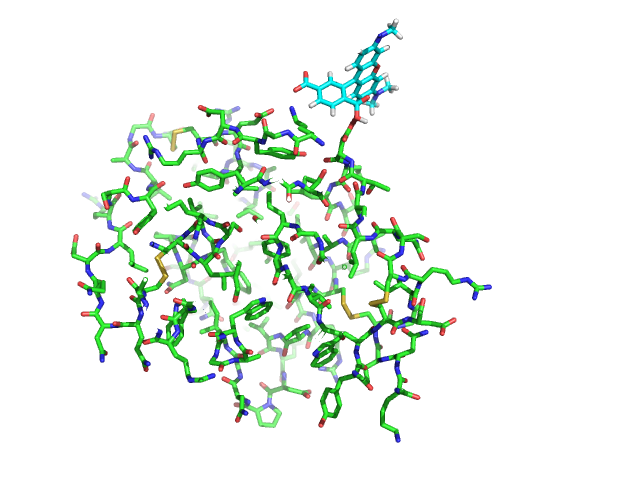 Рис. 4 1LMP с присоединенной через Asp86 меткой TAMRA.
Рис. 4 1LMP с присоединенной через Asp86 меткой TAMRA.
Полученный pdb-файл: fuse.pdb
5. Построение поли-аланиновой альфа-спирали
Альфа-спираль из 100 аланинов (рис. 5) построена с помощью скрипта: coil.pml.
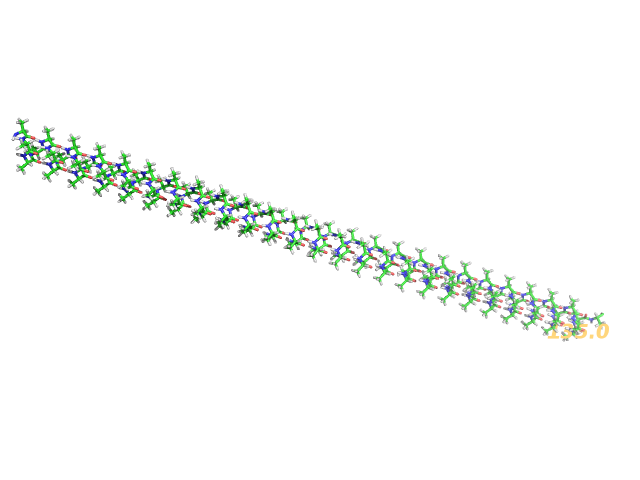 Рис. 5 Поли-аланиновая спираль
Рис. 5 Поли-аланиновая спираль
6. Построение третичной структуры вещества на основе SMILES
Предлагается построить структуру кубана.
SMILES: C12C3C4C1C5C2C3C45 кладем в файл 1.smi.
Командой obgen my.smi > my.mol получаем файл со структурой, откроем в PyMOL (рис.6).
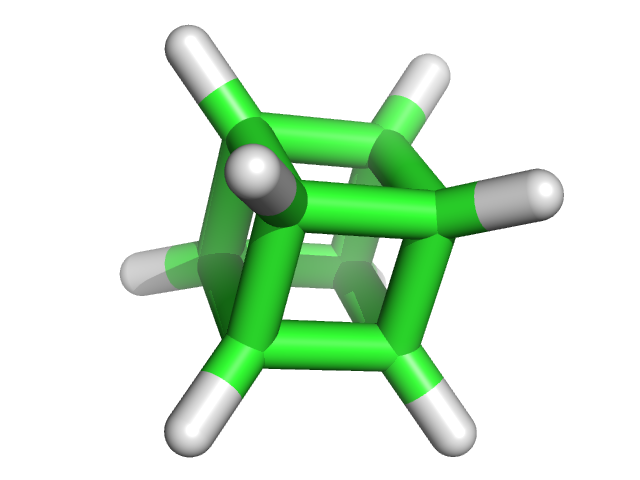
Рис. 6 Структура кубана.