Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
Цель задания - изучение реализации контроля температуры в молекулярной динамике на примере GROMACS. Объект исследования - одна молекула этана.
1. Начнем с того, что подготовим файл координат и файл топологии. Дан gro файл с 38 молекулами этана. Создадим индекс файл, в котором будет группа из одной молекулы этана:
make_ndx -f box_38.gro -o 1.ndx
После запуска команды появляется приглашение к вводу. Выбираем остаток номер 1. Нажимаем enter и видим новую группу. Теперь создадим gro файл с одной молекулой и зададим ячейку. При запуске ediconf выбираем номер соответствующей группе из одной молекулы.
editconf -f box_38.gro -o et1.gro -n 1.ndx
editconf -f et1.gro -o et.gro -d 2 -c
Построим файл топологии для этана et.top. Правильность проверяем с помощью команды:
grompp -f be.mdp -c et.gro -p et.top -o et_test.tpr
После долого подбора подходящих типов атомов, проверка продолжала выдавать ошибки. В итоге ошибки остались для графы [dihedrals], но после замены в ней последней колонки что-то было по-прежнему не так. Проблема оказалась в недостающей связи, после ее добавления ошибки прекратились.
2. Даны 5 файлов с разными параметрами контроля температуры:
be.mdp - метод Берендсена для контроля температуры
vr.mdp - метод "Velocity rescale" для контроля температуры
nh.mdp - метод Нуза-Хувера для контроля температуры
an.mdp - метод Андерсена для контроля температуры
sd.mdp - метод стохастической молекулярной динамики
3. Сначала строим входные файлы для молекулярно-динамического движка mdrun с помощью grompp:
grompp -f ${i}.mdp -c et.gro -p et.top -o et_${i}.tpr
# где i: be,vr,nh,an,sd см. выше список mdp файлов
4. Получилось 5 tpr файлов:
et_be.tpr
et_vr.tpr
et_nh.tpr
et_an.tpr
et_sd.tpr
Теперь для каждого из них запустим mdrun:
mdrun -deffnm et_${i} -v -nt 1
5. Для каждой из 5 систем проводим конвертацию в pdb:
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdb
Сравним полученные модели визуально. Можно пролистывать все состояния каждой модели и сравнивать разные методы.
et_be.pdb
et_vr.pdb
et_nh.pdb
et_an.pdb
et_sd.pdb
Начальные состояния во всех случаях одинаковы. Затем методы начинают демонстрировать разные результаты.
Метод Берендсена выдает вращение молекулы в пространстве, изменения длин связей и углов. При методе "Velocity rescale" также меняются
параметры молекулы, вращение усиливается. Метод Нуза-Хувера показал только вращение вокруг С-С связи, сама молекула практически не сдвигается с места.
Метод Андерсена демонстрирует лишь небольшие колебания параметров и легкий сдвиг молекулы,
а при методе стохастической молекулярной динамики молекула ведет себя совершенно хаотично.
6. Сравним потенциальную энергию связи и кинетическую энергию для каждой из 5 систем.
g_energy -f et_${i}.edr -o et_${i}_en.xvg
Полуены файлы:
et_be_en.xvg
et_be_en.xvg
et_be_en.xvg
et_be_en.xvg
et_be_en.xvg
Построим графики изменения потенциальной и кинетической энергий во времени. Границы оси значений в каждом случае выбирались исходя из исходных данных.
plot "et_be_en.xvg" using 1:2 title "Potential energy (kJ/mol)" pointtype 7 pointsize 1,
"et_nh_en.xvg" using 1:3 title "Kinetic energy (kJ/mol)" pointtype 8 pointsize 1
...
Метод Берендсена:
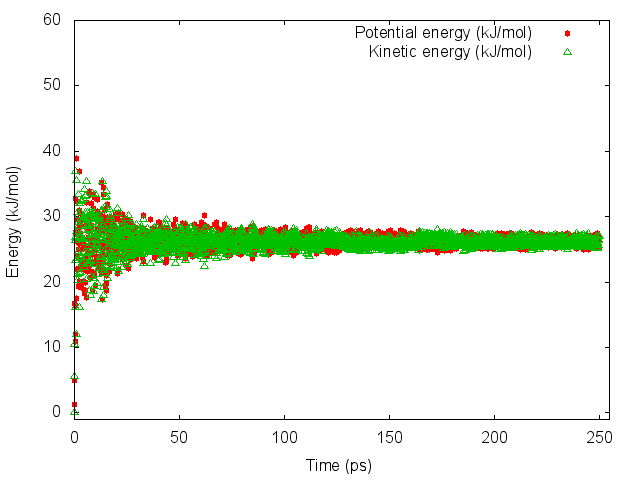
Метод "Velocity rescale":
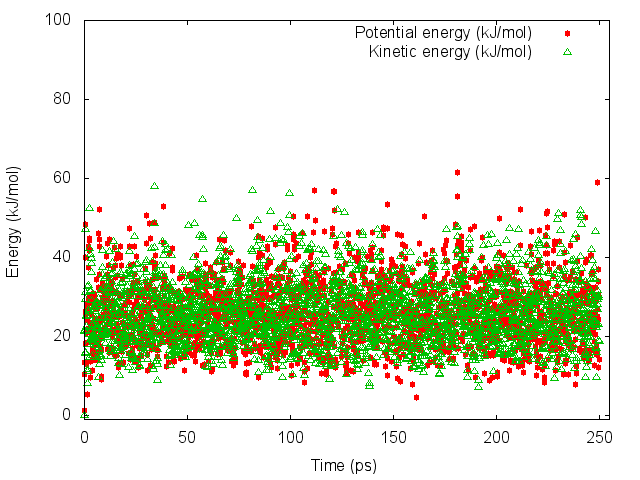
Метод Нуза-Хувера:
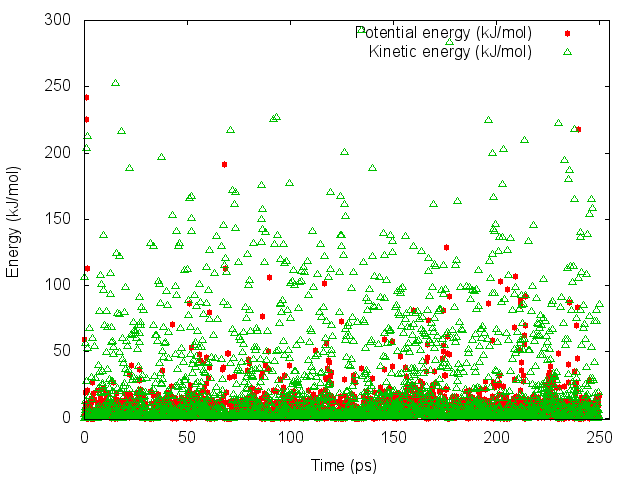
Метод Андерсена:
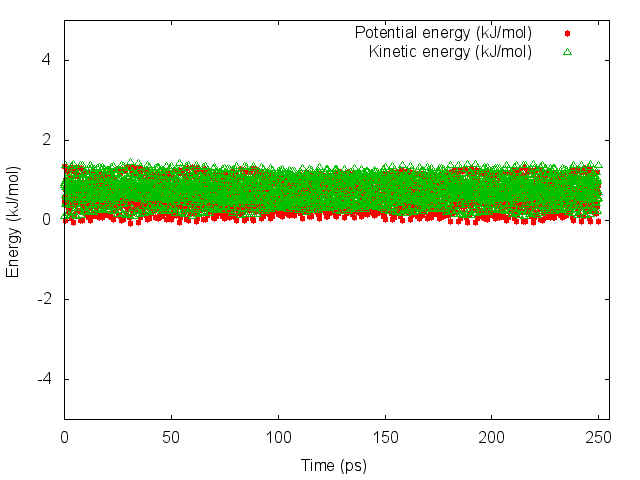
Метод стохастической молекулярной динамики:
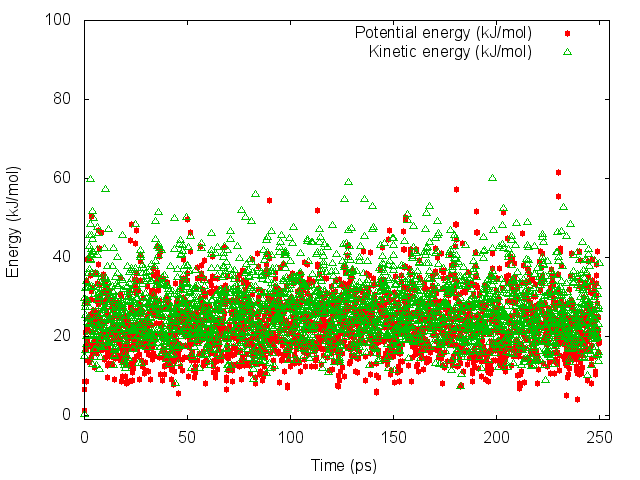
Значения и степени изменения энергии для разных методов сильно различаются. Методы Берендсена и Андерсена показали слишком слабые изменения энергий, что кажется неправдоподобным. При методе Нуза-Хувера большинство значений сконцентрировано у нуля, что также подозрительно.
7. Рассмотрим распределение длинны связи С-С за время моделирования. Сначала создадим индекс файл с одной связью. В текстовом редакторе создаем файл b.ndx и запускаем утилиту по анализу связей g_bond:
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
Получены файлы:
bond_be.xvg
bond_vr.xvg
bond_nh.xvg
bond_an.xvg
bond_sd.xvg
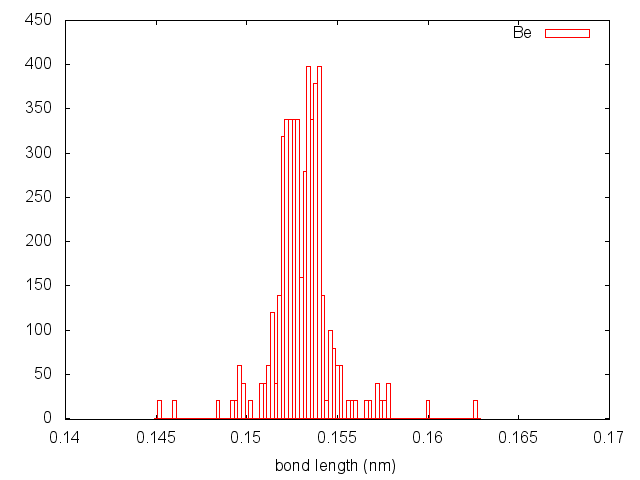
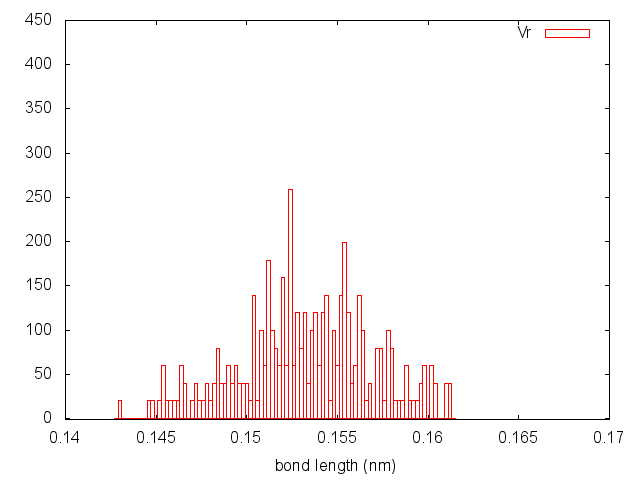
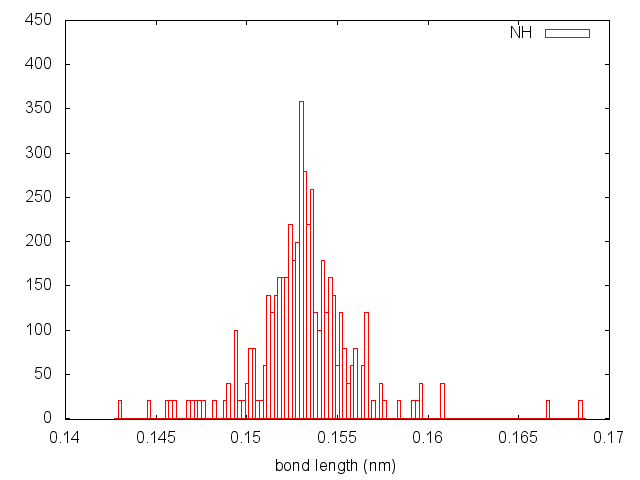
Построим графики распределения длин связей.
plot "bond_be.xvg" using 1:2 title "Be" w boxes
...
Метод Берендсена:
Метод "Velocity rescale":
Метод Нуза-Хувера:
Метод Андерсена:
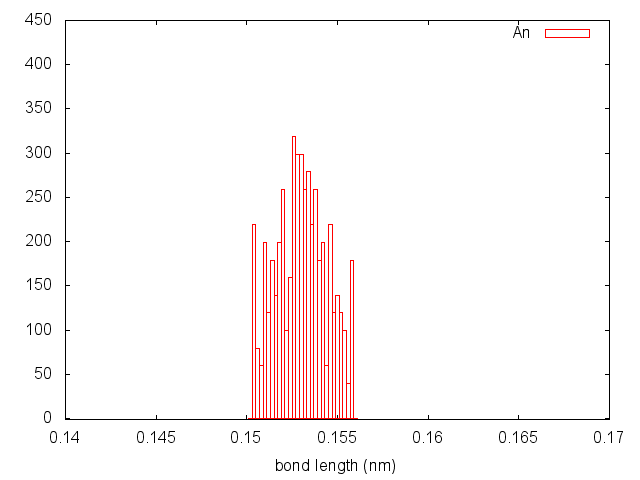
Метод стохастической молекулярной динамики:
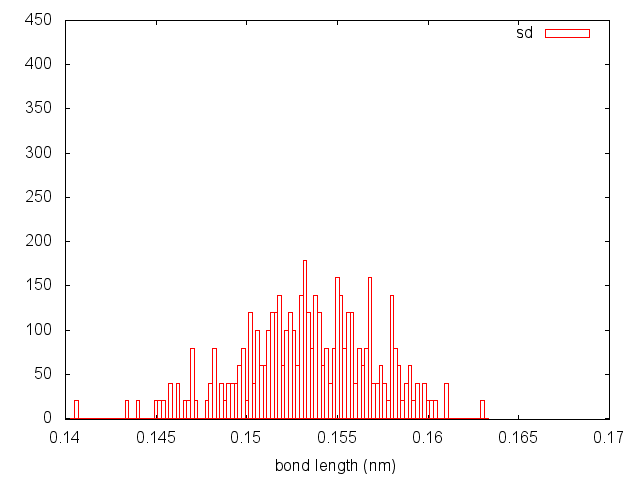
Распределения получились разными. Слишком высокие пики демонстрируют методы Берендсена, Нуза-Хувера и Андерсена.
8. Распределения у "Velocity rescale" и СМД больше напоминают распределения Больцмана. Учитывая результаты всего проведенного анализа, эти два метода, пожалуй, являются наиболее оптимальными для поддержания температуры в системе. А принимая во внимание итоги визуального анализа моделей, метод "Velocity rescale" будем считать наилучшим.