Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
Цель задания - ознакомится с возможностями гомологичного моделирования комплекса белка с лигандом.
Будем работать с белком LYS_BPPA2 бактериофага PA-2, с помощью программы Modeller. В качестве входных данных нужны: управляющий скрипт, файл pdb со структурой-образцом, файл выравнивания с дополнительной информацией. В качестве образца используем известную структуру лизоцима форели.
Построим выравнивание последовательностей из структуры 1lmp и LYS_BPPA2 с помощью ClustalW в формате PIR и модифицируем, получаем файл al.pir.
Модифицируем файл со структурой: удаляем воду и модифицируем имена атомов каждого остатка лиганда, добавляя в конец буквы A, B, C,
чтобы атомы остатка 130 имели индекс А, атомы остатка 131 имели индекс В и т.д.
После модификации имен атомов изменяем номера всех этих остатков на 130.
Получаем файл 1lmp_now.ent.
Создаем управляющий скрипт lys_bppa2.py. Для этого нужно было оперелить, какие водородные связи между белком и лигандом обязательно должны быть в будущей модели. Определим их для образца в PyMol (Рис.1).
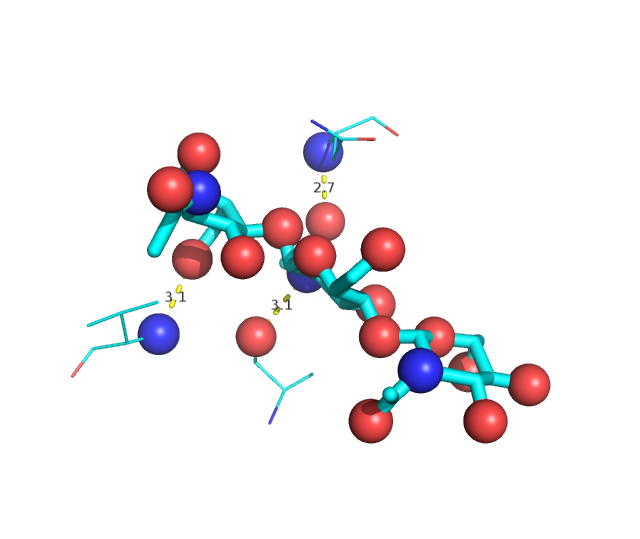
Рис.1 Водородные связи между остатками белка и лигандом в структуре 1LMP.
C помощью выравнивания определяем соответствующие атомы в LYS_BPPA2, номер для лиганда меняем на 166, т.к. в нашем белке 165 остатков.
Также для лиганда изменяем идентификатор цепи на B, т.к. мы оборвали цепь знаком "/."
Запускаем исполнение скрипта командой mod9v7 lys_bppa2.py &. Получаем пять моделей:
B99990001.pdb
B99990002.pdb
B99990003.pdb
B99990004.pdb
B99990005.pdb
Модели получились очень похожими (Рис.2), сходство растет с приближением к зоне контакта с лигандом. У всех моделей есть неструктурированные участки (здесь сходство наименьшее), обусловленные промежутками в выравнивании, но пятая модель получилась наиболее неструктурированной, с отсутствием некоторых спиралей там, где они есть в других моделях. Зато в ней присутствует бета-лист, который в некоторых моделях отсутствует.
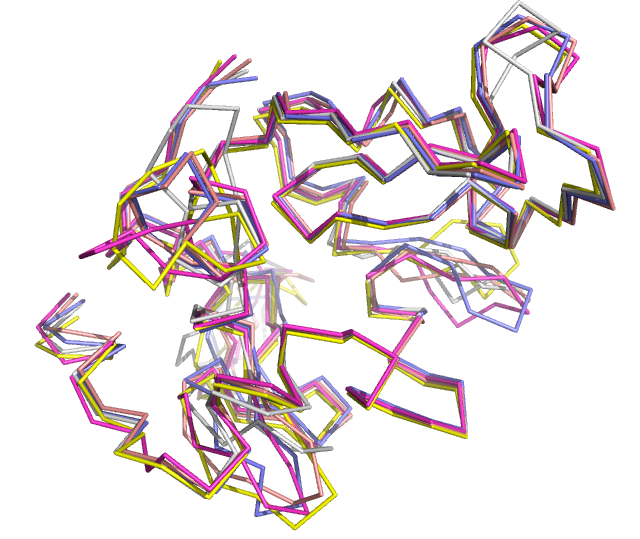
Рис.2 Структуры полученных моделей белка LYS_BPPA2.
Сравним качество моделей в WHATIF, "Protein model check". Ниже представлены некоторые результаты проверки по каждой модели.
B99990001.pdb:
Structure Z-scores, positive is better than average:
Resolution read from PDB file : -1.000
1st generation packing quality : -3.699
2nd generation packing quality : -5.340 (bad)
Ramachandran plot appearance : -1.937
chi-1/chi-2 rotamer normality : -2.765
Backbone conformation : -3.923 (poor)
Inside/Outside distribution : 1.288 (unusual)
B99990002.pdb:
Structure Z-scores, positive is better than average:
Resolution read from PDB file : -1.000
1st generation packing quality : -3.545
2nd generation packing quality : -5.201 (bad)
Ramachandran plot appearance : -1.759
chi-1/chi-2 rotamer normality : -3.448 (poor)
Backbone conformation : -3.089 (poor)
Inside/Outside distribution : 1.253 (unusual)
B99990003.pdb:
Structure Z-scores, positive is better than average:
Resolution read from PDB file : -1.000
1st generation packing quality : -3.033
2nd generation packing quality : -4.503 (bad)
Ramachandran plot appearance : -1.766
chi-1/chi-2 rotamer normality : -3.393 (poor)
Backbone conformation : -3.856 (poor)
Inside/Outside distribution : 1.278 (unusual)
B99990004.pdb:
Structure Z-scores, positive is better than average:
Resolution read from PDB file : -1.000
1st generation packing quality : -3.407
2nd generation packing quality : -5.165 (bad)
Ramachandran plot appearance : -1.862
chi-1/chi-2 rotamer normality : -2.823
Backbone conformation : -3.901 (poor)
Inside/Outside distribution : 1.258 (unusual)
B99990005.pdb:
Structure Z-scores, positive is better than average:
Resolution read from PDB file : -1.000
1st generation packing quality : -3.740
2nd generation packing quality : -5.439 (bad)
Ramachandran plot appearance : -1.620
chi-1/chi-2 rotamer normality : -3.582 (poor)
Backbone conformation : -3.947 (poor)
Inside/Outside distribution : 1.266 (unusual)
Судя по результатам проверки, все модели получились невысокого качества. Пятая модель уступает всем остальным, а четвертая модель в общем демонстрирует лучшие показатели.