Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
Изменение максимальной силы в ходе оптимизации геометрии:
начальное значение максимальной силы = Step 0 Fmax = 4.02438e+03;
конечное значение максимальной силы = Step 86 Fmax = 2.12381e+03.
Силовое поле используемое при построении топологии: amber99sb
Заряд системы: был -10 (ДНК отрицательно заряжена), стал 0 после нейтрализации.
Размер и форма ячейки: кубическая 5.01400 x 5.00700 x 5.26800
Минимизация энергии:
Алогритм минимизации энергии: integrator = l-bfgs; Goldfarb-Shanno (квази-Ньютоновский), метод аппроксимирует Гауссиановскую матрицу из предыдущих конфигураций
Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: cut-off ("двойное обрезание").
Модель, которой описывался растворитель: implicit_solvent = No, то есть ЯВНЫЙ
Утряска растворителя:
Число шагов: nsteps = 10000
Длина шага: delta_t = 0.001 (ps)
Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: pme (суммирование по Эвальду)
Алгоритмы термостата и баростата: температура Berendsen, контроля давления не проиходило
Основной расчёт МД:
Число шагов: 5000000
Длина шага: 0.002 ps
Алгоритм интегратора: md
Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: pme и cutt-off
Алгоритмы термостата и баростата: Berendsen
Любой анализ начинаем с визуального анализа движений молекул. При вопросе о выводе групп выбераем DNA.
trjconv -f dna_md.xtc -s dna_md.tpr -o dna_pbc_1.pdb -skip 20 -pbc mol
Открываем b_pbc_1.pdb в PyMol.
В ролике молекула хаотично двигается.
trjconv -f dna_md.xtc -s dna_md.tpr -o dna_fit_1.pdb -skip 20 -fit rot+trans
Открываем dna_fit_1.pdb в PyMol.
ДНК уже не "скачет", но молекула выходит за пределы ячейки и появляется с другой стороны.
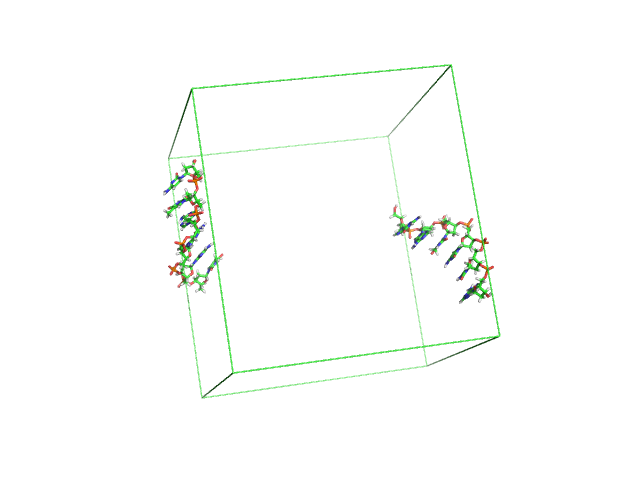
Время моделирования 0,8 нс.
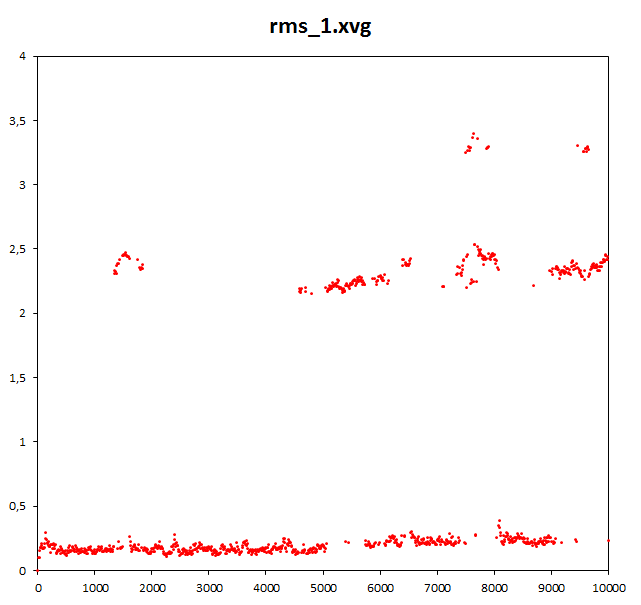
Определим средне-квадратичное отколнение в ходе моделирования. Так как происходит конформационный переход, сначала расчитаем отклонение в ходе всей симуляции относительно стартовой структуры.
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_1
И относительно каждой предыдущей структуры, на растоянии 400 кадров. Если ближе к концу закончился конформационный переход, то отколнение должно уменьшаться.
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_2
-prev 400
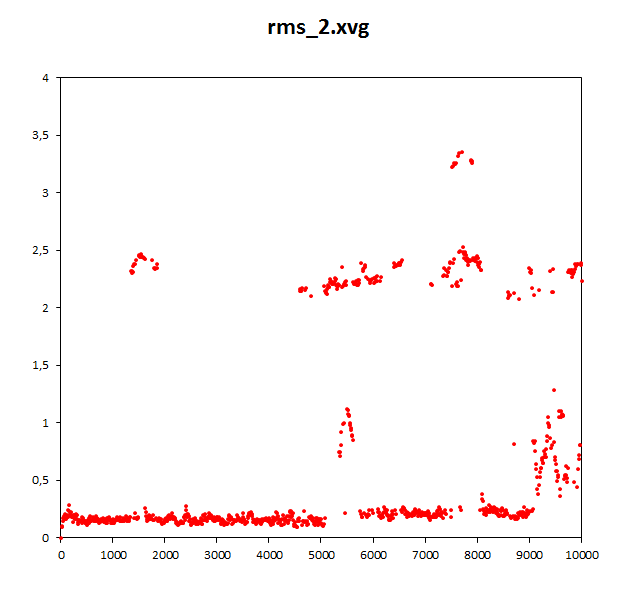
Графики очень похожи, но можно заметить, что к концу временной шкалы во втором случае квадратичное отклонение становится меньше.
Определим изменение гидрофобной и гидрофильной поверхности в ходе конформационного перехода.
g_sas -f dna_md.xtc -s dna_md.tpr -o sas_dna.xvg
В полученном файле второй и третий столбцы - гидрофобная и гидрофильная посверхности соответственно.
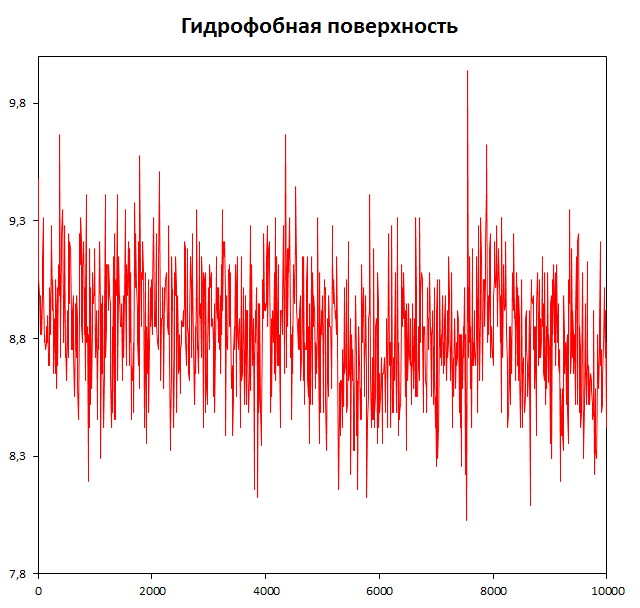
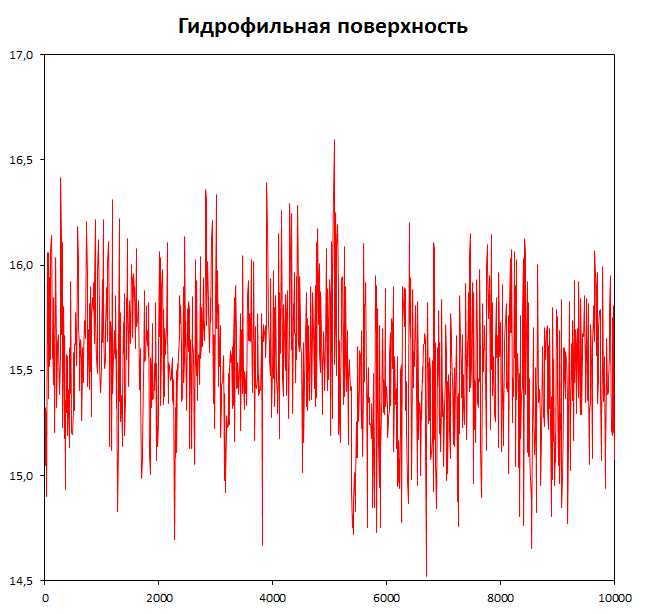
ДНК находится в воде. К концу временного промежутка гидрофобная поверхность незначительно снижается, видимо, это способствует конформационному переходу.
Традиционным анализом для ДНК является расчёт колчества образуемых водородных связей. Если мы будем исследовать связи между ДНК и ДНК, то это будут водородные связи между цепями ДНК. Для конца траектории:
g_hbond -f dna_md.xtc -s dna_md.tpr -num hbond_dna
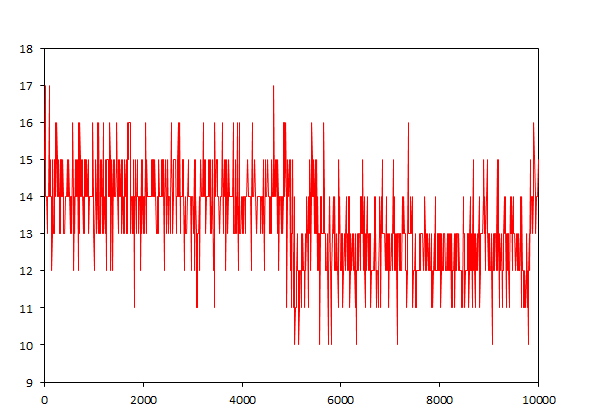
Вообще в каноническом дуплексе между цепями должно быть 14 связей, а тут мы видим значения от 12 до 15, что недалеко от 14. Имеются выбросы вплоть до 10 или 17, но, возможно, это связано с ошибками. Среднее значение колеблется между 13 и 14, и будем считать, что оно мало изменяется во времени.
Интересно будет изучить количество вдородных связей ДНК-Вода.
g_hbond -f dna_md.xtc -s dna_md.tpr -num hbond_dna_sol
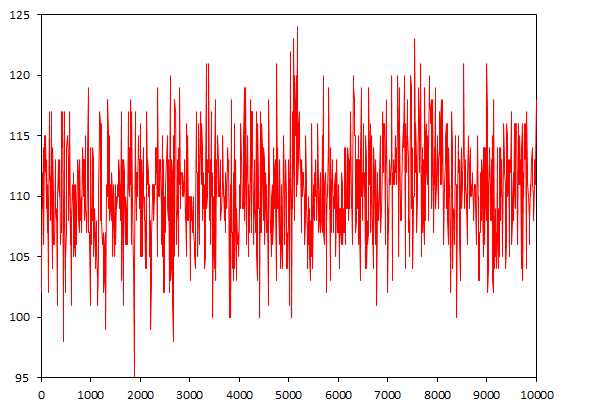
Количество водородных связей варьирует от 95 до 124 и в общем незначительно изменяется с течением времени. Видимо, небольшие изменения в количестве водородных связей не мешают ДНК перейти из одной формы в другую.