Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
1. Поиск в геноме участков, кодирующих белки, похожие на заданный
Предстоит определить, закодированы ли похожие белки в геноме другого организма, не пользуясь аннотацией генома. Создаем в рабочей директории индексные файлы пакета BLAST+ для поиска по заданному геному:
makeblastdb -in lm_genome.fasta -dbtype nucl -out lm
Выбираем программу tblastn и проводим с ее помощью поиск с порогом на E-value 0,001:
tblastn -query ysdc_bacsu.fasta -db lm -out usdc_lm.txt -evalue 0.001
Поиск гомологов белка ysdc_bacsu в геноме L.monocytogenes
| Число находок с E-value < 0,001 | 3 |
| E-value лучшей находки | 2е-159 |
| Название последовательности с лучшей находкой | AL591978 Listeria monocytogenes strain EGD, complete genome, segment 6/12 |
| Координаты лучшей находки (от-до) | 77934-79001 |
| Доля последовательности белка, вошедшая в выравнивание с лучшей находкой | 345/373=0,925 |
2. Нахождение записи EMBL по последовательности программой BLASTN
Рассмотрим первую находку со 100%-ным совпадением, запись CP000100:
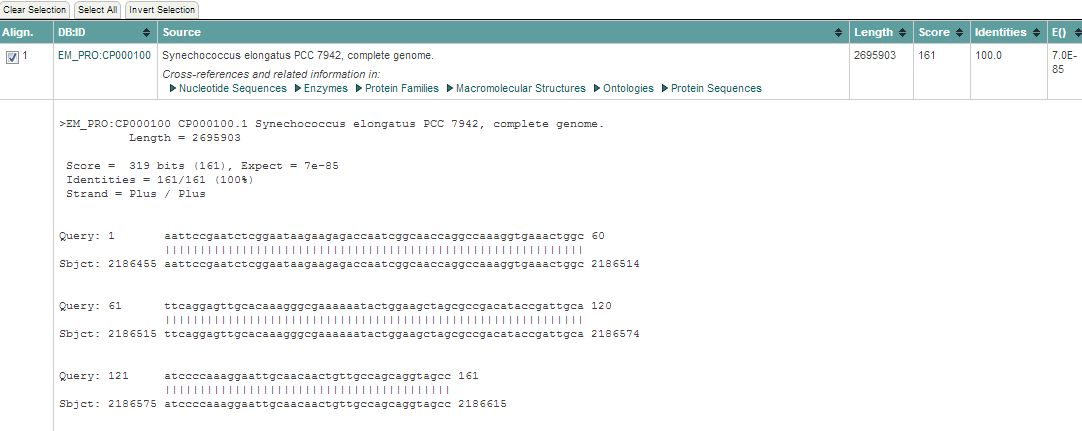
Координаты последовательности в записи: 2186455-2186615, направление последовательности совпадает с направлением записи.
В поле FT записи находим описание участка, включающего данную последовательность (2186064-2186855):
FT CDS complement(2186064..2186855) FT /codon_start=1 FT /transl_table=11 FT /locus_tag="Synpcc7942_2106" FT /product="nitrate transport permease" FT /db_xref="GOA:Q7BW13" FT /db_xref="InterPro:IPR000515" FT /db_xref="UniProtKB/TrEMBL:Q7BW13" FT /inference="non-experimental evidence, no additional FT details recorded" FT /protein_id="ABB58136.1" FT /translation="MVRTPVPLYLRWAVSILSVLAFLAIWQIAAASGFLGKTFPGSLRT FT LQDLFGWLSDPFFDNGPNDLGIGWNLLISLRRVAIGYLLATVVAIPLGIAIGMSALASS FT IFSPFVQLLKPVSPLAWLPIGLFLFRDSELTGVFVILISSLWPTLINTAFGVANVNPDF FT LKVSQSLGASRWRTILKVILPAALPSIIAGMRISMGIAWLVIVAAEMLLGTGIGYFIWN FT EWNNLSLPNIFSAIIIIGIVGILLDQGFRFLENQFSYAGNR"
Его направление - обратное относительно записи.
3. Поиск гомологов гена программой BLASTN
Выбираем одну из записей EMBL (Z75208). Находим координаты CDS:
FT CDS 14623..15708 FT /transl_table=11 FT /gene="ysdC" FT /product="hypothetical protein" FT /note="homology to celA of Clostridium thermocellum and FT pepA of Lactococcus lactis; putative" FT /db_xref="GOA:P94521" FT /db_xref="InterPro:IPR008007" FT /db_xref="InterPro:IPR023367" FT /db_xref="PDB:1VHE" FT /db_xref="UniProtKB/Swiss-Prot:P94521" FT /protein_id="CAA99585.1" FT /translation="MAKLDETLTMLKDLTDAKGIPGNEREVRQVMKSYIEPFADEVTTD FT RLGSLIAKKTGAENGPKIMIAGHLDEVGFMVTQITDKGFIRFQTVGGWWAQVMLAQRVT FT IVTKKGEITGVIGSKPPHILSPEARKKSVEIKDMFIDIGASSREEALEWGVLPGDMIVP FT HFEFTVMNNEKFLLAKAWDNRIGCAIAIDVLRNLQNTDHPNIVYGVGTVQEEVGLRGAK FT TAAHTIQPDIAFGVDVGIAGDTPGISEKEAQSKMGKGPQIIVYDASMVSHKGLRDAVVA FT TAEEAGIPYQFDAIAGGGTDSGAIHLTANGVPALSITIATRYIHTHAAMLHRDDYENAV FT KLITEVIKKLDRKTVDEITYQ"
Воспользуемся программой seqret, получаем файл c нуклеотидной последовательностью, соответствующей белку. Теперь воспользуемся программой blastn:
blastn -query z75208.fasta -db lm -out ysdc_lmn.txt -evalue 0.001 -task blastn
| Число находок с E-value < 0,001 | 2 |
| E-value лучшей находки | 2е-135 |
| Название последовательности с лучшей находкой | AL591978 Listeria monocytogenes strain EGD, complete genome, segment 6/12 |
| Координаты лучшей находки (от-до) | 77937-78964 |
| Доля последовательности белка, вошедшая в выравнивание с лучшей находкой | 1025/1086=0,944 |
Число находок оказалось меньше, значение e-value - больше, длина последовательности - меньше.