Транспортные белки
Построение парного выравнивания исследуемого белка и заданного прототипа
1.Поиск последовательностей белка-прототипа в БД PDB и БД UniProt.а) На сайте банка PDB по ID 1LDF был найден нужный белок. Его последовательность была скачана по ссылке FASTA Sequence.
б) Для поиска белка в БД UniProt я пользовалась SRS. Library Page -> UniProtKB/Swiss-Prot -> Query Form -> AC P0AER0.
2.Их сравнение.
Последовательности белка-прототипа, полученные из разных БД, отличаются по одной (200ой) позиции:
P0AER0 MSQTSTLKGQCIAEFLGTGLLIFFGVGCVAALKVAGASFGQWEISVIWGLGVAMAIYLTA 1LDF MSQTSTLKGQCIAEFLGTGLLIFFGVGCVAALKVAGASFGQWEISVIFGLGVAMAIYLTA P0AER0 GVSGAHLNPAVTIALWLFACFDKRKVIPFIVSQVAGAFCAAALVYGLYYNLFFDFEQTHH 1LDF GVSGAHLNPAVTIALWLFACFDKRKVIPFIVSQVAGAFCAAALVYGLYYNLFFDFEQTHH P0AER0 IVRGSVESVDLAGTFSTYPNPHINFVQAFAVEMVITAILMGLILALTDDGNGVPRGPLAP 1LDF IVRGSVESVDLAGTFSTYPNPHINFVQAFAVEMVITAILMGLILALTDDGNGVPRGPLAP P0AER0 LLIGLLIAVIGASMGPLTGFAMNPARDFGPKVFAWLAGWGNVAFTGGRDIPYFLVPLFGP 1LDF LLIGLLIAVIGASMGPLTGTAMNPARDFGPKVFAWLAGWGNVAFTGGRDIPYFLVPLFGP P0AER0 IVGAIVGAFAYRKLIGRHLPCDICVVEEKETTTPSEQKASL 1LDF IVGAIVGAFAYRKLIGRHLPCDICVVEEKETTTPSEQKASL
3.Выравнивание заданного белка (UniProt) и белка-прототипа (PDB).
Парное выравнивание
строилось программой needle на сервере kodomo.
Характеристики выравнивания:
# Length: 283 # Identity: 228/283 (80.6%) # Similarity: 253/283 (89.4%) # Gaps: 4/283 ( 1.4%) # Score: 1198.5 (при Gap_penalty: 10.0; Extend_penalty: 0.5)
Разметка мембранных сегментов на выравнивании
По ID 1LDF PDB белка-прототипа было найдено описание ориентации белка в мембране в БД OPM (Orientations of Proteins in Membranes database). В соответствии с этим в программе GeneDoc к парному выравниванию заданного белка и прототипа была добавлена еще одна строка-"последовательность", где символами "H" обозначили позиции аминокислот мембранных сегментов, "+" - позиции цитоплазматических петель (и хвостов), "-" - остальные (в периплазме).Предсказание топологии заданного белка с помощью наиболее популярной программы(TMHMM)
-- результат предсказания с сервера
В программе GeneDoc к выравниванию была добавлена
четвертая "последовательность", где на основании предсказания TMHMM
символами "H" обозначили позиции аминокислот мембранных сегментов, "+" -
позиции цитоплазматических петель (и хвостов), "-" - позиции вне клетки.
В результате получили следующие данные:
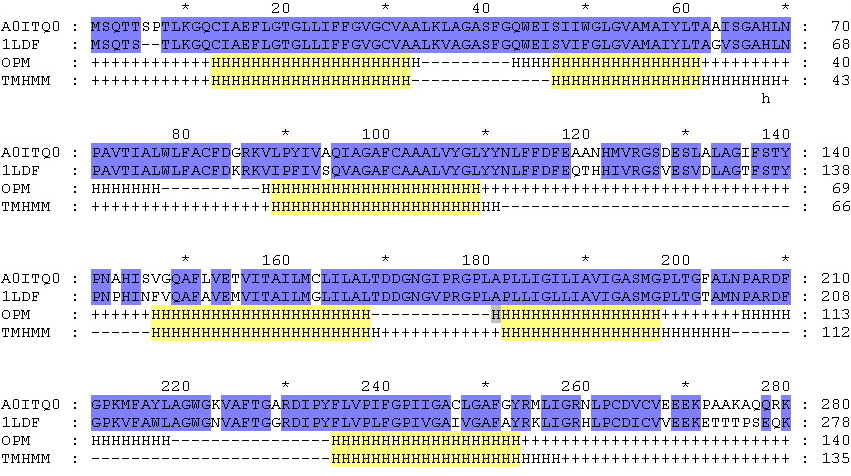
где контрастно раскрашены: синим - выровненные последовательности белков,
желтым - позиции мембранных аминокислот, одинаково предсказанные обоими
описанными выше способами.
Можно посмотреть то же выравнивание в виде текстового файла формата Clustal.
Оценка качества предсказания
Результаты предсказания топологии мембранного белка A0ITQ0| Число а.к. остатков | |
| Всего а.к. остатков | 281 |
| Остатки, предсказанные как локализованные в мембране (всего) | 135 |
| Правильно предсказали (true positives, TP) | 113 |
| Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным ОРМ таковыми не являются, false positives, FP) | 22 |
| Правильно не предсказали ( не предсказаны, и по данным ОРМ не находятся в мембране, true negatives, TN) | 126 |
| Не предсказали то, что нужно (остатки по данным ОРМ находятся в мембране, false negatives, FN) | 27 |
| Чувствительность (sensivity) = TP / (TP+FN) | 0,80 |
| Специфичность (specificity) = TN / (TN+FP) | 0,85 |
| Точность (precision) = TP / (TP+FP) | 0,84 |
| Сверхпредсказание = FP/ (FP+TP) | 0,16 |
| Недопредсказание = FN / (TN+FN) | 0,17 |
В целом по выравниванию видно, что основные мембранные участки белка предсказаны достаточно хорошо. Хоть сервис TMHMM и не обнаружил 2 таких участка, показанных на прототипе в OPM.
Показатели Чувствительность- Специфичность-Точность согласуются между собой и имеют, в принципе, высокие значения. Сверхпредсказание и Недопредсказание, напротив, достаточно низкие.