Поиск гипотетических гомологов белка ECOT_ECOLI в разных БД:
| Поиск по БД Swiss-Prot | Поиск по БД PDB | Поиск по БД "nr" | |
| 1. Лучшая находка (в принципе должна соответствовать заданному белку) | |||
| Идентификатор БД | ECOT_ECOLI | 1ECZ | AAA16410.1 |
| E-value | 4e-92 | 5e-81 | 3e-91 |
| Вес (в битах) | 335 | 295 | 336 |
| % идентичности | 100% | 100% | 100% |
| Найдены ли другие белки с теми же значениями E-value и веса в битах? Если найдены, то укажите общее число и приведите один идентификатор (любой, но желательно Swiss-Prot ID) |
Больше белков с тем же значеним E-value не найдено. | Больше белков с тем же значеним E-value не найдено. | Больше белков с тем же значеним E-value не найдено.Но ближе всего подходит белок:NP_416713.1 |
| 2. Сколько хороших кандидатов в гомологи найдено? (число находок в списке описаний, Descriptions, с E-value < 1E-10) | 44.E-value меняется от 2e-91 ECOT_ECOL6 до 3e-11 ECOTL_SYNPV |
9.E-value меняется от 2e-80 1SLX до 5e-29 1FI8 | 166.E-value меняется от 6е-91 NP_416713.1 до 9e-10 YP_001492593.1 |
| 2. "Худшая" находка (последняя в выдаче с E-value < 1) | |||
| Номер находки в списке описаний (Descriptions) | 45. | 10. | 175. |
| Идентификатор БД | ECOT4_TRYBB | 1FI8 | YP_253379.1 |
| E-value | 7e-07 | 5e-29 | 0.89 |
| Вес (в битах) | 52.8 | 122 | 36.2 |
| % идентичности | 28% | 100% | 34% |
| % сходства | 46% | 100% | 50% |
| Длина выравнивания | 127 | 58 | 69 |
| Координаты выравнивания (номера первых и последних а.о.) | 34-156 | 105-162 | 28-90 |
| % гэпов | 4% | 0% | 8% |
Да,в Swiss-Prot и в "nr" была найдена полная последовательность моего белка,да практически вся найдена в PDB!
Относительно выравниваний можно сказать следующее:идентичность выравнивания белка с самим собой по всем трём базам данных равна 100%!Отличия видны в значениях E-value,которое объясняется тем,что значение этой величины зависит от объёма БД,в которой проводился поиск.В итоге результат с самым низким E-value найден в БД "SwissProt",хотя логичнее было бы предположить,что самый низкий показатель E-value будет обнаружен в банке PDB,как в наименьшем по объёму из данных.
Количество потенциальных гомологов прямолинейно зависит от объёма БД.Таким образом,результат поиска точно отобразил "объёмную характеристику"каждой БД.Действительно,самой крупной БД является "nr",а самой маленькой PDB. Но значение E-value зависит также и от веса выравнивания.А в PDB очень часто имеем не всю последовательность ,а только некоторые фрагменты.Поэтому вес выравнивания,найденного в PDB,меньше,чем соответствующий параметр из банка Swiss-Prot. В данном случае:в банке PDB представлена последовательность длиной в 142 а.о.,а в Swiss-Prote - 162 а.о.,поэтому и вес соответствующего выравнивания,найденного в PDB меньше. .
Результат поиска "худшей находки" заставил меня удивиться!:) Дело в том,что у "худшей находки",найденной в PDB E-value равно 5e-29!И идентичность выравниваний соответственно равна 100%!И процентное содержание гепов равно 0!
Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам.
| Идентификатор БД | ECOT_SHEON ; Q8EEQ7.1 |
| E-value | 5e-29 |
| Вес (в битах) | 121 |
| % идентичности | 41% |
| % сходства | 62% |
| Длина выравнивания | 148 |
| Координаты выравнивания (номера первых и последних а.о.) | 6-153 |
| % гэпов | 1% |
Данный белок принадлежит таксону Alteromonadales.К сожалению среди других таксонов обнаружить гомолога вообще не удалось (с соответственным значениием E-value).
Этот белок принадлежит организму Shewanella oneidensis.Его таксономия: Bacteria; Proteobacteria; Gammaproteobacteria; Alteromonadales;
Shewanellaceae; Shewanella.
Они выполняют очень схожие функции.Вероятнее всего они действительно гомологи.
Среди других тасонов не обнаружено гомологов, скорее всего потому что белок ECOT_ECOLI малоизучен. Либо "возможные гомологи" ещё не открыты.
Поиск белка по его фрагменту.
Мне был дан следующий фрагмент последовательности:
>seq2
CPDGKKTEAFVPVVGDGFLLRYN.
Бластом был выдан следующий результат:белок ECOT_PSEPG
fasta-формат:
>gi|189027957|sp|B0KU25.1|ECOT_PSEPG RecName: Full=Ecotin; Flags: Precursor MRPTPMTAILALTLAAAAPAMAASLKDVAPYPEAEKGFTRQVIHLPAQADESAYKLEILAGKTLKVDCNR QRLGGSLEERTLEGWGYNYYRLDKVSGPASTLMACPDGKKTEAFVPVVGDGFLLRYNSKLPVVVYVPKDV EVRYRVWSASQDVQKANVE
| Поиск по фрагменту | Поиск по полной последовательности |
|
| АС лучшей находки | B0KU25 | B0KU25.1 |
| E-value | 9e-15 | 2e-89 |
| Вес (в битах) | 77 | 327 |
| Найдены ли другие белки с теми же значениями E-value и веса в битах? | Да,найдены ещё два белка с тем же весом и E-value.Их АС: Q88IC7.1 и A5W3S6.1 |
Нет.Больше белков с такими же параметрами поиск не выдал. |
Итак,мы видим,что E-value,выданное в поиске по фрагменту больше,чем результат,полученный при поиске полной последовательности.
Это объяснется тем,что в формуле по вычислению E-value существует такая зависимость:чем больше вес выравнивания,тем меньше E-value.
А соответственно,вес выравнивания больше получен по поиску последовательности,так как её длина больше длины фрагмента. Поэтому поиск по
полной последовательности более эффективен, т.к. вероятность, что найдется белок с таким же весом выравнивания гораздо меньше, чем при поиске по фрагменту.
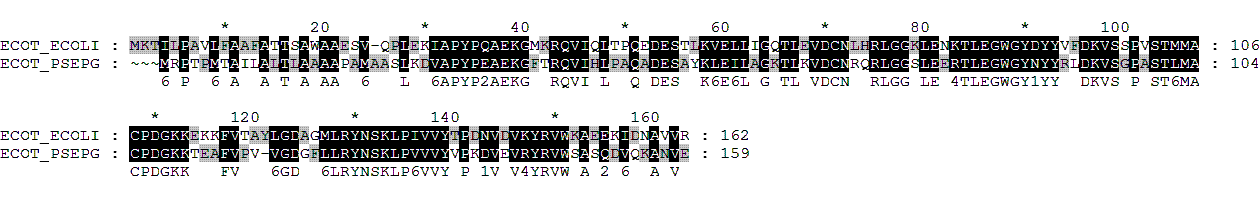
Зная последовательность белка B0KU25.1,произведём выравнивания (глобальное и локальное).
Глобальное:
Длина: 163

Локальное:
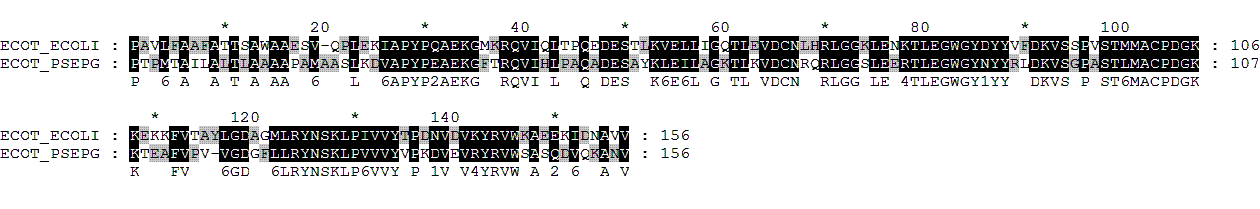
Выравнивание полученное бластом:
Score = 183 bits (465), Expect = 2e-46, Method: Compositional matrix adjust.
Identities = 90/157 (57%), Positives = 112/157 (71%), Gaps = 2/157 (1%)
Query 3 PTPMTAILALTLAAAAPAMAASLKDVAPYPEAEKGFTRQVIHLPAQADESAYKLEILAGK 62
P + A A T A AA ++ L+ +APYP+AEKG RQVI L Q DES K+E+L G+
Sbjct 6 PAVLFAAFATTSAWAAESVQ-PLEKIAPYPQAEKGMKRQVIQLTPQEDESTLKVELLIGQ 64
Query 63 TLKVDCNRQRLGGSLEERTLEGWGYNYYRLDKVSGPASTLMACPDGKKTEAFVPV-VGDG 121
TL+VDCN RLGG LE +TLEGWGY+YY DKVS P ST+MACPDGKK + FV +GD
Sbjct 65 TLEVDCNLHRLGGKLENKTLEGWGYDYYVFDKVSSPVSTMMACPDGKKEKKFVTAYLGDA 124
Query 122 FLLRYNSKLPVVVYVPKDVEVRYRVWSASQDVQKANV 158
+LRYNSKLP+VVY P +V+V+YRVW A + + A V
Sbjct 125 GMLRYNSKLPIVVYTPDNVDVKYRVWKAEEKIDNAVV 161
Выравнивание из BLASTP отличается от выравнивания, сделанного вручную, наличием гэпа на 117 позиции первой последовательности.
Выравнивание, сделанное BLASTP, имеет больший вес, что в принципе логично, ведь делая выравнивание вручную,можно не заметить более выгодного варианта.
Сравнение выравниваний, выданных программой BLASTP, с оптимальными глобальным и локальным выравниваниями.
Все три выравнивания практически не отличаются, заисключением первых 5 позиций глобального выравнивания.
Т.к. выравнивания сделанные BLASTP-ом и программой water являются локальными, вполне закономерно, что они выкинули
невыравненный участок на одном из концов. Штрафы за гэп во всех выравниваниях одинаковые(11 - за открытие гэпа;
1- за его продление. Во всех выравниваниях только один гэп(на 117 позиции первой последовательности, например в выравнивании BLASTP).