Аннотация
В данной статье представлен обзор генома Aquibium oceanicum. Исследуется распределение последовательностей, кодирующих белки и РНК по репликонам, длины белков, частоты стоп-кодонов, а также cumulative GC-skew.
1 ВВЕДЕНИЕ
Aquibium oceanicum — грамотрицательная, палочковидная, каталаза- и оксидаза-позитивная аэробная бактерия, выделенная из культуры Microcystis aeruginosa. Рост наблюдается в температурном диапазоне 23–45 °C (оптимум, 33 °C), при значениях pH 6–11 (оптимум, 8), в присутствии 0–4% NaCl (оптимум, 0%). Основными полярными липидами являются фосфатидилэтаноламин, фосфатидилхолин и фосфатидилметилэтаноламин. В таблице (таблица 1) представлена информация о систематическом положении Aquibium oceanicum. Геном данной бактерии состоит из одной хромосомы (5158483 пар оснований) (Рис.1) и одной плазмиды (166078 пар оснований) [1].

Рис. 1. Кольцевая хромосома Aquibium oceanicum [7].
Данная бактерия является довольно малоизученной и была открыта недавно. Согласно полученным при анализе 16S рРНК данным, наиболее близкородственный Aquibium oceanicum организм — Mesorhizobium carbonis (97,2% сходства) [1]. В данной работе был проанализирован геном Aquibium oceanicum и описаны некоторые его особенности, в частности, распределение последовательностей по репликонам, длины белков, частоты стоп-кодонов и GC-scew.
2 МАТЕРИАЛЫ И МЕТОДЫ
Данные о геноме Aquibium oceanicum, такие как feature table, геномная последовательность, последовательность кольцевой хромосомы и кодирующие последовательности получены с сайта NCBI [2]. Для анализа распределения генов белков и РНК по репликонам, длин белков и частоты стоп-кодонов была использована платформа Google Sheets [3]. Методы на платформе Google Sheets:
-
Подсчет количества белков разных длин и распределения по репликонам с помощью функции «СЧЕТЕСЛИМН».
-
Построение гистограмм для всех результатов.
Для нахождения частоты стоп-кодонов к файлу кодирующих белки последовательностей был применен авторский программный код на языке Python 3 [4]. Для анализа cumulative GC-skew к файлу последовательности кольцевой хромосомы был применен авторский программный код на языке Python 3 [5], а также использована программа Webskew [6]. Схема кольцевой хромосомы получена с помощью программы SnapGene Viewer [7].
| Ранг таксона | Название таксона |
|---|---|
| Домен | Bacteria |
| Филум | Pseudomonadota |
| Класс | Alphaproteobacteria |
| Отряд | Hyphomicrobiales |
| Семейство | Phyllobacteriaceae |
| Род | Aquibium |
| Вид | Aquibium oceanicum |
Таблица 1. Таксономическое положение бактерии.
3 РЕЗУЛЬТАТЫ
3.1 Распределение генов белков и РНК по репликонам
В таблице (таблица 2) представлены данные о количестве последовательностей разных типов: генов белков и разных типов РНК. Можно сделать вывод, что у исследуемой бактерии есть одна хромосома и одна плазмида. На хромосоме закодированы последовательности белков (CDS), а также все виды РНК (tRNA=тРНК — транспортная РНК, tmRNA=тмРНК — транспортно-матричная, rRNA=рРНК — рибосомальная РНК, ncRNA — некодирующая РНК). На рисунке (Рис. 2) наглядно изображены доли разных последовательностей на хромосоме. Нетрудно заметить, что подавляющее большинство составляют CDS (98,9%). В это время на плазмиде расположены только CDS.
| genomic_accession | seq_type | CDS | tRNA | ncRNA | rRNA | tmRNA |
|---|---|---|---|---|---|---|
| NZ_CP018171.1 | chromosome |
4996 |
48 |
3 |
6 |
1 |
| NZ_CP018172.1 | plasmid |
186 |
0 |
0 |
0 |
0 |
Таблица 2. Гены белков и РНК на разных репликонах.

Рис. 2. Доли разных последовательностей на хромосоме.
3.2 Длины белков
Данные о длинах белков Aquibium oceanicum отражены на гистограмме (Рис. 3). Наибольшее число белков рассматриваемой бактерии состоят из 200-300 аминокислотных остатков (а.о.). От этого диапазона при увеличении длины белка, количество убывает. Длина наибольшего белка расположена в диапазоне 1800-1900 а.о. Также имеется большое количество белков длиной 100-200 а.о. (1202 штуки) и меньше длиной 0-100 а.о. (480 штук). Видно, что от пика количества белков (200-300 а.о.) в сторону уменьшения длины, число белков меняется более резко, чем в сторону увеличения длины.

Рис. 3. Гистограмма длин белков.
3.3 Определение частот стоп-кодонов генов
Данные о частотах стоп-кодонов в кодирующих белки последовательностях представлены на гистограмме (Рис. 4). Видно, что у Aquibium oceanicum все три стандартных стоп-кодона располагаются на концах CDS. Можно сделать вывод, что они не утратили свою функцию. Помимо них, в последовательности содержат и 23 кодона, нестандартных для последнего кодона кодирующей белок последовательности. Но они встречаются 1–4 раза каждый. К такому могли привести точечные замены нуклеотида в стоп-кодоне (в случаях, где стоп-кодон отличается от стандартного на один нуклеотид), а также мутации со сдвигом рамки считывания. Возможно, это и артефакт. Также нельзя не обратить внимание на преобладание частоты стоп-кодона TGA над другими стандартными стоп-кодонами. В случае преобладания над кодоном TAA это может быть связано c GC-составом. А также с разным количеством факторов высвобождения (release factor, RF1 и RF2), которые распознают стоп-кодоны при терминации трансляции. Также согласно одному исследованию, стоп-кодоном TAA часто заканчиваются гены с высоким уровнем экспрессии. Это связано с тем, что оба фактора высвобождения узнают TAA [8]. Значит, стоп-кодон, которым заканчивается каждый конкретный ген, может быть связан с функцией этого гена.

Рис. 4. Гистограмма частот стоп-кодонов.
3.4 Определение cumulative GC-skew
Зависимость cumulative GC-skew от конкретного участка генома Aquibium oceanicum представлена на графиках (Рис. 5 и Рис. 6). Первый график получен в результате работы авторского программного кода [5], а второй при помощи сторонней программы [6]. Оба анализа дали близкие значения, следовательно, авторский код работает верно. Согласно исследованию, по минимуму значения cumulative GC-skew можно предсказывать расположение точки начала репликации на кольцевой бактериальной хромосоме [9]. По полученным в ходе работы данным можно предположить, что точка начала репликации находится вблизи точки 4684000 пар оснований (base pairs, bp) от начала последовательности. Однако проверка предположения в настоящее время невозможна, так как геном бактерии изучен плохо, точка начала репликации не обнаружена и в данных о геноме не указана.

Рис. 5. Зависимость cumulative GC-skew от конкретного участка генома.
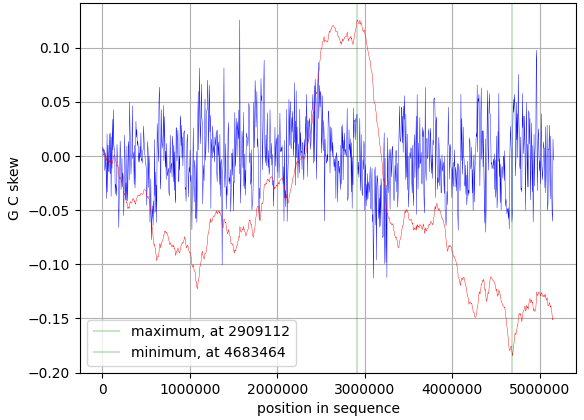
Рис. 6. Зависимость cumulative GC-skew от конкретного участка генома. Нужный график выделен красным цветом[4].
4 СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
-
Таблица на платформе Google Sheet
Общая таблица данных по геному на странице «feature_table». Распределение последовательностей по репликонам на странице «per-replicones». Длины белков на странице «protein_len-hist». Частоты стоп-кодонов на странице «stop_codons». Анализ cumulative GC-skew на странице «GC-skew».
- Программный код на языке Python 3 для нахождения частоты стоп-кодонов
- Программный код на языке Python 3 для нахождения cumulative GC-skew
ЛИТЕРАТУРА
-
Kim M, Kim W, Park W. Aquibium microcysteis gen. nov., sp. nov., isolated from a Microcystis aeruginosa culture and reclassification of Mesorhizobium carbonis as Aquibium carbonis comb. nov. and Mesorhizobium oceanicum as Aquibium oceanicum comb. nov. Int J Syst Evol Microbiol. 2022 Jan;72(1). doi: 10.1099/ijsem.0.005230. PMID: 35038289.
- source_2
- source_6
- source_7
-
Korkmaz G, Holm M, Wiens T, Sanyal S. Comprehensive analysis of stop codon usage in bacteria and its correlation with release factor abundance. J Biol Chem. 2014 Oct 31;289(44):30334-30342. doi: 10.1074/jbc.M114.606632. Epub 2014 Sep 12. PMID: 25217634; PMCID: PMC4215218.
-
Arakawa K, Tomita M. The GC skew index: a measure of genomic compositional asymmetry and the degree of replicational selection. \ Evol Bioinform Online. 2007 Sep 6;3:159-68. PMID: 19461976; PMCID: PMC2684130.