Цель этого практикума — попробовать разные алгоритмы поиска BLAST через веб-интерфейс и локально.
1 Поиск в геноме нейроспоры гена, кодирующего δ-субъединицу АТФ-синтазы
Идентификатор белка: XP_957271.2
Последовательность белка:
ATP_synthase_subunit_de.faa
Идентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок:
NC_026503
Файл с последовательностью небольшой окрестности этого гена в FASTA-формате:
NC_026503.1[3664975..3666379].fa
Рис. 1. Neurospora crassa
Автор изображения: Namboori B. Raju
2 Использование разных вариантов BLAST для фрагмента ДНК
Я решил изучить консервативность АТФ-синтазы, одного из наиболее широко распространённых
белков среди аэробов. Для своей цели я использовал последовательность δ-субъединицы,
входящей в состав АТФ-синтазы Neurospora crassa.
Для поиска и сравнения в BLAST я выбрал семейство Felidae (кошачьи), так как оно
достаточно удалено от моего организма в эволюционном смысле (ещё бы, гриб и кошка!).
Ну и меня Лев зовут :-)
Поиск проводился в базе refseq_genomes по 15 геномам кошачьих.
Вначале я решил использовать megablast, так как он предназначен для
поиска последовательностей высокой схожести, а я предположил,
что АТФ-синтаза должна быть достаточно консервативна, учитывая её важность для клеток.
Позже окажется, что это совершенно проигрышная стратегия, извините, тупанул.
Длина слова была установлена по умолчанию на 28.
Однако результатов найдено не было, поэтому я решил, что гриб нейроспора и кошачьи
слишком далеки друг от друга эволюционно, чтобы использовать megablast. Поэтому я решил проверить blastn.
Длина слова была установлена по умолчанию на 11. Однако всё равно находок не нашлось... Изменение длины слова до 7,
максимального числа находок до 5000, а также уменьшение штрафов за несовпадение и гэп также не помогло.
В общем этого и следовало ожидать, так как я сравнивал гены белков отдаленных организмов на уровне нуклеотидов, а не аминокислот.
Затем я решил провести поиск при помощи tblastx с параметрами по умолчанию. Я предпочел именно его tblastn,
так как tblastx принимает на вход нуклеотидную последовательность и проводит
сравнение между разными вариантами (шесть штук)
аминокислотных последовательностей, полученных из нуклеотидных.
Для tblstn же нужен белковый запрос, а у меня есть нуклеотидный.
То есть шанс найти совпадения выше. На этот раз нашлось 15 результатов (Рис. 4.),
как раз по числу геномов, по которым шёл поиск.
Все результаты из разных геномов. Успех, победа, это и было нужно!
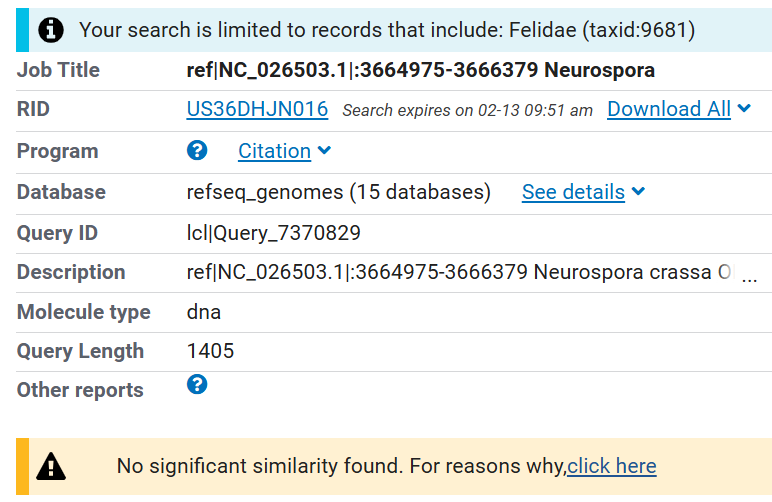
Рис. 2. Результаты megablast
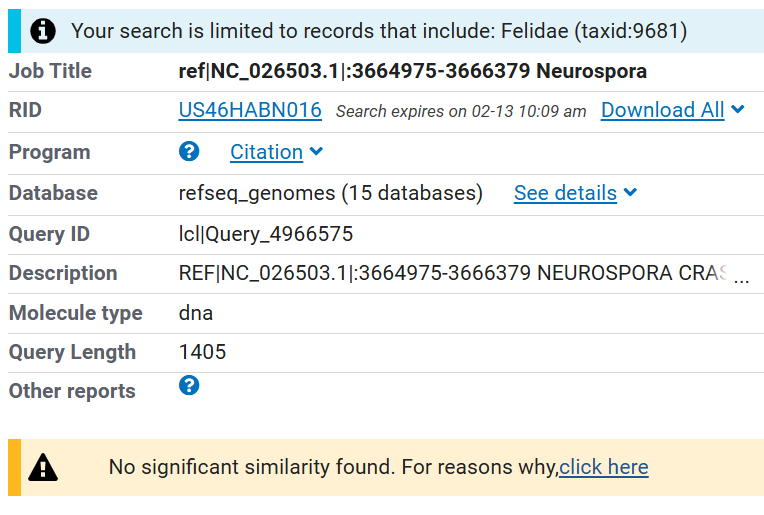
Рис. 3. Результаты blastn
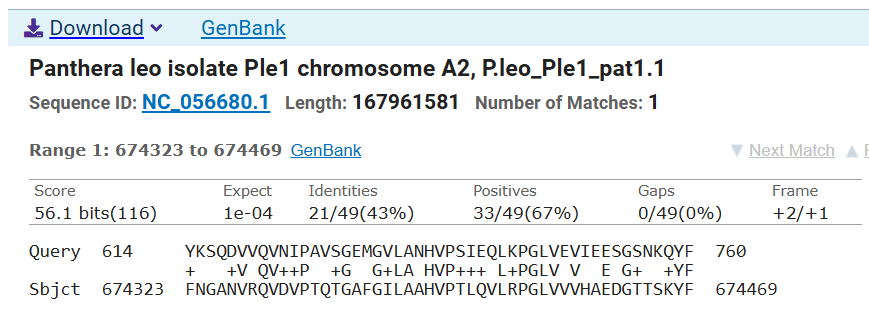
Рис. 5. Лев — находка tblastx
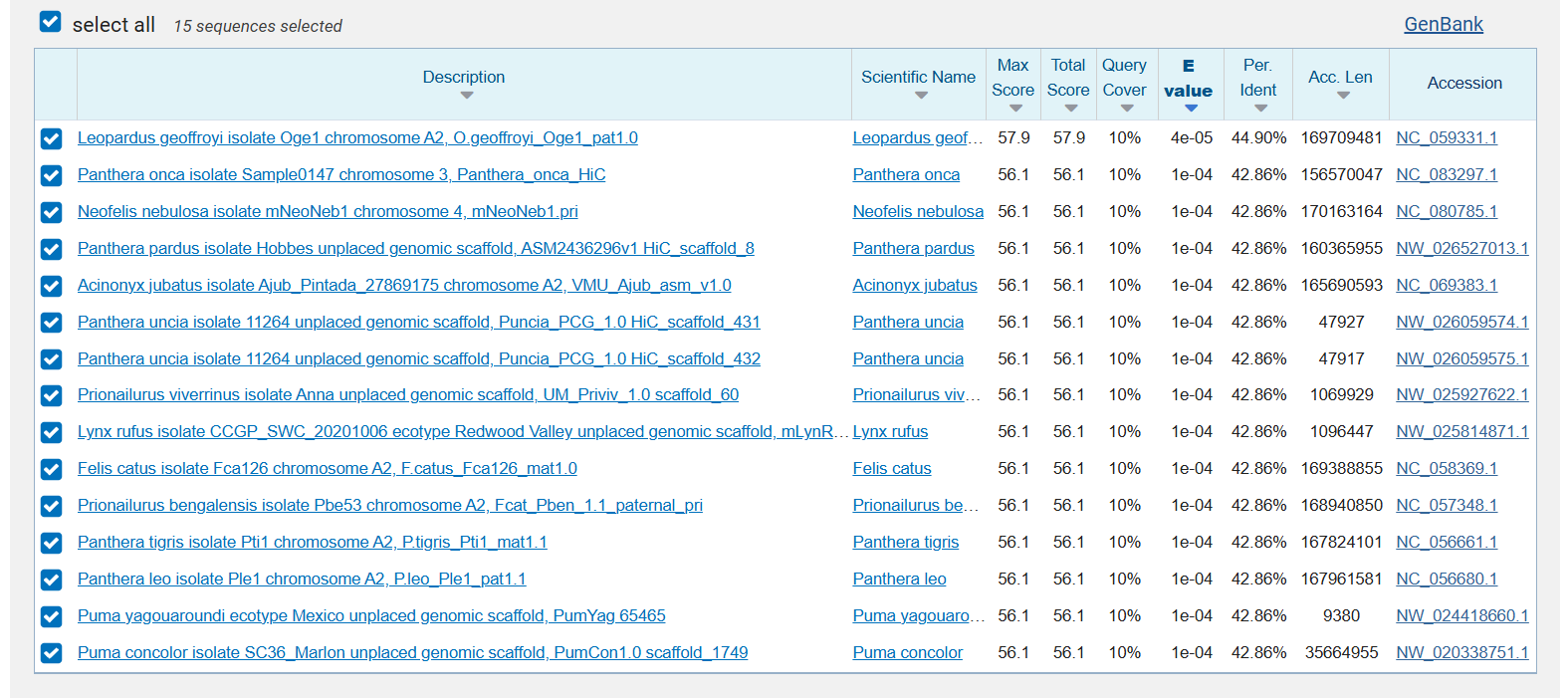
Рис. 4. Результаты tblastx
3 Поиск в геноме нейроспоры генов основных рибосомальных РНК по далекому гомологу
Для индексации генома нейроспоры использовал следующую команду:
makeblastdb -in GCF_002007445.2_ASM200744v3_genomic.fna -dbtype nucl
Я провел локальный поиск BLAST по 16S и 23S рРНК Escherichia coli.
16S рРНК находится в малой субъединице рибосомы прокариот.
Она играет важную роль в распознавании последовательности
Шайн-Дальгарно на мРНК и обеспечивает структурную поддержку
рибосомы, что способствует ее стабильности и функционированию.
23S рРНК, расположенная в большой субъединице рибосомы прокариот,
участвует напрямую в синтезе белка. Она формирует активный сайт,
помогает перемещать транспортную и матричную РНК во время трансляции
и выступает одним из ключевых катализаторов при образовании пептидных связей.
Для поиска был выбран blastn, так как он как раз подходит для поиска некодирующих
белки нуклеотидных последовательностей у неблизкородственных организмов.
Из параметров я поменял длину слова на 7, чтобы нашлось больше.
Команды:
blastn -task blastn -query 16S_rRNA.fa -db GCF_000182925.2_NC12_genomic.fna -out blastn_16S.out -evalue 0.05 -word_size 7 -outfmt 7
blastn -task blastn -query 23S_rRNA.fa -db GCF_000182925.2_NC12_genomic.fna -out blastn_23S.out -evalue 0.05 -word_size 7 -outfmt 7
Результаты для 16S рРНК — 13 находок, из них к
к гомологичным последовательностям относятся последовательности в локусах NCU15719 (Рис. 6.), NCU15690, NCU15770, NCU17362, NCU15761, NCU15785 - это гены 18S рРНК -
это и есть 6 гомологов (остальное: псевдогены 19S из митохондриального генома). Я посмотрел на координаты subject (их можно найти по ссылке
Результаты для 16S рРНК, Рис. 7.)
и нашёл что находится по этим координатам на аннотированной
последовательности в геномном браузере. По e-value отсечь ничего не удалось. В качестве примера картинка из геномного браузера для локуса NCU15719 (Рис. 6.)
Для остальных локусов действия аналогичны.
Результаты для 23S рРНК - 20 находок, из них к
к гомологичным последовательностям относятся последовательности в локусах NCU15828, NCU17319, NCU15701, NCU15772, NCU15807 -
это гены 28S рРНК - это и есть 5 гомологов (остальное: псевдогены 29S из митохондриального генома). Нашел эти локусы тем же путем, что и для 18S.
Функции 28S и 16S рРНК у эукариот примерно аналогичны таковым у бактерий.
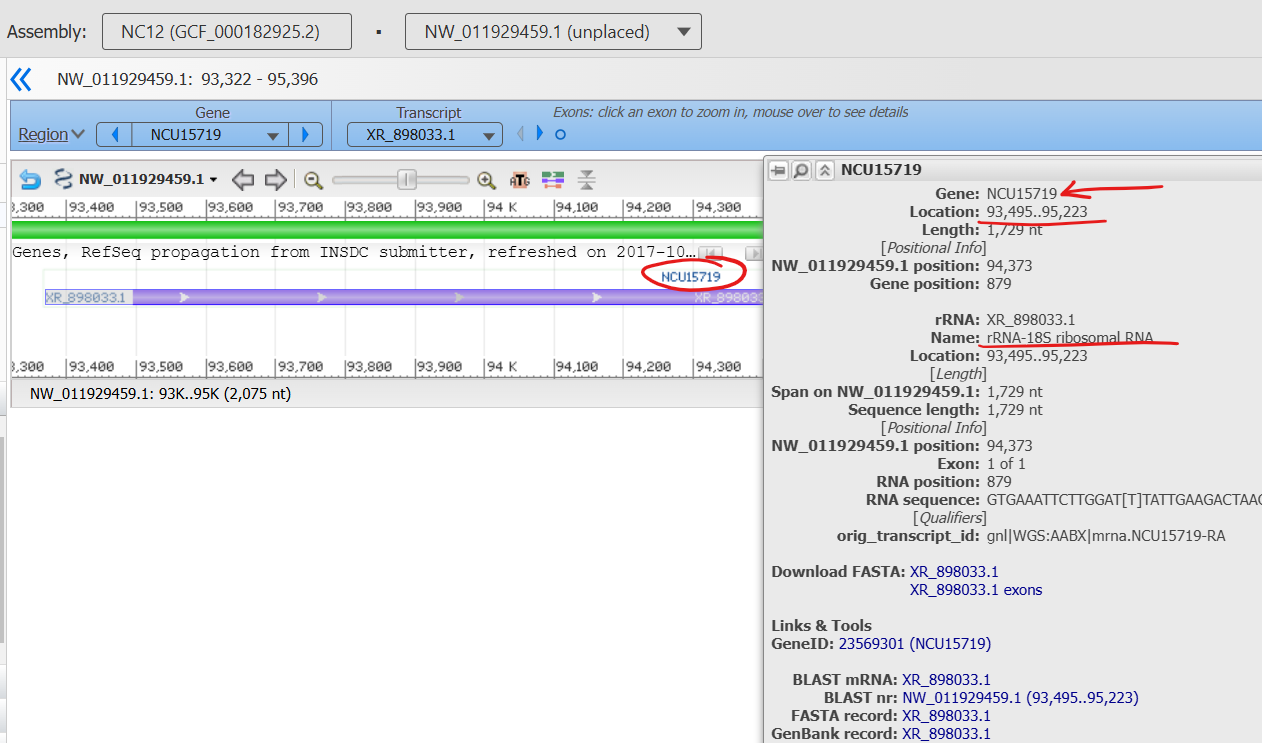
Рис. 6. Пример гомолога — ген 18S рРНК в локусе.
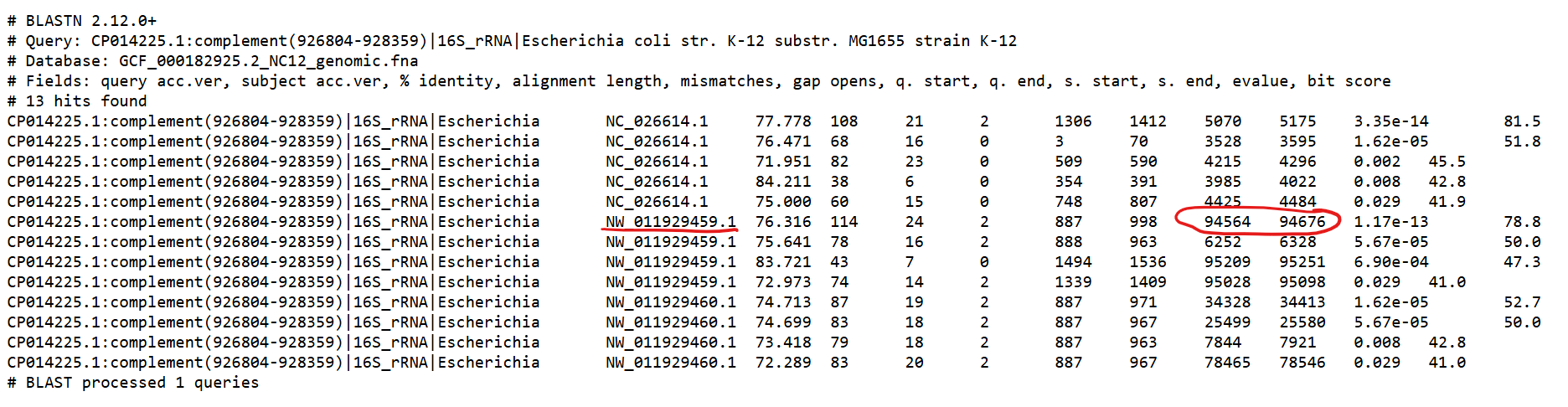
Рис. 7. Координаты subject по которым был найден локус NCU15719.
ЛИТЕРАТУРА
-
Galagan, James E et al. «The genome sequence of the filamentous fungus Neurospora crassa.» Nature vol. 422,6934 (2003): 859-68. doi:10.1038/nature01554