Практикум 10
или "ну что ж, подождем 4 утра, когда заработает BLAST"
Программа BLAST
1. Поиск в Swiss-Prot гомологов белка A0A0X8GZS8_9FIRM
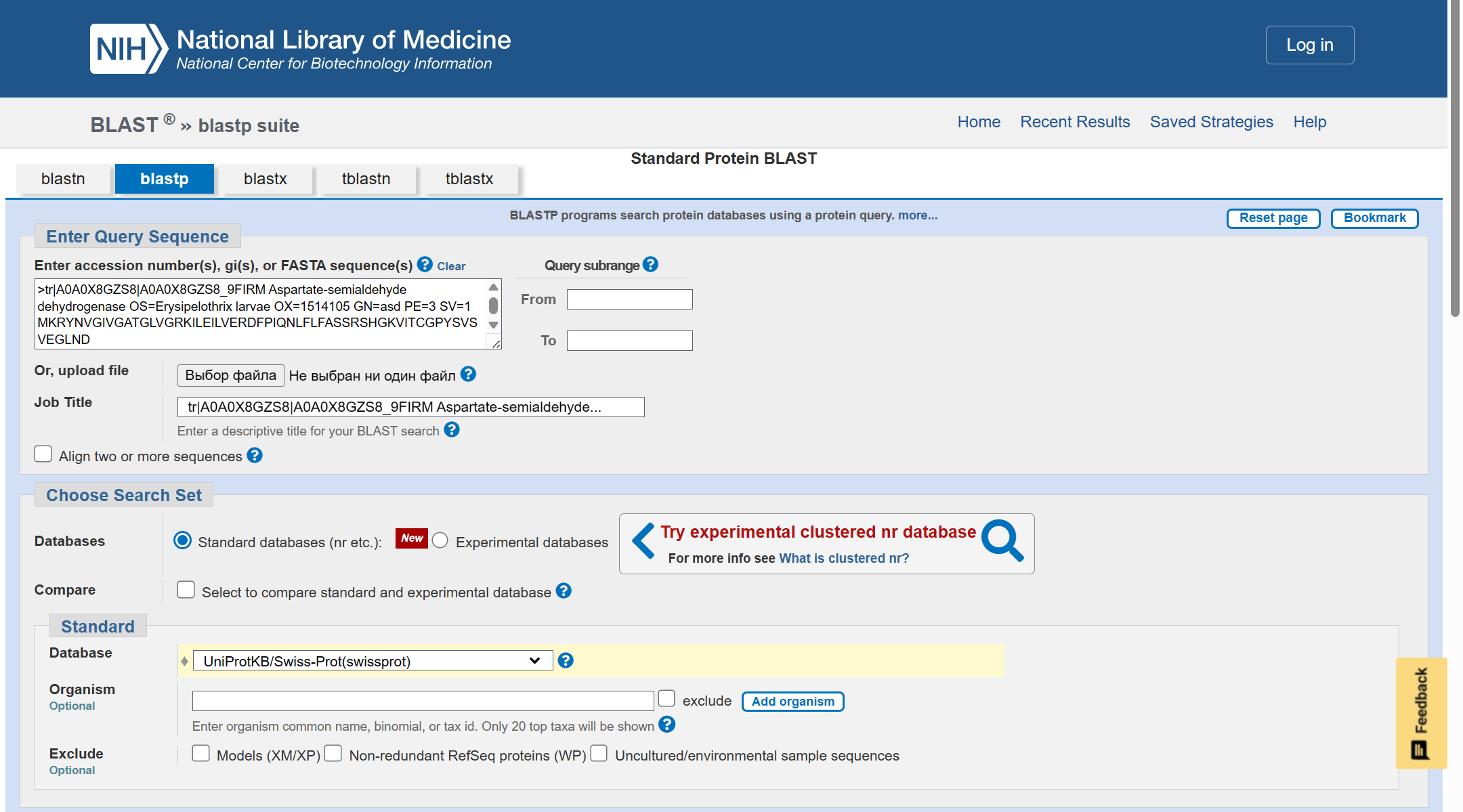
Параметры, использованные при запуске BLAST:
Enter Query Sequence: последовательность белка в fasta-формате
Databases: Stabdart databases(nr etc.)
Database: UniProtKB/Swiss-Prot(swissprot)
Algorithm: blastp(protein-protein BLAST)
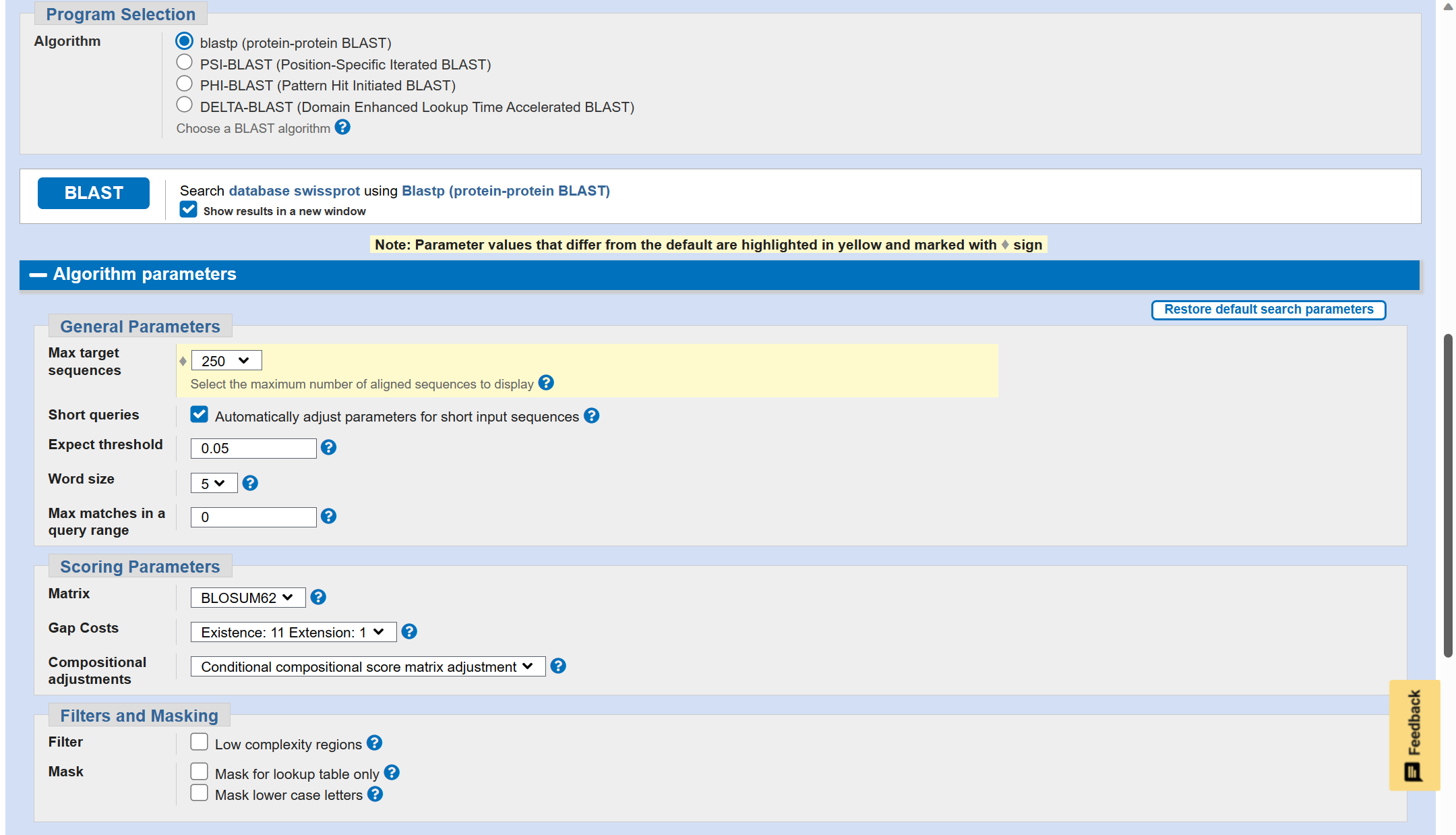
Algorithm parameters
Max target sequences: 250
Short queries: Automatically adjust parameters for short input sequences
Expect threshold: 0.05
Word size: 5
Matrix: BLOSUM62
Gap Costs: Existence: 11 Extension: 1
Выводы:
Текстовая выдача программы: ссылка на выдачу
Находки в fasta-формате: ссылка на находки
Т.к. моего исходного белка не было в Swiss-Prot, я добавила его в файл для выравнивания вручную. Использовала Muscle with defaults, а затем сделала окрашивание по проценту идентичности.
Множественное выравнивание: ссылка на выравнивание
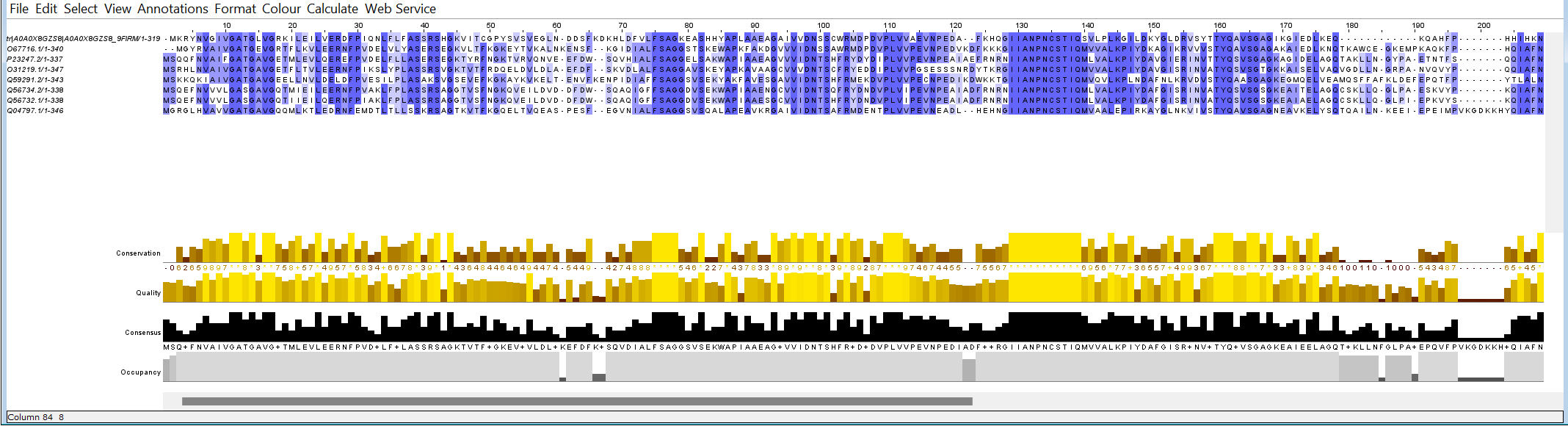
Из полученных данных можно заметить, что нередко белок с AC Q59291.2 "выпадает" из окрашенных блоков. Его E-value, при этом, существенно отличается от остальных: 3e-86(наибольший разрыв только у белка с AC P23247.2: 7e-92, но он в выравнивании вопросов не вызвал). В остальном же, можно сделать вывод, что оставшиеся белки гомологичны.
2. Поиск в Swiss-Prot гомологов зрелого вирусного белка, вырезанного из полипротеина
Выбор полипротеина
Небольшой дисклеймер: данный полипротеин был выбран только из-за его очень прикольного названия:)
OS: New York virus (NYV)
ID: GP_NYV
AC: Q83887
FT CHAIN: Glycoprotein C, 653-1140
Параметры, которые были использованы при запуске BLAST: аналогично пункту 1.
Выводы:
Вырезанная последовательность белка: ссылка на последовательность
Текстовая выдача программы: ссылка на выдачу
Находки с ненулевым E-value: ссылка на подходящие находки
Множественное выравнивание: ссылка на выравнивание
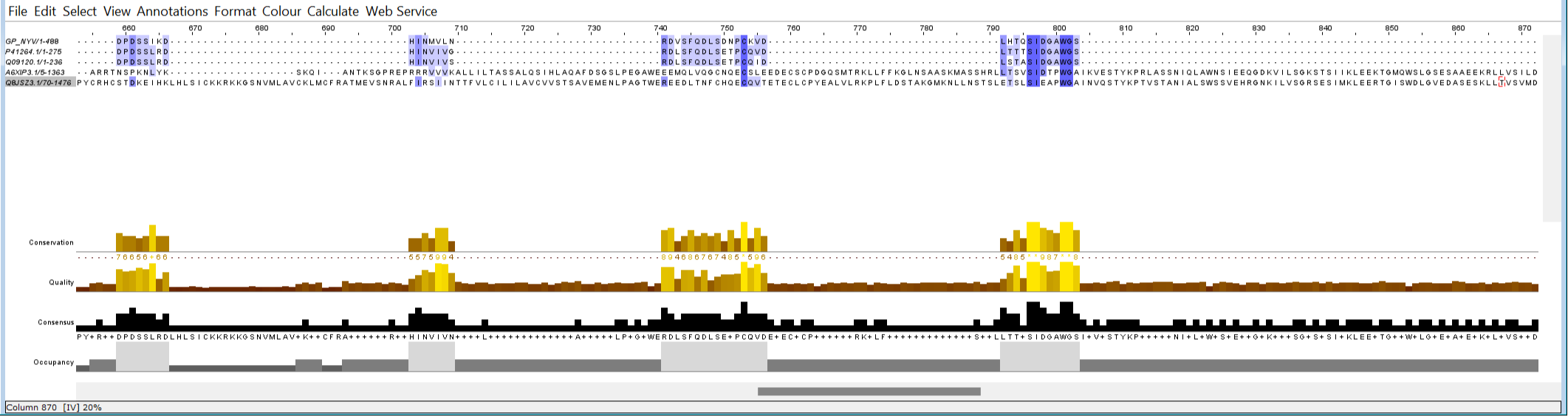
Из полученных данных можно сделать следующий вывод: исходный вирусный белок Glycoprotein C и белки с AC P41264.1, Q09120.1 гомологичны(большое количество идентичных блоков в выравнивании). Два оставшихся белка — нет(большое количество гэпов и несовпадений в выравнивании).
3. Исследование зависимости E-value от объёма банка
Я повторила предыдущий поиск, оставив те же параметры BLAST, лишь применила фильтр по организмам, ограничив поиск вирусами (Viruses). Список находок BLAST остался неизменным. Так, во втором пункте из заданий их было 22, после применения фильтра их также осталось 22. Однако изменились значения E-value. Так, например, до применения фильтра у белка с AC Q09120.1 E-value было 1e-115, а у белка с AC A6XIP3.1: 1e-04. После применения фильтра эти значения поменялись на 4e-117 и 4e-06, соответственно.
Применив формулу (E-value для поиска по вирусам)/(E-value для обычного поиска без фильтров) получила:
4e-117/1e-115 = 0.04 = 4%
4e-06/1e-04 = 0.04 = 4%
Таким образом получилось, что доля вирусных белков в Swiss-Prot составила ~ 4%