Практикум 9
или "What a ... Waterman?"
Выравнивание последовательностей
1. Глобальное парное выравнивание гомологичных белков
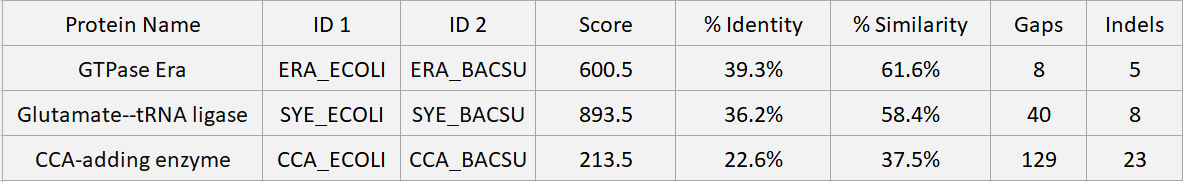
Таблица 1. Характеристики глобального парного выравнивания трёх пар белков
2. Локальное парное выравнивание гомологичных белков
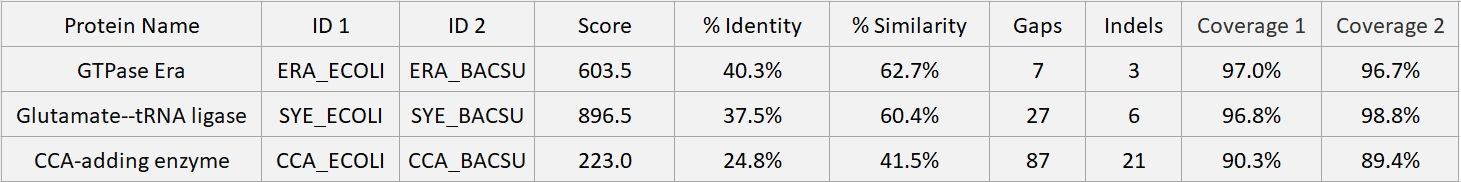
Таблица 2. Характеристики локального парного выравнивания трёх пар белков
3. Результат применения программ выравнивания к неродственным белкам
Для выполнения этого задания были выбраны два неродственных белка с разными мнемониками функций, а именно: AAT_ECOLI(Aspartate aminotransferase) и PAP_BACSU(Protein-arginine-phosphatase)
Таблица 3. Характеристики глобального парного выравнивания пары неродственных белков

Исходя из полученных данных глобального парного выравнивания можно увидеть, что процент идентичности очень низок, лишь 8.4%; процент схожести почти в два раза выше, ровно 16%. Однако все эти показатели крайне малы, что как раз и доказывает то, что выбранные белки не являются родственными.
Таблица 4. Характеристики локального парного выравнивания пары неродственных белков

Полученные в результате локального парного выравнивания данные хотя и выше, чем в глобальном выравнивании, однако все равно довольно низкие. Процент идентичности составил 24.3%, процент схожести — 39.8%. Эти значения, как и предыдущие, доказывают, что белки являются неродственными.
4. Множественное выравнивание белков и импорт в Jalview
Для данного задания были выбраны белки с мнемоникой функции ERA(GTPase Era). Мне просто очень понравилось звучание :)
Используя расширенный поиск Uniprot, я нашла все белки с этой мнемоникой. Затем выбрала те, которые есть в Swiss-Prot. Таких белков вышло 349. Недолго думая, я выбрала первые 7 белков из списка: ERA_ECOLI, ERA_BACSU, ERA_MYCS2, ERA_STRMU, ERA_THET8, ERA_MYCTA, ERA_STRR6
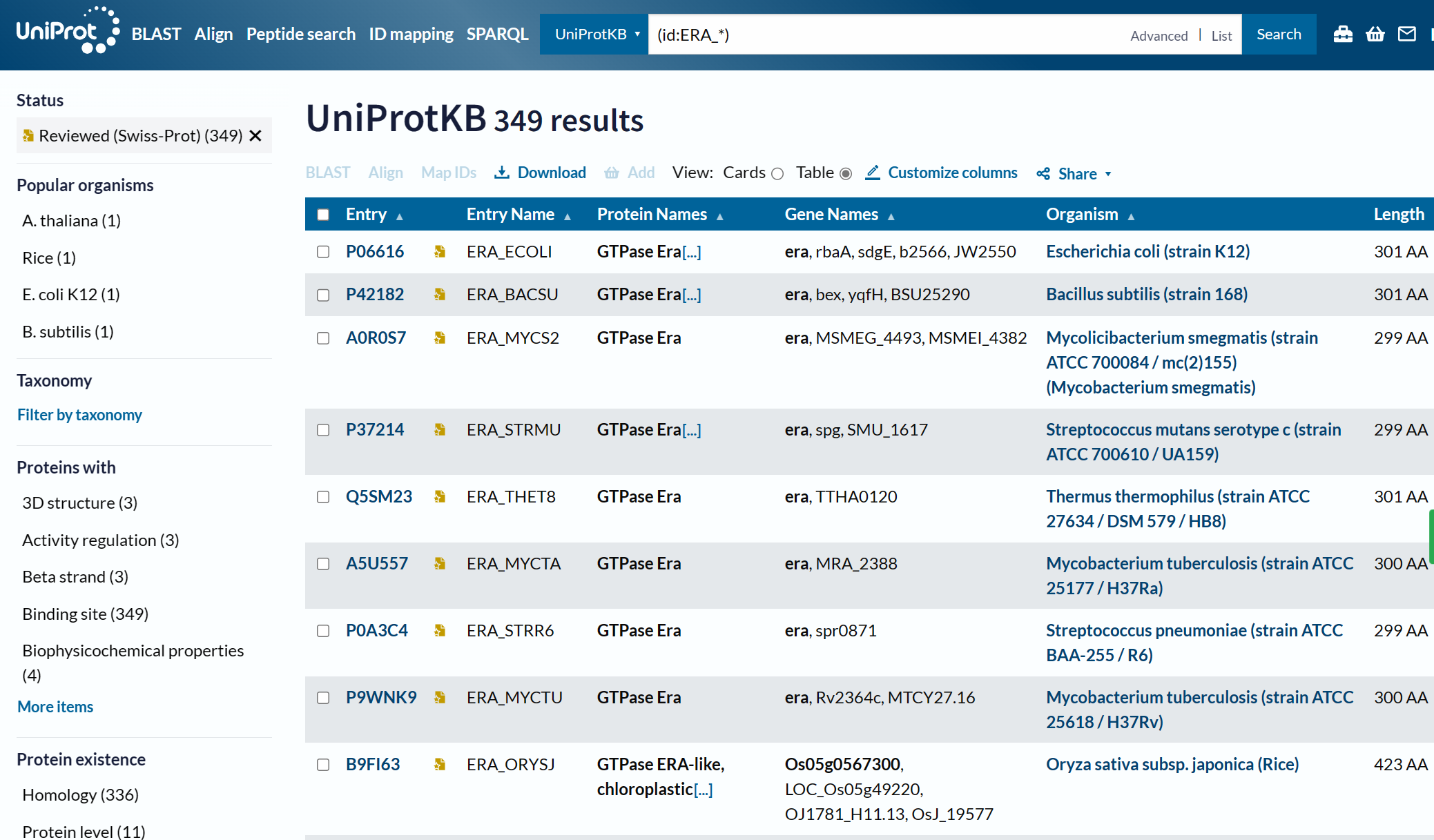
С помощью Jalview было построено множественное выравнивание. Если коротко рассмотреть процесс по пунктам, то: File => Fetch sequences => выбор базы Uniprot => ввод в поисковую строку выбранных заранее белков => Web service => Alignment => Muscle with defaults => раскраска по проценту идентичности => готово:)
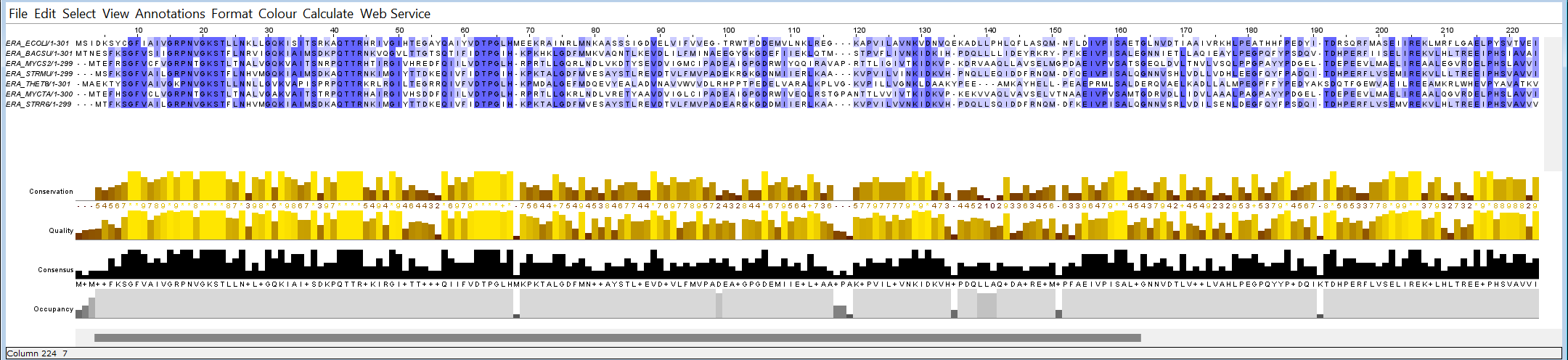

Исходя из результатов множественного выравнивания я бы не сказала, что все выбранные белки являются гомологичными. Слишком много белых, незакрашенных участков присутствует в получившемся выравнивании. Так, белок ERA_THET8 довольно часто "выпадает" из общей концепции. Подобное также можно заметить и с ERA_MYCTA и ERA_MYCS2. На участках 8, 19, 32, 38-39, 72, 76 и т.д. данные "пробелы" заметны очень хорошо.
Конечно, есть и абсолютно консервативные участки, как, например, 9-10, 20-23, 41-44, 62-65 и др. Но даже несмотря на наличие подобных консервативных блоков, я не могу сделать вывод о гомологичности данных белков.