Практикум 10
или "у кого какой мотив?"
Мотивы в белках
Задание 1.
Для данного практикума было решено выбрать PF09579 , Sporulation protein YtfJ (Spore_YtfJ). YtfJ был подтвержден в спорах B.subtilis , по-видимому, он экспрессируется в передней поре под контролем SigF. Для этого домена в seed находится 193 последовательности, ссылка на SEED.
Были скачаны последовательности seed и удалены те, у которых схожесть больше 85% (т.к. зачастую берут именно такую границу для последовательностей выше 100 позиций). После удаления у осталось 179 последовательностей. Более-менее консервативный участок, который получилось найти имел паттерн >IIP.S.V.{2}GF< . После поиска оказалось, что данный паттерн находится в 64 последовательностях из 179 упомянутых. Очевидно, что это не просто случайное совпадение.
Если перевести данный паттерн в формат Prosite, то получится следующее: >I(2)-P-x-S-x-V-x(2)-G-F< . К сожалению, ничего не нашлось :(
Попытка не пытка! Попробуем поискать еще, но в этот раз укоротим наш паттерн до >IIP.{3}V< .
Для такого паттерна уже нашлись 73 находки в выравнивании. Если перевести его в формат Prosite, то получится: >I(2)-P-x(3)-V< . И вот теперь поиск увенчался успехом! Нашлись целых 2585 последовательностей с 2596 хитами, то есть в некоторых последовательностях паттерн встречается более одного раза. По-моему, это круто :)
Файл с итоговым выравниванием для задания 1: ссылочка
Задание 2.
По нашему выравниванию было построено дерево. Было решено рассмотреть кладу, выделенную на дереве красным. Картинка дерева представлена ниже. Выбранные последовательности были вырезаны отдельно, а затем для них было построено отдельное выравнивание для нахождения какого-либо консервативного паттерна. На подходящем уровне консервативности таковым оказался следующий паттерн: >PL[IALVST]DV[STN][FVC][AG]< . В таком виде он присутствует во всех последовательностях данной клады. При этом при поиске по всему выравниванию данный паттерн не встретился больше нигде, поэтому мы нашли что-то действительно хорошее и специфичное для данной клады.
Файл с итоговым выравниванием для задания 2: ссылочка
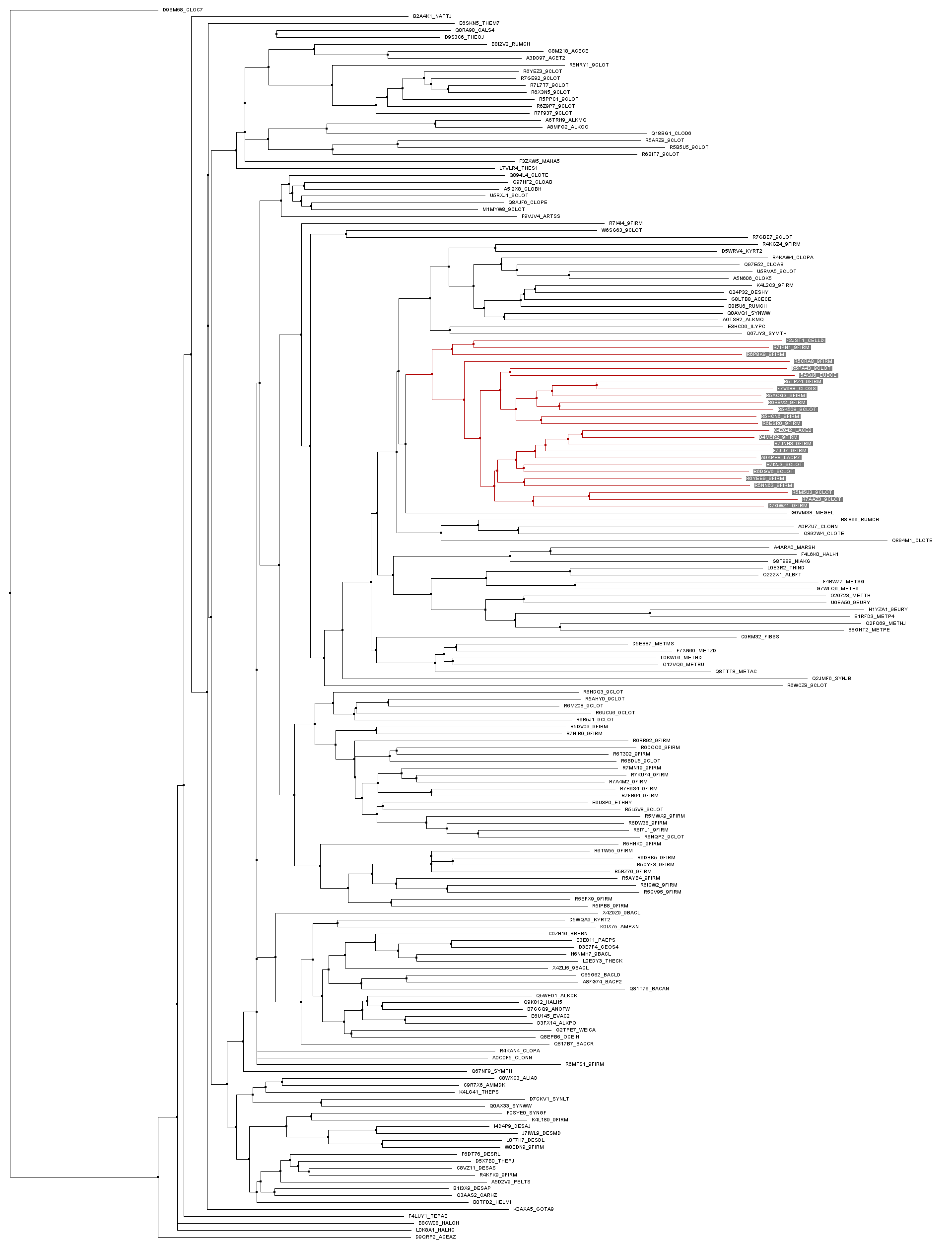
Рис. 1. Построенное дерево, описание выделенной красным клады представлено в тексте
Задание 3.
Для этого задания было решено использовать белок Q7VDL2 . Данный белок является ингибитором клеточного деления, который нарушает сборку белка FtsZ. Также можно заметить, что данный белок принадлежит к Prochlorococcus marinus.
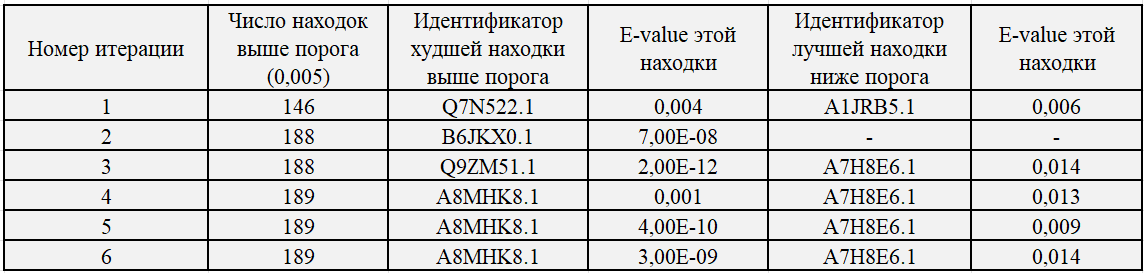
По итогу итераций получилась таблица, представленная ниже. Уже на 2 и 3 итерациях хорошо отделилось семейство, причем это семейство - MinC. И именно к этому семейству принадлежит наш исходный белок, что и ожидалось увидеть.
Однако на следующих итерациях к нашему белку добавился какой-то странный иной белок, не относящийся к нашему семейству. Возможно, в нем есть похожий домен, из-за чего он и попал в это "семейство". В остальном же, семейство выделилось прекрасно.
Таблица 1. Результат итераций PSI-BLAST при пороге 0.005
Задание 4.
В данном задании было решено рассмотреть архитектуру PF09579-PF09579, ссылочка на fasta. Для данной архитектуры в базе данных есть 10 белков, все они являются белками споруляции, что неудивительно, ведь данный домен и отвечает за споруляцию. Для скачивания и дальнейшего поиска мотивов использовали команду:
meme architect.fasta -o meme_results -minw 4 -nmotifs 5
Теперь поищем наши мотивы. Для этого возьмем вторую архитектуру, содержащую всего один домен, ссылочка на fasta . Для такой архитектуры в базе данных есть 6571 белок. По итогу поиска нашлось 29159 представленностей сайтов. Вероятнее всего, это связано с тем, что исходно мотивы были очень похожи и отражали примерно одно и то же, но были из двух соседних доменов архитектуры.
Задание 5.
Для этого задания была выбрана бактерия Mycobacterium smegmatis. Был скачан геном, ссылка на геном fasta и загружен на Кодомо. Затем был создан файл со всеми возможными сайтами, ссылка на сайты , и запущена программа на кодомо. Использованная команда выглядела так:
cbcalc -s sites -M -o res.tsv M_smegmatis.fasta
После того как мы получили файл, который хотели, построили гистограмму и, используя "метод пристального взгляда", определили, что наш сайт представлен в геноме настолько, насколько он ожидается статистически. Также интересно заметить, что в геноме сильно недопредставлен сайт CTAG. А еще можно заметить, что в большинстве случаев сайты недопредставлены в геноме, только три из них перепредставлены и лишь наш сайт, GATC, находится в золотой середине.
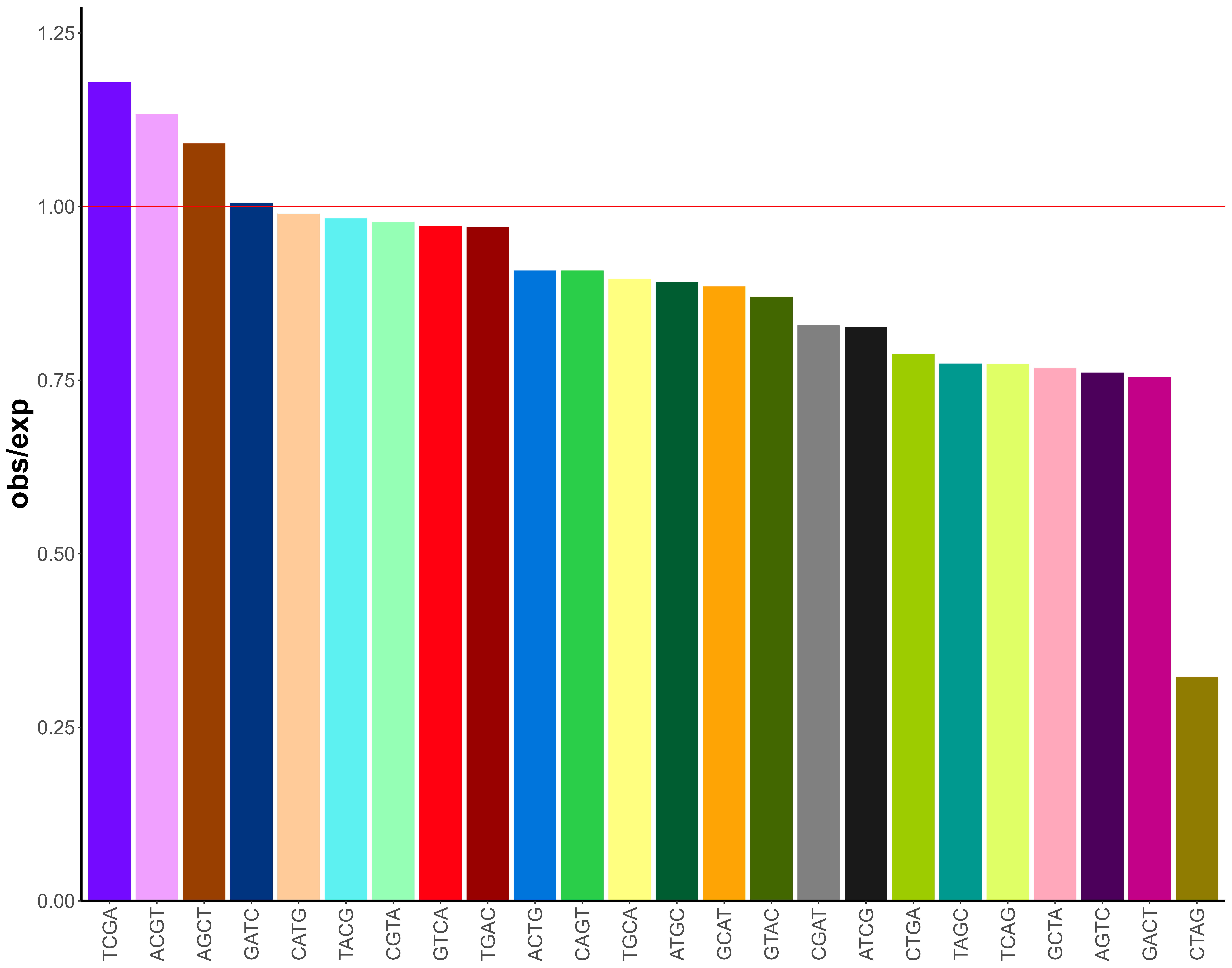
Рис. 1. Гистограмма представленности для всех сайтов длины 4, полученных перестановками (без повторений) букв A, T, G, C