Практикум 4
или красим ветки в "красный" цвет
Филогенетическое дерево
Отобранные бактерии
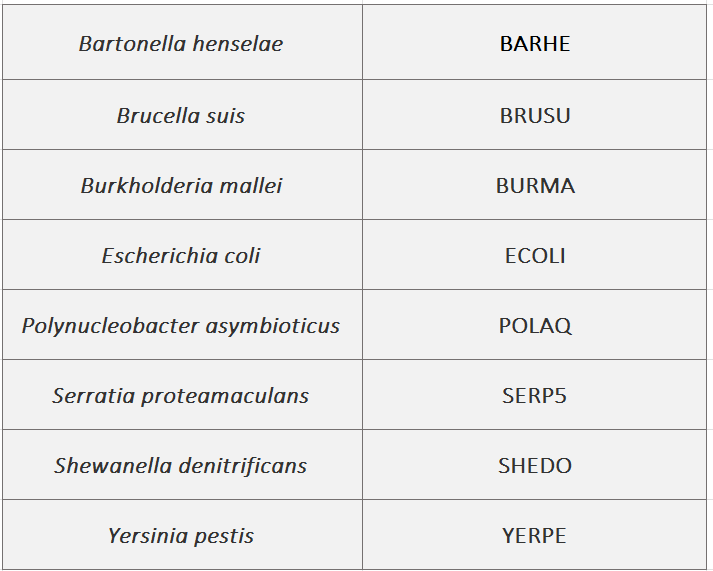
Скобочная формула дерева
((BRUSU, BARHE), ((POLAQ, BURMA), (SHEDO, (ECOLI, (YERPE, SERP5)))));
Изображение дерева
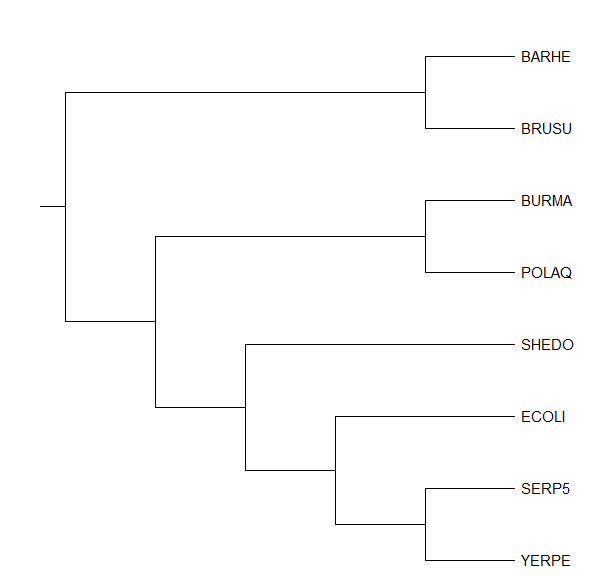
Рис.1. Полученное дерево
Составление списка гомологичных белков, включающих паралоги
Используя BLASTP, в протеомах выбранных ранее бактерий были найдены гомологи белка CLPX_ECOLI(АТФ-связывающая субъединица протеазы Clp).
Список находок можно посмотреть тут: тык
Последовательности находок здесь: тык
Реконструкция и визуализация
Дерево было создано программой FastME с параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик.
Newick формулу дерева можно посмотреть тут: тык
Ортологи: HSLU_BARHE и HSLU_BRUSU, CLPX_POLAQ и CLPX_ECOLI, RUVB_BARHE и RUVB_BRUSU
Паралоги: RUVB_BRUSU и HSLU_BRUSU, A4SXL5_POLAQ и A4SZP7_POLAQ, HSLU_SERP5 и CLPX_SERP5
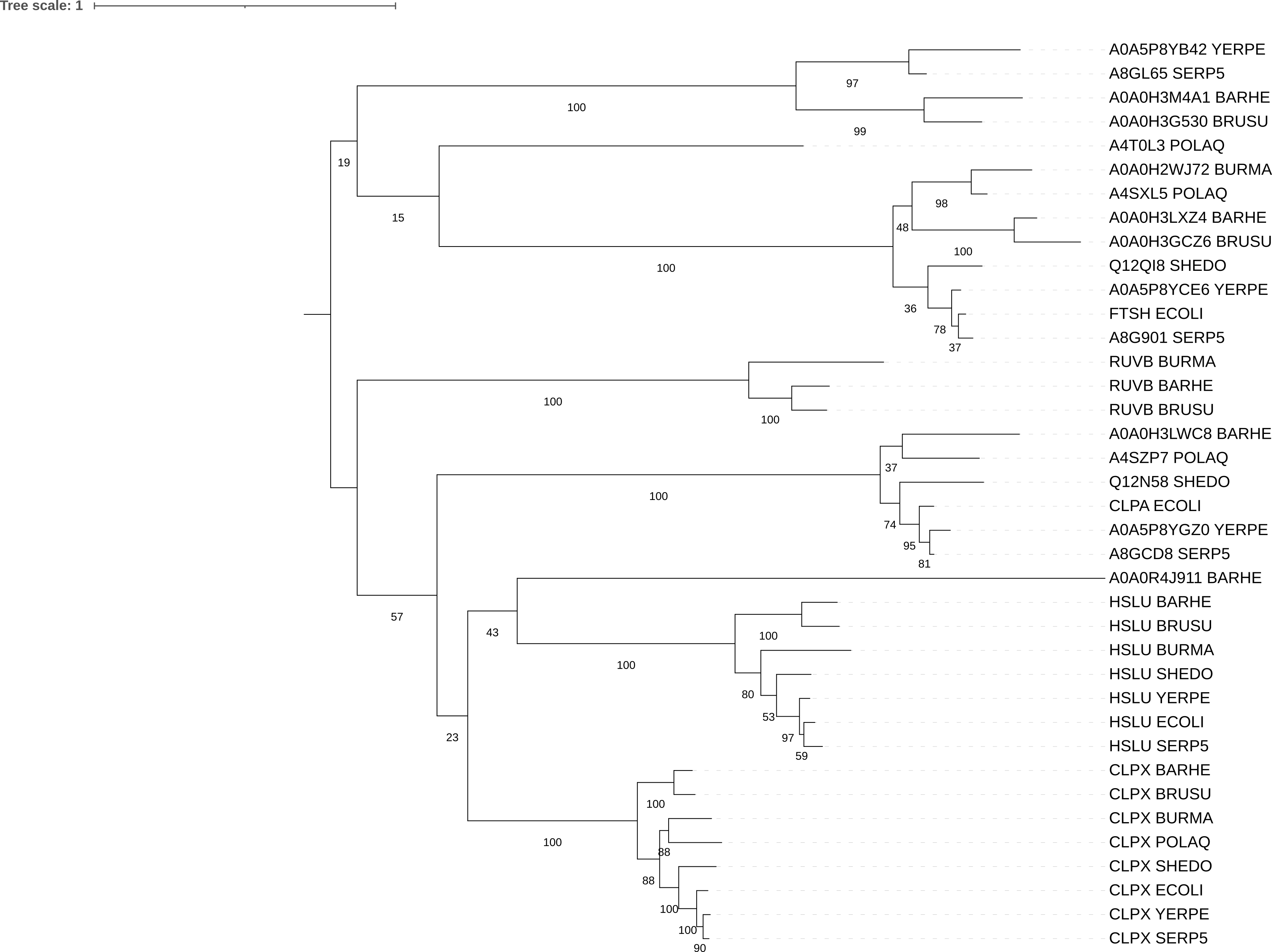
Рис.2. Дерево гомологов белка CLPX_ECOLI в протеомах выбранных бактерий
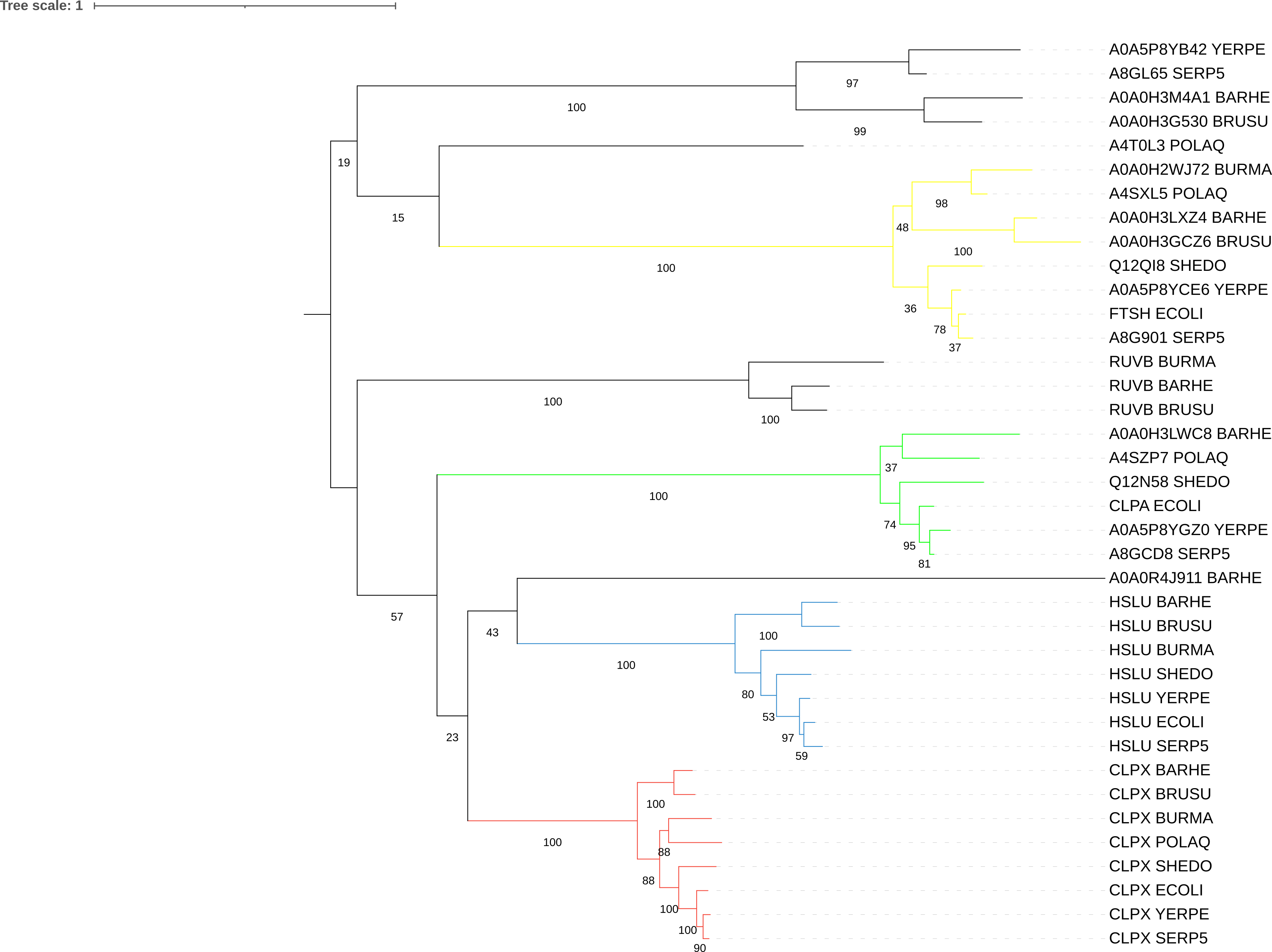
Рис.3. Дерево гомологов белка CLPX_ECOLI в протеомах выбранных бактерий с выделенными группами ортологичных белков, сверху-вниз: желтый цвет — АТФ-зависимые цинковые металлопротеазы FtsH, зеленый цвет — АТФ-связывающая субъединица ClpA протеазы Clp, синий цвет — АТФ-связывающая субъединица HslU протеасомоподобного комплекса деградации, красный цвет — АТФ-связывающая субъединица ClpX протеазы Clp. Реконструированная филогения белков-ортологов соответствует филогении бактерий.
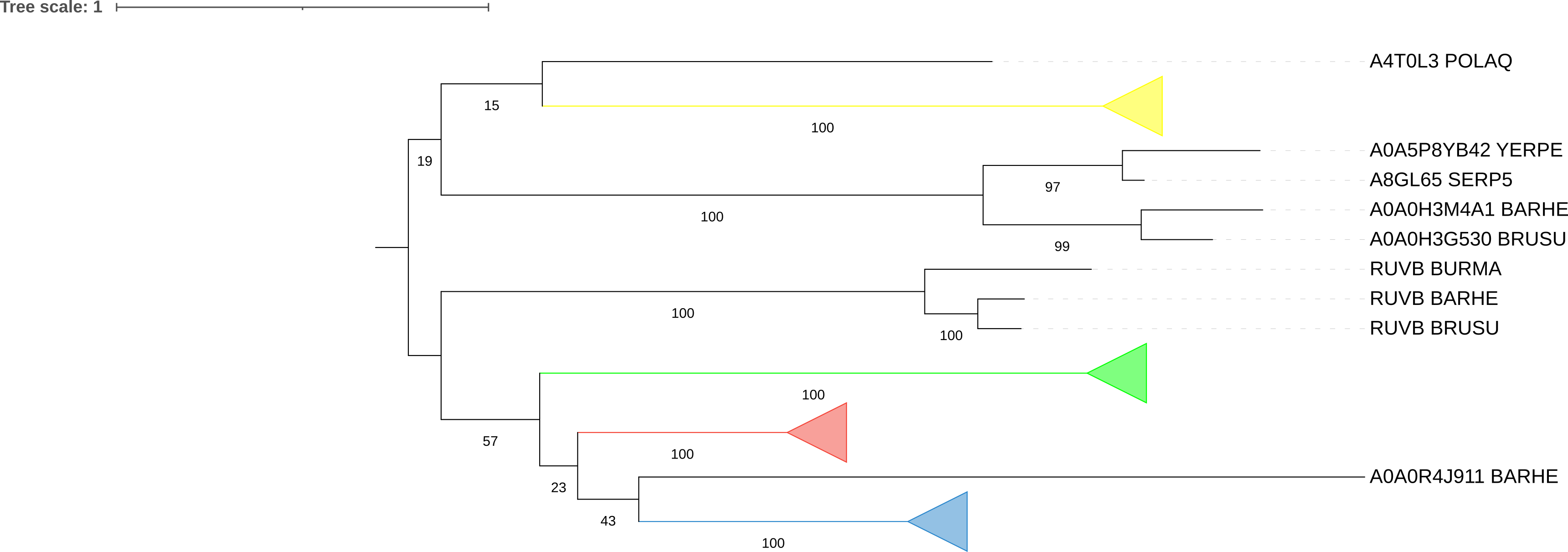
Рис.4. Дерево гомологов белка CLPX_ECOLI в протеомах выбранных бактерий. Ветви белков-ортологов схлопнуты, цветовой код аналогичен цветовому коду рис.3