Практикум 6
или "белочки мои белочки"
Трансмембранные белки
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения данного задания был выбран активатор плазминогена Yersinia Pestis Pla (Native). Данный белок локализован в бактериальной грам-отрицательной внешней мебране. Краткую информацию, а также необходимые ссылки можно увидеть ниже:
Класс — трансмембранный бета-бочонок
Суперсемейство — протеазы внешней мембраны (omptin)
Семейство — OMPT
Организм — Yersinia pestis (чумная палочка)
Локализация — внешняя мембрана грам-отрицательной бактерии
Uniprot — ссылка
PDB — ссылка
Толщина гидрофобного слоя — 25.6 Å
Число трансмембранных структур — 10
Средняя длина трансмембранных структур — 10, 11 (10.4)
Трансмембранные участки — 1(13-22), 2(51-60), 3(65-73), 4(111-121), 5(126-136), 6(174-183), 7(188-197), 8(224-235), 9(239-248), 10(282-292)
Выполняемая функция — расщепление плазминогена с образованием плазмина, который, предположительно, способствует распространению бактерий по всему организму от очага заражения
Последовательность белка была скачана из базы Uniprot. Ссылка на файл с последовательностью.
Предсказанные программой координаты трансмембранных участков:
(34,46)
(67,79)
(85,98)
(129,139)
(148,159)
(191,202)
(209,222)
(240,253)
(260,268)
(299,310)
Графическое изображение результатов DeepTMHMM
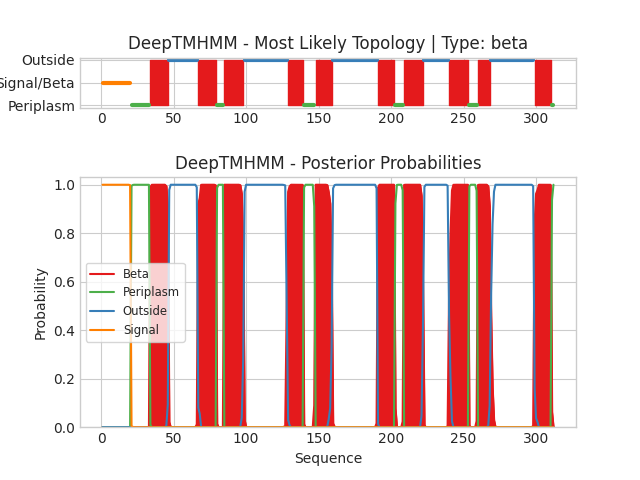
Рис.1. Графическое изображение результатов. Верхняя картинка — соответствие аминокислотных остатков различным классам: периплазматическая часть белка (зеленый), внешняя часть белка (синий), сигнальная последовательность (оранжевый) и трансмембранный участок (красный). Нижняя картинка — более подробное пояснение распределения участков, а именно: есть линии трех цветов (зеленый, синий, оранжевый); они отражают вероятность того, что данная аминокислота принадлежит определенному участку (периплазма, внешняя часть, сигнальная последовательность, соответственно). Также имеются участки красного цвета, трансмембранные, они определяются по пороговой вероятности того, что аминокислотный остаток принадлежит какому-либо из вышеуказанных ранее участков
Работа с файлами
Полученные файлы: .3line format, .gff3 format
Разберем подробнее полученные файлы
3line: файл содержит три строки: первая строка — просто AC, вторая строка — последовательность белка, третья строка — последовательность категорий аминокислот, где S — сигнальный участок, B — бета-лист (трансмембранный участок), P — периплазматический участок, O — внешний участок.
gff3: файл содержит табличку с конкретными координатами участков в виде TSV со следующими колонками: AC, категория участка, координата начала участка, координата конца участка.
А что по сравнению программ?
Если сравнивать выдачи двух программ, то можно прийти к следующему выводу: глобально обе программы нашли по 10 трансмембранных участков, однако в большинстве случаев эти регионы даже не пересекались. При этом, пересекающиеся участки почти полностью совпадали (3 и 2 участки, 5 и 4 участки, 7 и 6 участки, 9 и 8 участки)
Дополнительно можно увидеть визуализацию данного сравнения ниже:
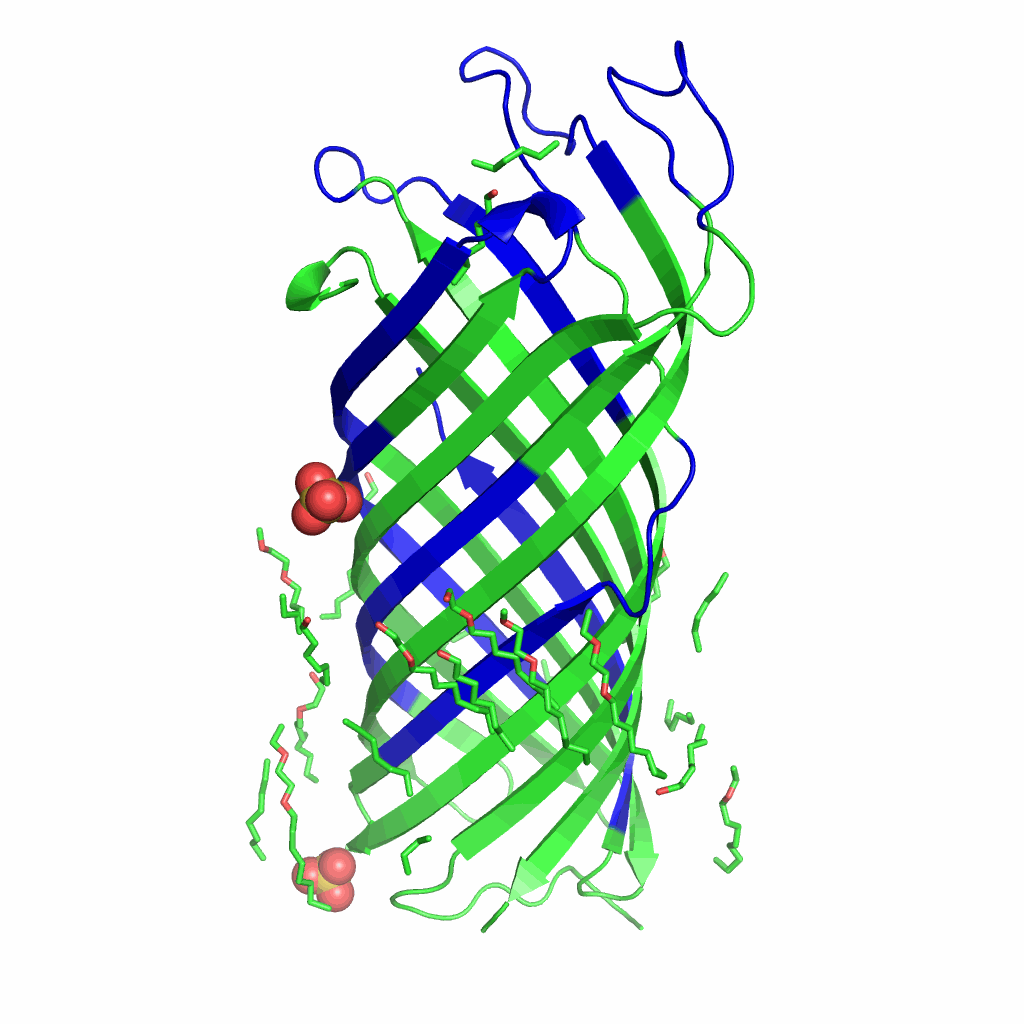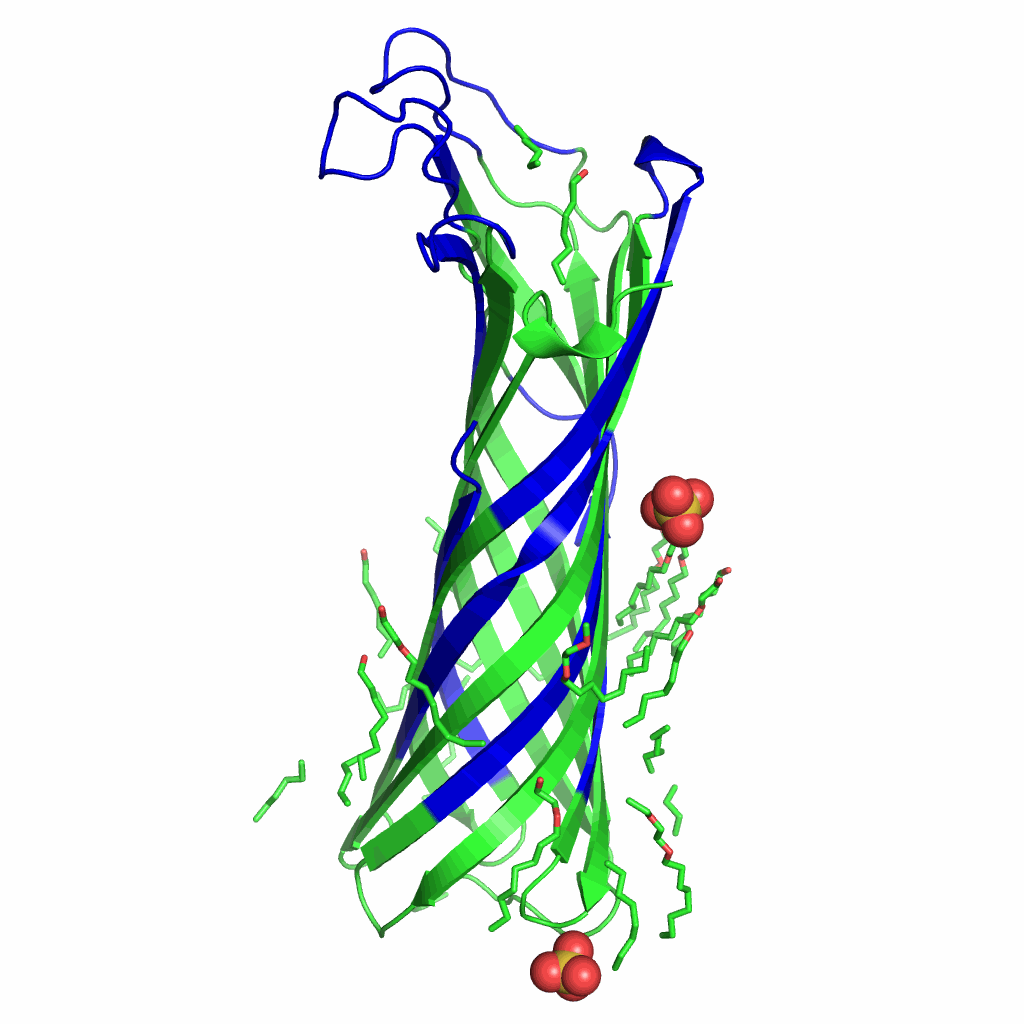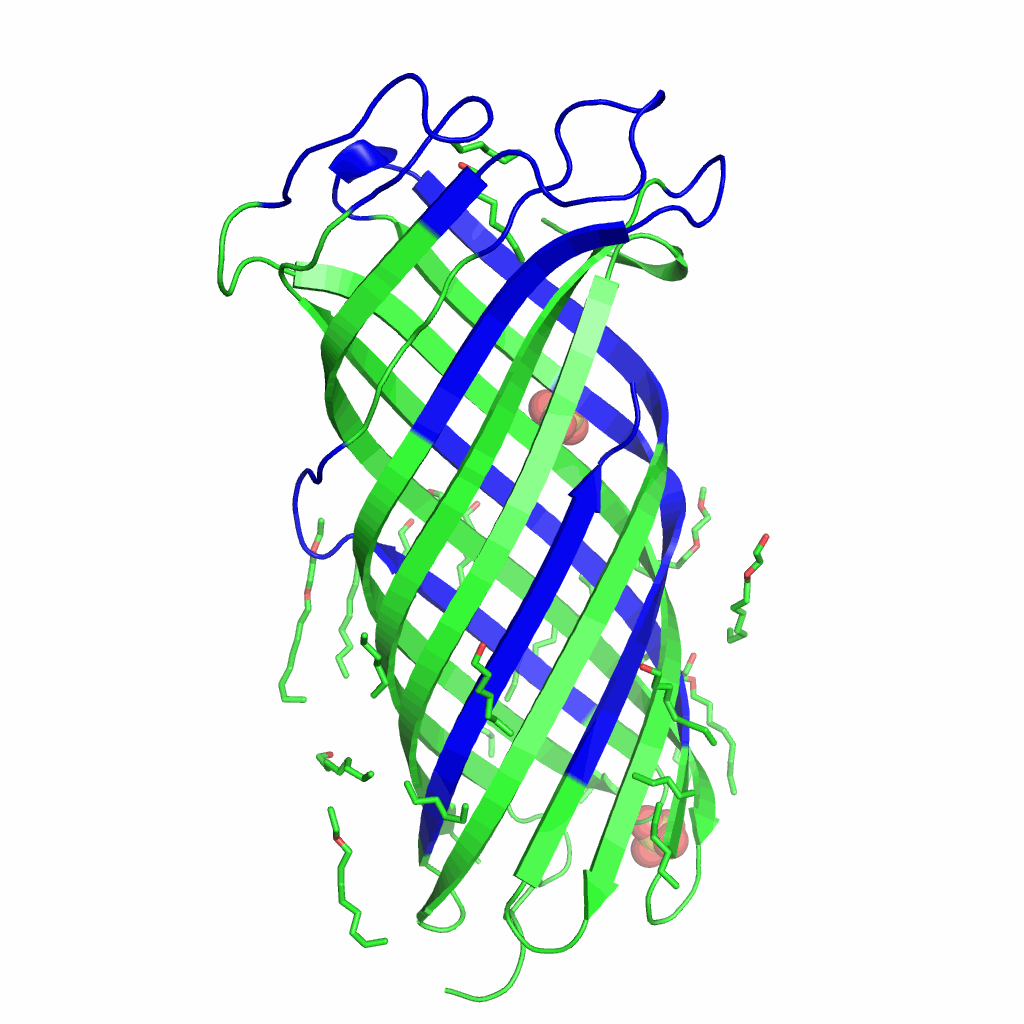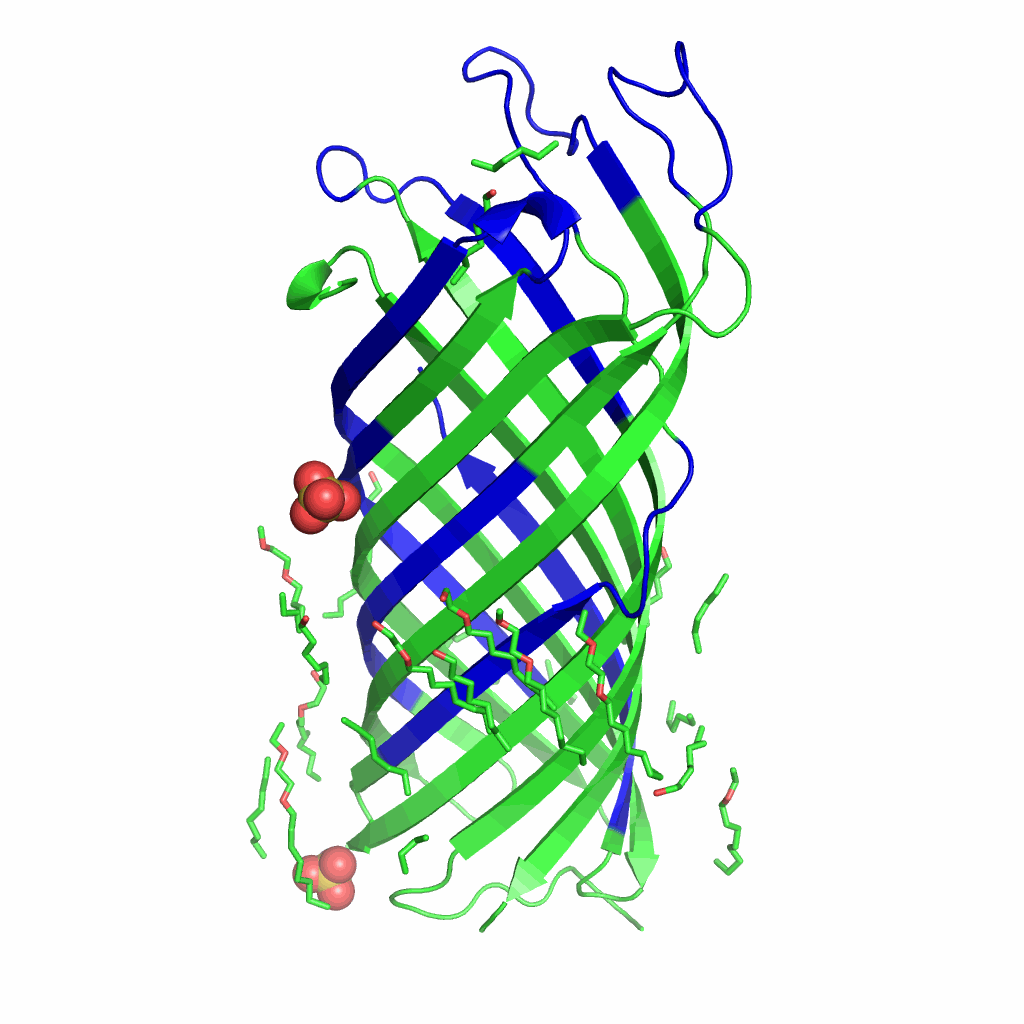
Рис.2. Визуализация предсказаний DeepTMHMM. Трансмембранные участки выделены синим цветом
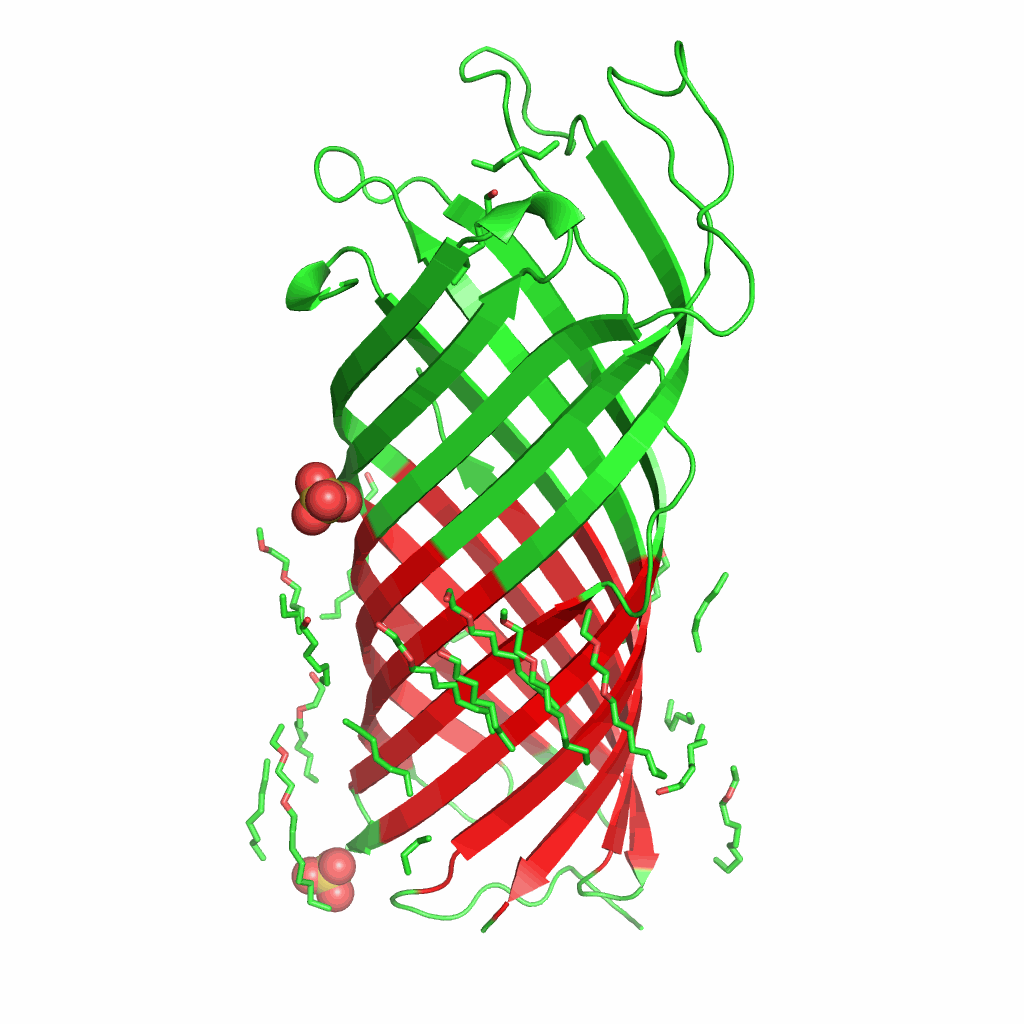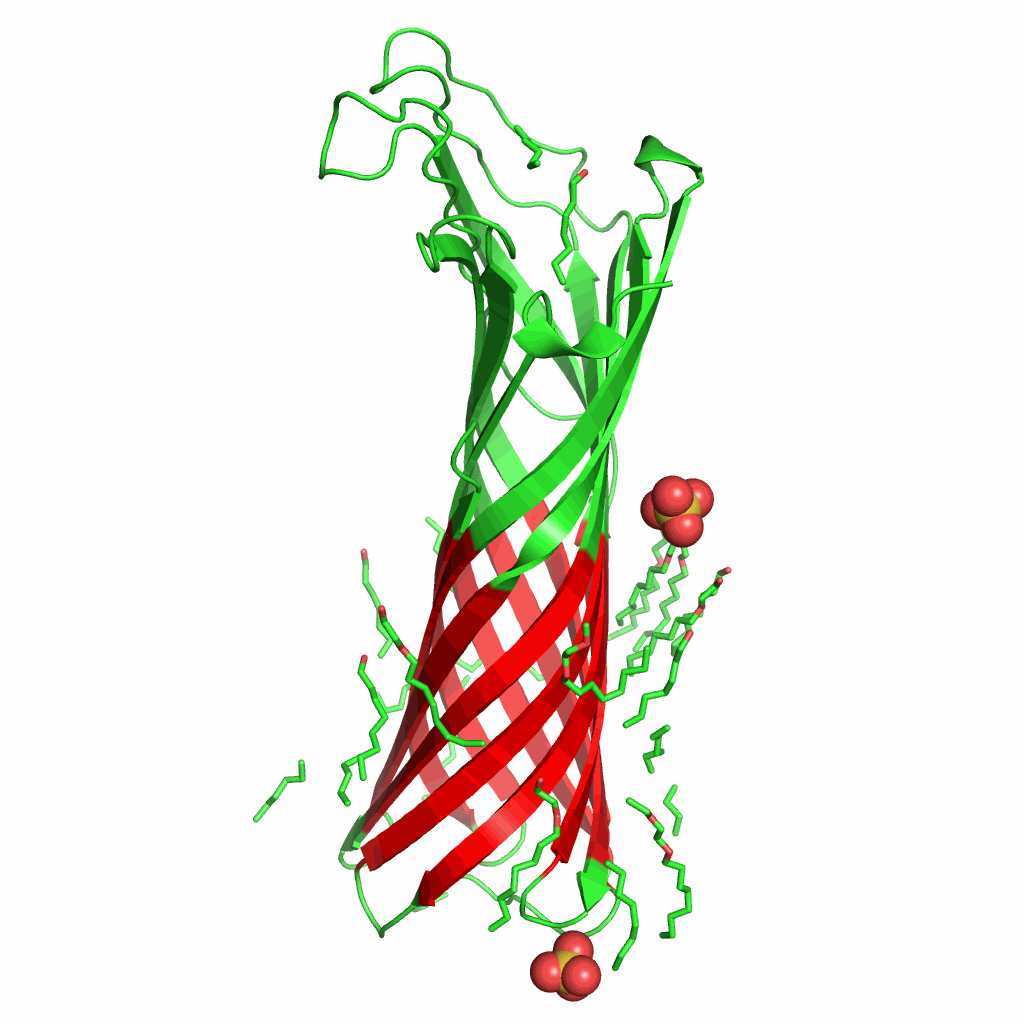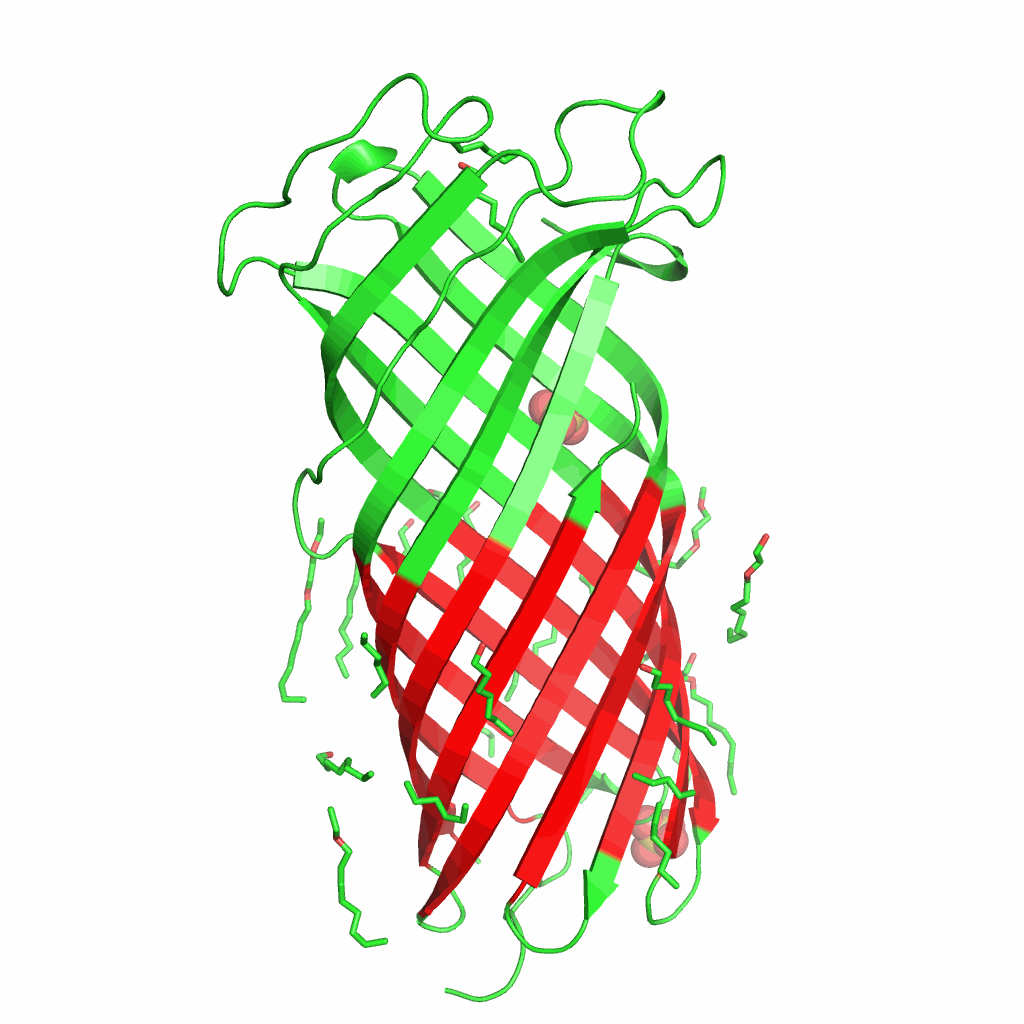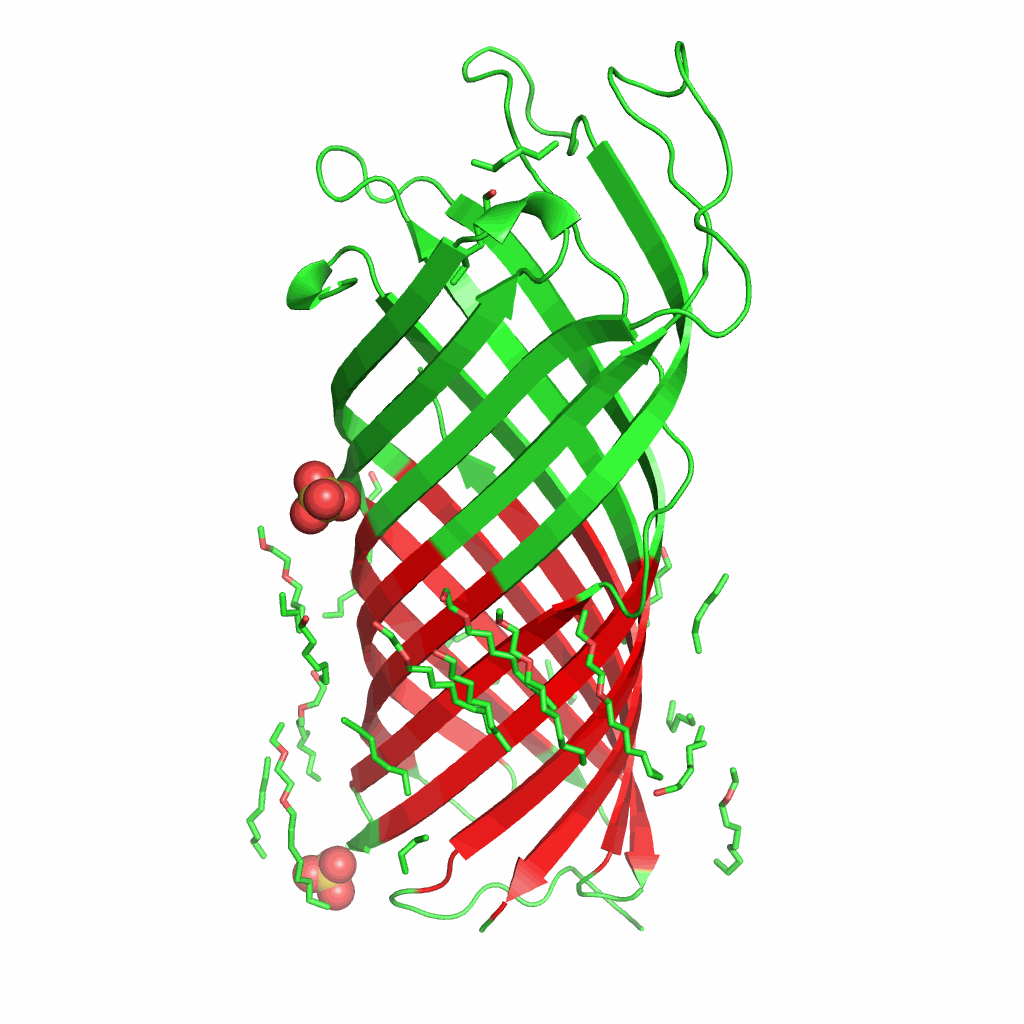
Рис.3. Визуализация предсказаний OPM. Трансмембранные участки выделены красным
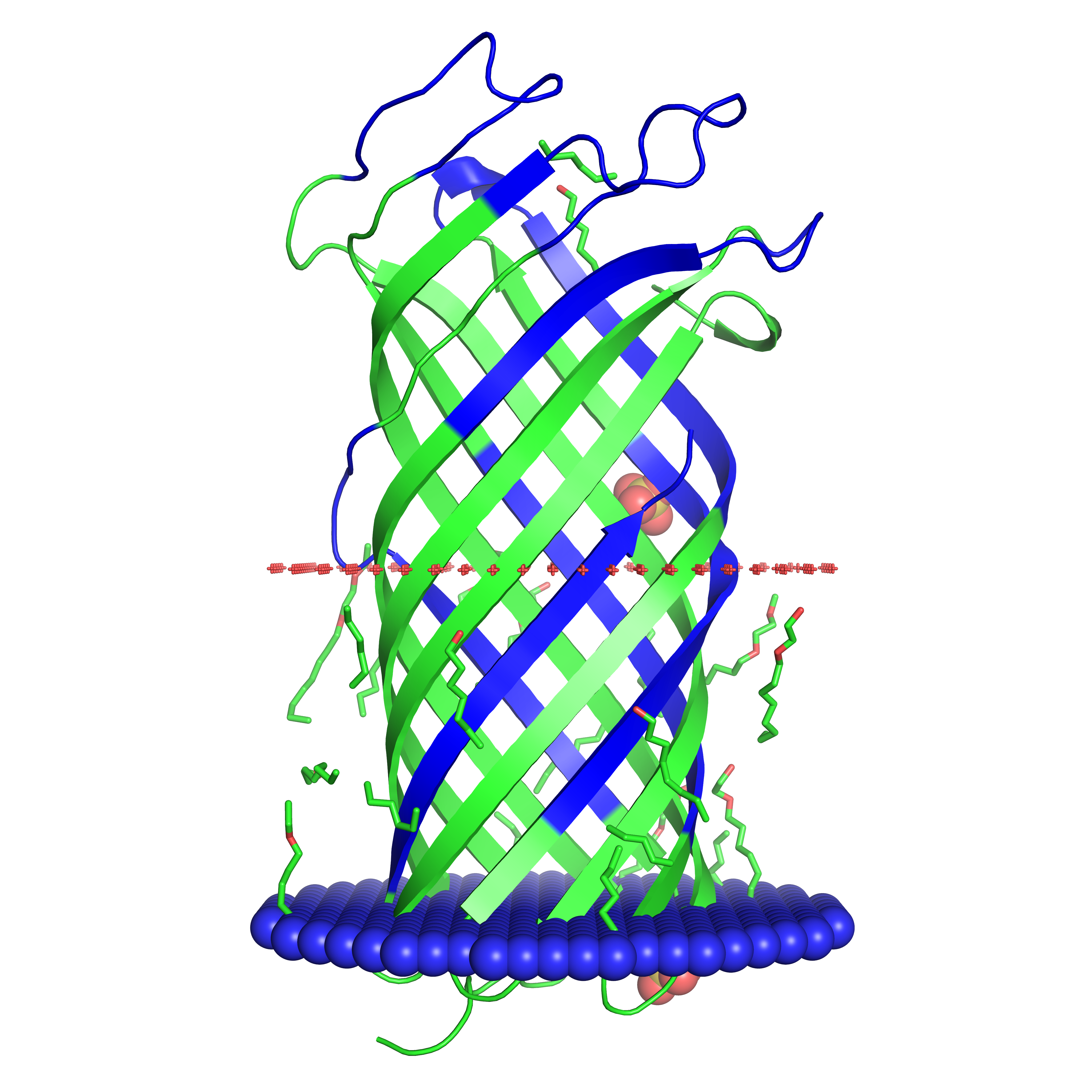
Рис.4. Объединенная версия предсказаний двух программ: OPM и DeepTMHMM. Синим цветом на белке выделены аминокислоты, определенные DeepTMHMM, как трансмембранные. Трансмембранные участки, определенные OPM не выделены, но есть внутренняя (синяя) и внешняя (красная) стороны мембраны, которые были определены именно этой программой
Таким образом, на рисунке 4 представлено совмещение того, что красиво показано на двух предыдущих gif-изображениях 2 и 3.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был выдан белок с идентификатором CTAA_AGRVS . Heme A synthase, он же гем-А-синтаза, белок, который катализирует реакцию превращения гема А в гем О, которые участвуют в реакциях восстановления кислорода. Белок локализован в Allorhizobium ampelinum.
Координаты предсказанных DeepTMHMM трансмембранных спиралей:
(28,47)
(111,130)
(139,159)
(174,192)
(211,231)
(272,292)
(305,325)
(330,350)
Данное предсказание говорит нам о том, что N-концевой участок находится внутри, а не снаружи, поэтому для PPM 3.0. укажем именно такую настройку. Кроме того, также сообщим программе, что этот белок находится во внутренней мембране грам-отрицательной бактерии, что мы узнали ранее из базы Uniprot.
Предсказанные PPM 3.0. трансмембранные участки:
1( 26- 49), 2( 105- 128), 3( 139- 159), 4( 171- 193), 5( 211- 232), 6( 269- 290), 7( 304- 325), 8( 329- 350)
Ниже представлена визуализация предсказаний сервера PPM 3.0:
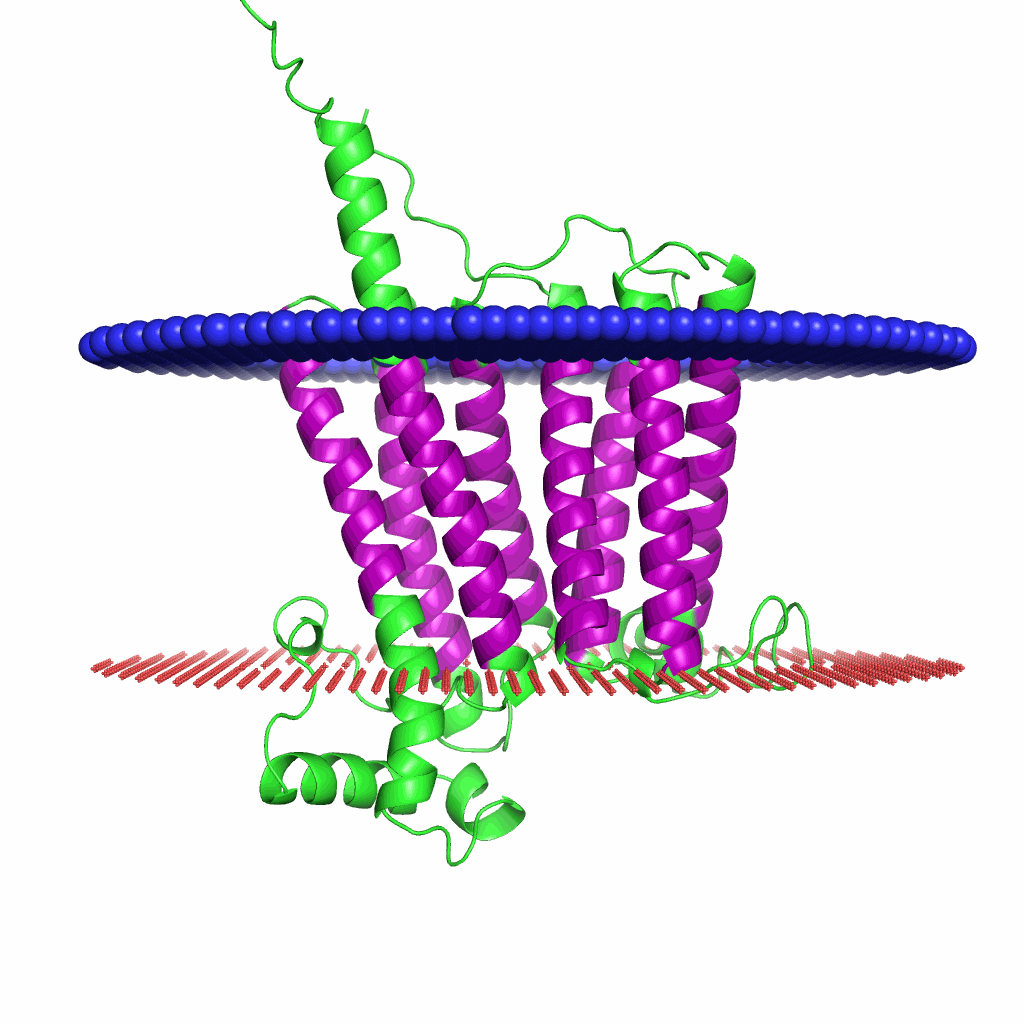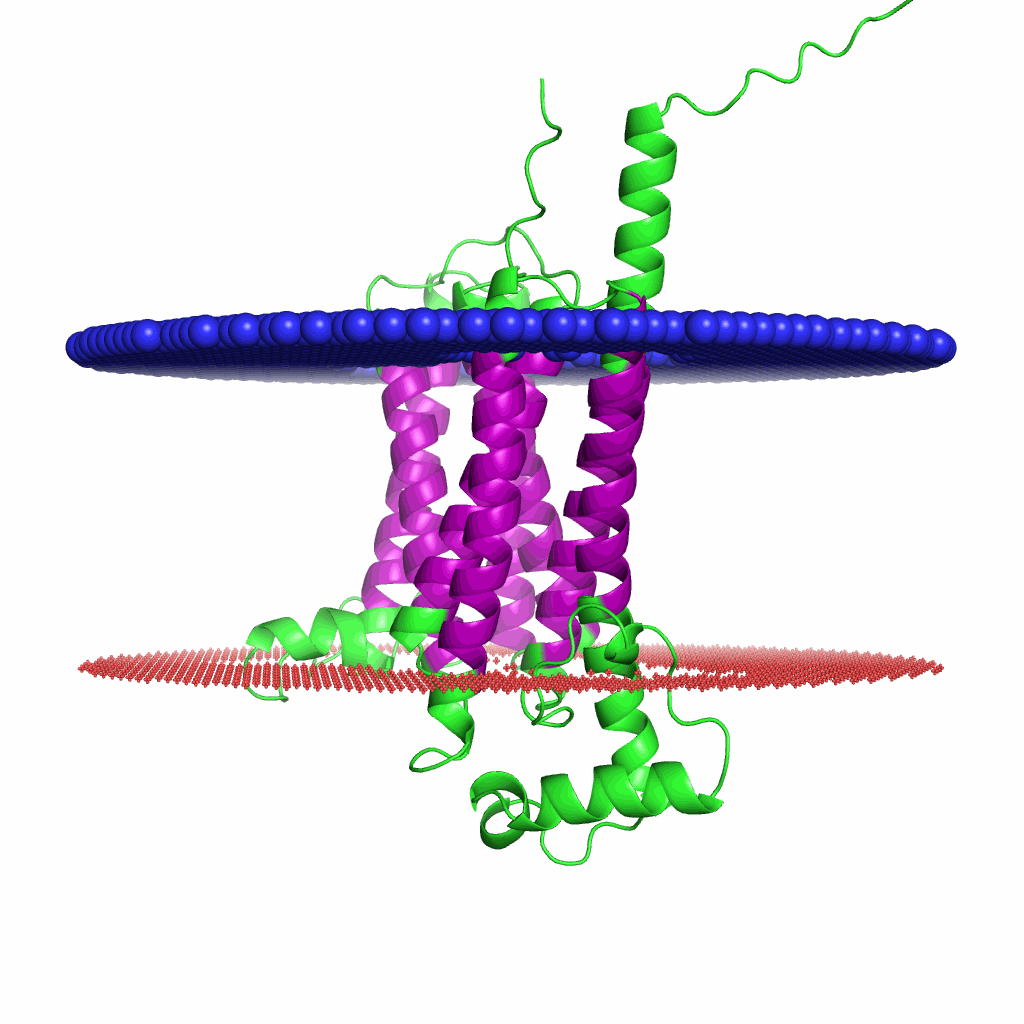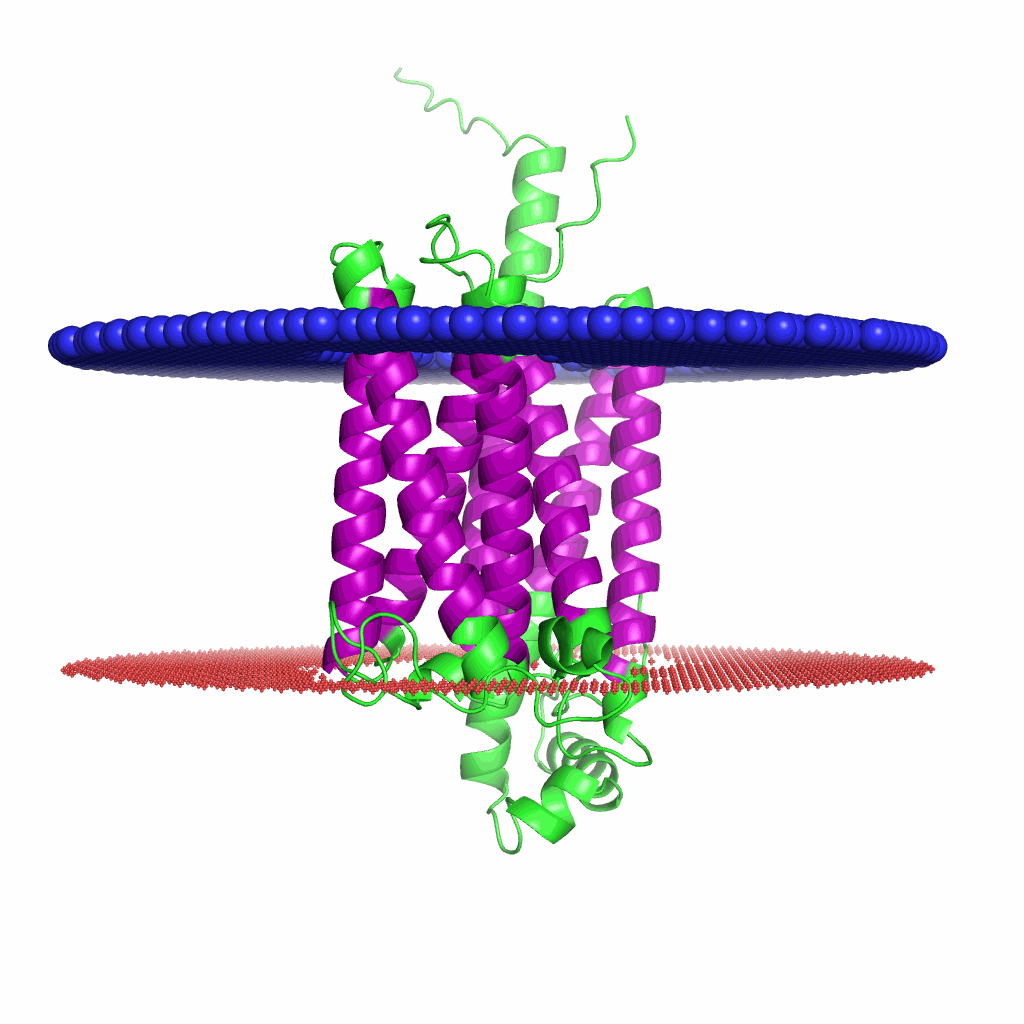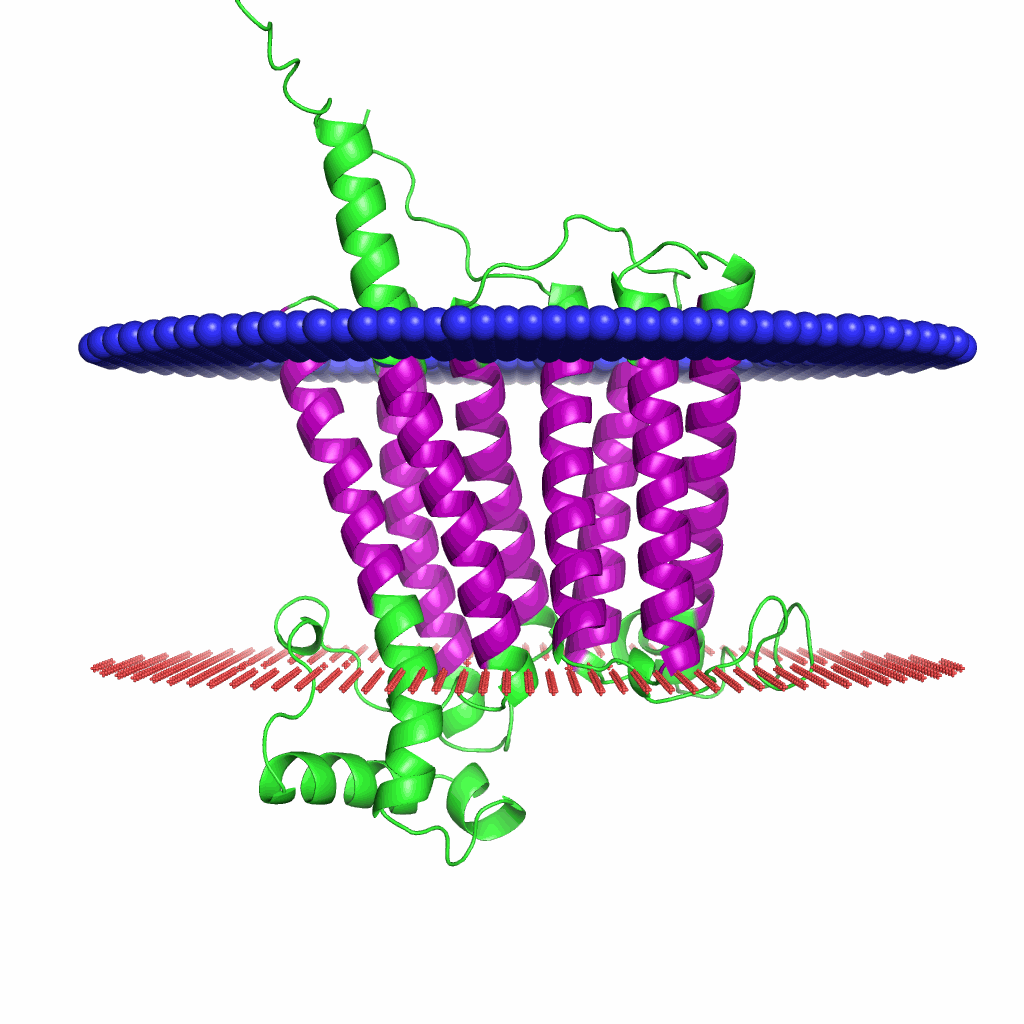
Рис.5. Полученная при помощи PPM 3.0. модель предсказанного расположения. На изображении n-сторона мембраны со стороны синего слоя, р-сторона — со стороны красного.
Если говорить про предсказания альфа-спирального белка, то они соотносятся достаточно хорошо. Возможно это связано с тем, что в альфа-спиралях заключено больше аминокислот, из-за чего предсказателю проще сработать, ведь он основан на скрытой марковской модели.
Задание 3. Дополнительное. База данных TCDB
Для начала найдем наш бета-листовой белок.
Если использовать AC, который указан на сайте Uniprot, то не получится обнаружить никакие находки, однако, воспользовавшись PDB ID, данный белок получилось найти в базе данных.
Код нашего белка 9.B.50.1.4, что соответсвует следующим данным:
9 — неполностью описанные транспортные системы
B — предполагаемые транспортные белки
50 — эндопротеазы внешней мембраны, содержащие бета-бочонки
1.4 — соответствие организму, в нашем случае это Yersinia pestis
Интересно заметить, что после изучения кросс-ссылок оказалось, что в TCDB имеется ссылка на этот белок, но она соответствует AC записи, которая была удалена из Uniprot, поэтому исходно этот белок найти не получилось.
Что касается альфа-спирального белка... с ним тоже не все так просто :)
В TCDB нет именно того белка, что был выдан, вернее, нет белка из необходимого нам организма. Поэтому по ключевым словам был найден гомолог из организма Bacillus subtilis, который описан ниже:
Код данного белка 3.D.4.4.1, где:
3 — транспортеры, использующие первичный источник энергии для активного транспорта веществ
D — транспортеры, использующие энергию окислительно-восстановительного движения электронов
4 — семейство протон-транслоцирующих оксидоредуктаз
4.1 — код организма, в нашем случае Bacillus subtilis
Также инетресно заметить, что длина гомологов отличается значимо: белок, который был выдан и рассматривался в предыдущем задании, имеет длину 364 аминокислоты, а его гомолог из Bacillus subtilis всего лишь 306 аминокислот.
На этом, пожалуй, все :)