Практикум 9
или "не ХЕ-ХЕ, а МЕМЕ"
Поиск de novo сигналов в ДНК
Введение и подготовка данных
В данной работе было решено рассмотреть уже ставший классикой геном Escherichia coli (как говорится, это база), а именно ее оперонную организацию и промоторные участки. Для этого был скачан сам геном бактерии. Затем таблица генов была обработана так, чтобы определить оперонные участки. Для этой цели был написан скрипт, ссылочка , который определяет оперонную структуру, основываясь на расстоянии между концом и началом соседних генов. Обоснование выбранных расстояний есть в статье, представленной в самом конце файла.
Другие файлы для подготовки данных:
Геном Escherichia coli: E.coli fasta
Таблица генов: genes.tsv
Оперонные участки: оперонные участки "для обучения", оперонные участки "тестовые"
МЕМЕ
На основании разбиения на опероны были отобраны участки длиной 150 нуклеотидов до начала первого гена оперона, 50 этих последовательностей затем использовались для "обучения" МЕМЕ, а для тестирования использовались вообще все такие участки (файлы представлены выше). После этого был произведен запуск программы МЕМЕ на сервере kodomo. Параметры, которые были модифицированы: мод - zoops, количество мотивов - 7, минимальная ширина мотива - 5, максимальная ширина мотива - 50, максимальный порядок марковской модели - 0, максимальное число итераций - 1000.
Такие широкие границы необходимы ввиду того, что значимые участки мотива могут распологаться с промежутком на незначимые. Также стоит сказать, что от числа итераций повышается точность нахождения самих мотивов.
По результатам работы МЕМЕ получилось следующее распределение мотивов: ссылочка
Визуальное представление:
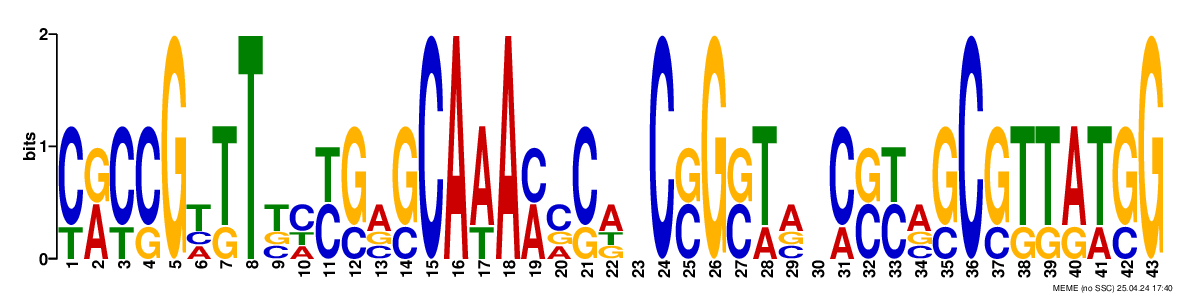

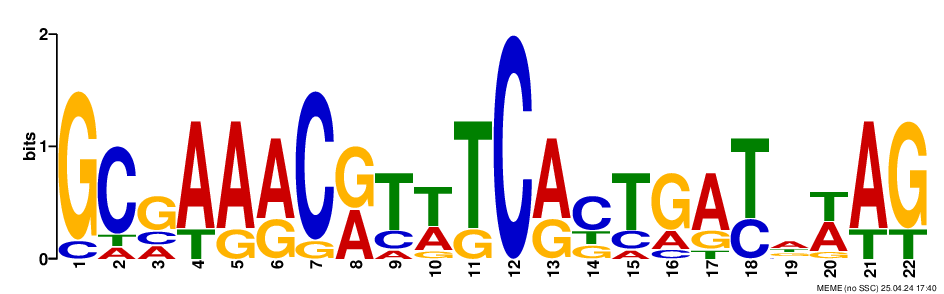

Рис.1. Мотивы 1, 2, 3, 4 с E-value 1.3e+000, 2.8e+002, 2.8e+002, 7.9e+002, соответственно



Рис.2. Мотивы 5, 6, 7 с E-value 1.2e+003, 1.4e+003, 2.1e+003, соответственно

Рис.3. Расположение мотивов в оперонных участках
Выдача МЕМЕ по итогу: MEME
Можно увидеть, что в первом мотиве наблюдаются участки комплементарные -35 и -10 боксам. Это склоняет нас к тому, чтобы выбрать именно этот мотив, несмотря на то, что у него высокий e-value, что вызвано его большой длиной и большой длиной незначимых участков в нем. Поэтому выберем именно его для поиска с помощью программы FIMO.
FIMO
Для запуска программы был использован файл этого мотива и набор тестовых последовательностей, о котором говорилось ранее. Паттерн данной последовательности - CRCCGHTTBHYGVG(CAAA)MVCDNCSGSTVNCSYV(GCGTTAT)GG, где скобками помечены последовательности боксов.
В итоге получилось 18 находок с достаточно хорошим p-value. Причем во всех из них имеются константные участки тех самых -35 и -10 боксов. Это доказывает нам, что мы нашли что-то значимое исходно, несмотря на низкий e-value.
Выдача FIMO в текстовых форматах: fimo.tsv, fimo.gff
Итоговая выдача FIMO: FIMO
Выводы
По результатам можно говорить о том, что найденный мотив был искомым и соответствует сигналу посадки рибосомы. У всех находок очень низкое значение p-value, поэтому можно считать, что все из них были достоверными. Таким образом, из всех оперонов, 18 штук имеют полностью сходные -35 и -10 боксы.
Использованная литература
Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J. Operons in Escherichia coli: genomic analyses and predictions. Proc Natl Acad Sci U S A. 2000 Jun 6;97(12):6652-7. doi: 10.1073/pnas.110147297. PMID: 10823905; PMCID: PMC18690