
1AH5 PDB
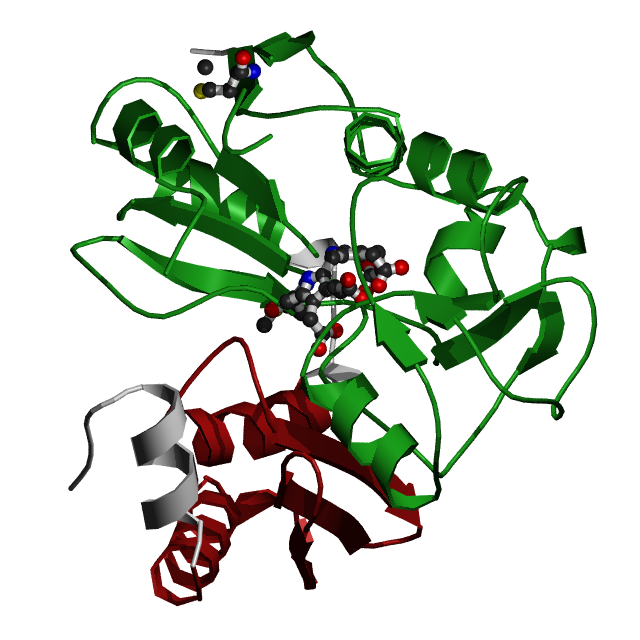
Зеленым цветом показан домен Porphobil_deam, красным-домен Porphobil_deamC, cерым -остальные части белка. Разноцветными шариками отмечены лиганды.
| Схематичное изображение доменной структуры HEM3_ECOLI. Здесь будет рассматриваться только домен Porphobil_deam |
 |
 |
1AH5 PDBЗеленым цветом показан домен Porphobil_deam, красным-домен Porphobil_deamC, cерым -остальные части белка. Разноцветными шариками отмечены лиганды. |
|
Идентификатор Pfam
|
Название записи
|
Полное название домена
|
Положение в последовательности HEM3_ECOLI
|
|
PF01379
|
Porphobil_deam
|
Порфобилиногендеаминаза,домен, связывающий кофактор дифурометан
|
5-217
|
|
PF01379
|
Porphobil_deamС
|
ПорфобилиногендеаминазаС,терминальный домен
|
225-298
|
|
Таксон
|
Количество белков с доменом PF01379
|
|
| Эукариоты | Растения | 13 |
| Грибы | 31 | |
| Животные | 34 | |
| Прокариоты | Бактерии | 507 |
| Прокариоты | Археи | 31 |

|
Porphobil_deam ,G3DSA:3.30.160.40 |

|
Porphobil_deam , PD002745 |

|
Porphobil_deam ,PF01379 |

|
Porphobil_deamC ,PF03900 |

|
4pyrrol_synth_OHMeBilane_synth ,PIRSF001438 |

|
PORPHBDMNASE , PR00151 |

|
PORPHOBILINOGEN_DEAM ,PS00533 |

|
Porphobil_deam ,PTHR11557 |

|
Porphobil_deam ,SSF54782 |

|
hemC , TIGR00212 |

|
G3DSA:3.40.190.10 , G3DSA:3.40.190.10 |

|
SSF53850 , SSF53850 |
|
Название подписи
|
Положение в последовательности HEM3_ECOLI
|
Полное название базы данных
|
* Название организации, поддерживающей данную БД
|
* Город и страна
|
|
Porphobil_deam
|
221 - 313
|
Gene3D
|
University College London
|
Манчестер, Англия
|
| Porphobil_deam | 93-206 | ProDom | INRA and CNRS - Национальный институт аграрных исследований
|
Франция, Париж |
| Porphobil_deam | 5 - 217 | Pfam | Wellcom Trust Sunger Institute
Stockholm Bionformatic Centre (SBC) Howard Hughes Medical Institute (HHMI) Institut National de la Recherche Agronomique (INRA) |
Кембридж, Великобритания
Стокгольм, Швеция Chevy Chase,США; Северная Корея Париж, Франция |
Porphobil_deamC | 225-298 | Pfam | Wellcom Trust Sunger Institute
Stockholm Bionformatic Centre (SBC) Howard Hughes Medical Institute (HHMI) Institut National de la Recherche Agronomique (INRA) |
Кембридж, Великобритания
Стокгольм, Швеция Chevy Chase,США; Северная Корея Париж, Франция |
4pyrrol_synth_OHMeBilane_synth | 4-309 | PIRSF | University of Bristol University of Texas at Austin Stanford University |
Вашингтон, США |
PORPHBDMNASE | 45 - 65,76 - 95, 124 - 141 ,143 - 160 ,230 - 247 | PRINTS | University of Manchester Bioinformatics Information and Research |
Манчестер, Англия |
PORPHOBILINOGEN_DEAM | 231-247 | PROSITE pattern | Swiss Institute of Bioinformatics |
Швейцария, Женева |
Porphobil_deam | 78-313 | PANTHER | GeneOntology |
Porphobil_deam | 220-313 | SuperFamily | University of Bristol University of Texas at Austin Stanford University |
Бристол, Англия Аустин,США,штат Техас Стенфорд, США |
hemC | 6-298 | TIGRFAMs | U.S. Department of Energy (DOE),
National Science Foundation (NSF),
The Institute for Genomic Research
|
США, Роквиль |
G3DSA:3.40.190.10 | 1 - 120 | Gene3D | University College London |
Лондон, Англия |
SSF53850 | 3-219 | SuperFamily | University of Bristol University of Texas at Austin Stanford University |
Бристол, Англия Аустин,США,штат Техас Стенфорд, США |
© Bareeva Lilia,2007