Часть I: подготовка чтений
0. Создание рабочей директории. Была создана директория /nfs/srv/databases/ngs/liliavasilyeva, в нее были скопированы файлы chr6.fastq, chr6.fasta. 1. Анализ качества чтений. Был сделан контроль качества чтений с помощью программы FastQC. Комманда:fastqc chr6.fastq2. Очистка чтений Была сделана очистка чтений с помощью программы Trimmomatic. С конца каждого чтения были удалены нуклеотиды с качеством ниже 20, оставлены только чтения длиной не меньше 50 нуклеотидов. После работы программы из 10289 ридов осталось 10123 (98,39%). Команда:java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr6.fastq chr6trimmed.fastq TRAILING:20 MINLEN:50 fastqc chr6trimmed.fastqДо чистки Выдача FastQC per base quality:
| До чистки | После чистки |
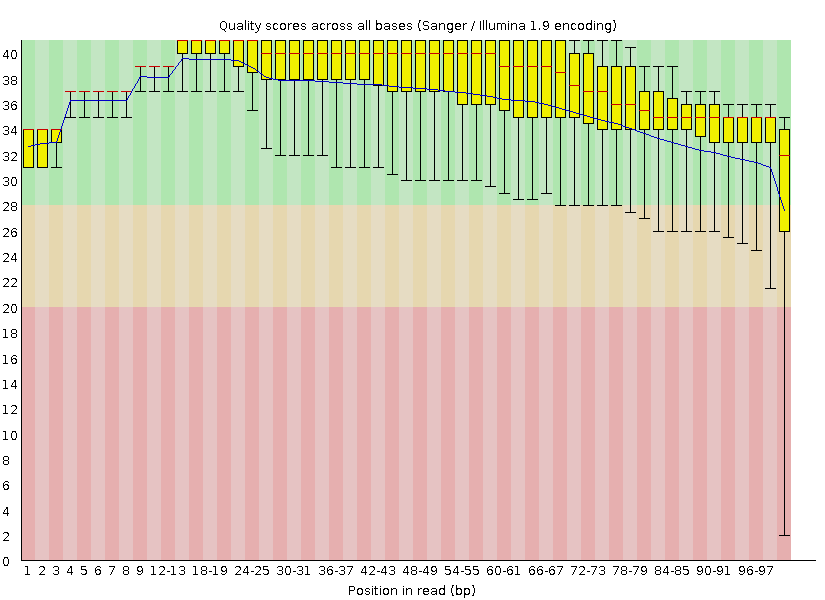 |
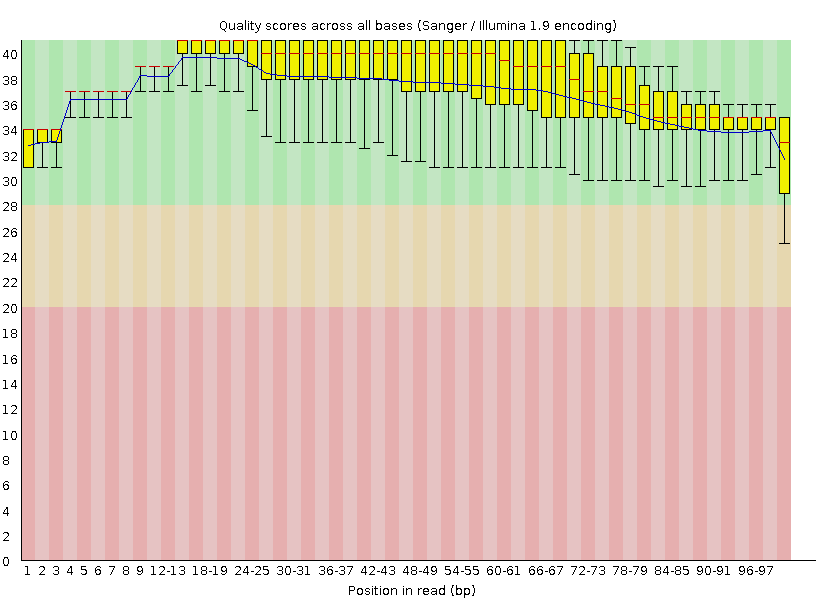 |
Часть II: картирование чтений
3. Картирование чтений. Был экспортирован пакет программ:export PATH=${PATH}:/home/students/y06/anastaisha_w/hisat2-2.0.5
Была проиндексирована референсная последовательность с помощью команды
hisat2-build chr6.fasta chr6index
Затем было построено выравнивание прочтений и референса в формате .sam. Команда:
hisat2 -x chr6index.fasta -u chr6trimmed.fastq --no-spliced-alignment --no-softclip>chr6.sam
4. Анализ выравнивания
Выравнивание было переведено в бинарный формат командой
samtools view chr6.sam -b -o align.bam
Выравнивание чтений с референсом было отсортировано по координате в референсе начала чтения. Команда:
samtools sort align.bam -T align.txt -o alignsorted.bam
Отсортированный .bam файл был проиндексирован командой
samtools index alignsorted.bam
10045 чтений было откартировано. В выдаче Hisat2 также можно посмотреть количество некартированных(78, то есть 0.77%) и
неспаренных чтений, чтений, картированных более одного раза.
В выходном файле прописаны: имя рида, сумма числовых обозначений тэгов(рид один из пары, у рида нет выравниваний и т. д.),
название референсной последовательности, координаты начала выравнивания рида на референсной последовательности, качество
картирования и т. д.
Часть III: Анализ SNP
5. Поиск SNP и инделей. С помощью командыsamtools mpileup -uf chr6.fasta alignsorted.bam -o align.bcf
был создан файл с полиморфизмами в формате .bcf.
С помощью команды bcftools call -cv align.bcf -o align.vcf
был создан файл со списком отличий между референсом и чтениями в формате .vcf.
| Координата | Тип полиморфизма | В референсе | В чтениях | Глубина покрытия | Качество чтений | |
| 1. | 106961119 | Замена | T | C | 6 | 36.0297 |
| 2. | 107016838 | Вставка | CTTT | CTTTTT | 36 | 217.468 |
| 3. | 154437252 | Замена | A | G | 1 | 11.3429 |
perl /nfs/srv/databases/annovar/convert2annovar.pl -format vcf4 align.vcf > align.avinput
Выдача: A total of 83 locus in VCF file passed QC threshold, representing 78 SNPs (49 transitions and 29 transversions) and 6 indels/substitutions
NOTICE: Finished writing 78 SNP genotypes (49 transitions and 29 transversions) and 5 indels/substitutions for 1 sample
- то есть, 78 SNP и 5 инделей.
Сначала все 5 инделей были удалены вручную.
Затем были использованы команды:
perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.1000g -buildver hg19 -dbtype 1000g2014oct_all align.avinput /nfs/srv/databases/annovar/humandb/
perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.snp -build hg19 -dbtype snp138 align.avinput /nfs/srv/databases/annovar/humandb/
perl /nfs/srv/databases/annovar/annotate_variation.pl -out rs.refgene -build hg19 align.avinput /nfs/srv/databases/annovar/humandb/
perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.clinvar -build hg19 -dbtype clinvar_20150629 align.avinput /nfs/srv/databases/annovar/humandb/
perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out rs.gwas -build hg19 -dbtype gwasCatalog align.avinput /nfs/srv/databases/annovar/humandb/
и получены файлы с информацией из соответствующих баз данных.
В базе RefGene 14 SNP принадлежит к экзонам, 60 к интронам, 4 к UTR3.
Названия генов с полиморфизмами указаны во второй колонке RefGene таблицы.
6 полиморфизмов не имеют Rs(DBSNP).
Максимальную частоту встречаемости имеет полиморфизм с координатой 107008422(98.78%), минимальную - 154382572(0.66%)(1000 genomes).
Клиническую аннотацию имеет только один(138196066) из данных полиморфизмов(Clinvar).
Остальные данные представлены в таблице:
| Координата | Референс | Чтение | Качество | Глубина покрытия | Refgene | 1000g | DBSNP | Clinvar | GWAS | ||
| 106961119 | T | C | 36.0297 | 6 | intronic | AIM1 | het | 0.0253594 | rs73519199 | ||
| 106966701 | A | G | 65.5132 | 4 | intronic | AIM1 | hom | 0.925319 | rs783414 | ||
| 106966801 | T | C | 137.008 | 25 | intronic | AIM1 | het | 0.207069 | rs13194986 | ||
| 106967185 | A | C | 177.009 | 38 | exonic | AIM1 | het | 0.20647 | rs1159148 | ||
| 106967778 | T | C | 183.009 | 26 | exonic | AIM1 | het | 0.174121 | rs3747787 | ||
| 106967833 | G | A | 191.009 | 25 | exonic | AIM1 | het | 0.0181709 | rs61734885 | ||
| 106968091 | C | T | 114.008 | 19 | exonic | AIM1 | het | 0.0177716 | rs61740946 | ||
| 106968369 | G | A | 218.009 | 41 | exonic | AIM1 | het | 0.0177716 | rs73761859 | ||
| 106974933 | T | G | 81.013 | 5 | intronic | AIM1 | het | 0.168331 | rs9486379 | ||
| 106975415 | A | T | 225.009 | 46 | intronic | AIM1 | het | 0.498403 | rs2219666 | ||
| 106976802 | G | T | 3.01618 | 170 | intronic | AIM1 | het | - | - | ||
| 106978314 | G | A | 183.009 | 1 | intronic | AIM1 | het | 0.210863 | rs2306099 | ||
| 106980118 | G | C | 10.4247 | 1 | intronic | AIM1 | hom | 0.512979 | rs1321250 | ||
| 106982532 | G | T | 11.3429 | 1 | intronic | AIM1 | hom | 0.391773 | rs898895 | ||
| 106982560 | G | A | 9.52546 | 1 | intronic | AIM1 | hom | 0.912141 | rs1111830 | ||
| 106982642 | G | A | 5.46383 | 1 | intronic | AIM1 | hom | 0.389177 | rs7454837 | ||
| 106982672 | T | C | 6.20226 | 1 | intronic | AIM1 | hom | 0.386382 | rs898894 | ||
| 106982931 | G | T | 7.79993 | 1 | intronic | AIM1 | hom | 0.264377 | rs9480681 | ||
| 106987019 | T | C | 65.9724 | 4 | intronic | AIM1 | hom | 0.666933 | rs783398 | ||
| 106987161 | A | G | 221.999 | 57 | intronic | AIM1 | hom | 0.666933 | rs783397 | ||
| 106987370 | A | C | 221.999 | 88 | exonic | AIM1 | hom | 0.935903 | rs783396 | ||
| 106989212 | G | A | 221.999 | 36 | intronic | AIM1 | hom | 0.640375 | rs783392 | ||
| 106991195 | G | A | 180.999 | 25 | intronic | AIM1 | hom | 0.123802 | rs7851278 | ||
| 106992464 | A | G | 221.999 | 58 | exonic | AIM1 | hom | 0.96885 | rs1799693 | ||
| 106992592 | A | T | 221.999 | 77 | intronic | AIM1 | hom | 0.833466 | rs2247335 | ||
| 106992844 | C | G | 221.999 | 25 | intronic | AIM1 | hom | 0.833666 | rs2066201 | ||
| 106998973 | T | C | 4.13164 | 1 | intronic | AIM1 | het | 0.787939 | rs4946762 | ||
| 106999658 | A | T | 221.999 | 36 | intronic | AIM1 | hom | 0.879193 | rs1676010 | ||
| 107001456 | A | T | 169.009 | 25 | intronic | AIM1 | het | 0.829273 | rs1770731 | ||
| 107001581 | T | C | 113.008 | 9 | intronic | AIM1 | het | 0.793131 | rs1770732 | ||
| 107003444 | G | C | 81.0075 | 10 | intronic | AIM1 | het | 0.755391 | rs2297971 | ||
| 107003810 | T | C | 225.009 | 65 | intronic | AIM1 | het | 0.695088 | rs2066202 | ||
| 107006336 | G | C | 225.009 | 51 | intronic | AIM1 | het | 0.167931 | rs9320179 | ||
| 107008422 | C | T | 101.264 | 5 | intronic | AIM1 | hom | 0.987819 | rs968838 | ||
| 107008430 | C | T | 116.133 | 6 | intronic | AIM1 | hom | 0.938299 | rs968839 | ||
| 107009090 | G | A | 94.0077 | 9 | intronic | AIM1 | het | 0.744609 | rs965347 | ||
| 107009119 | A | G | 114.008 | 11 | intronic | AIM1 | het | 0.744609 | rs4946764 | ||
| 107011930 | T | C | 176.001 | 12 | intronic | AIM1 | hom | 0.970847 | rs9400027 | ||
| 107014734 | A | G | 11.3429 | 1 | intronic | AIM1 | hom | 0.192292 | rs9486403 | ||
| 107016127 | G | C | 145.008 | 28 | intronic | AIM1 | het | 0.250399 | rs9480687 | ||
| 107016135 | A | G | 159.009 | 33 | intronic | AIM1 | het | 0.251198 | rs9486405 | ||
| 107016136 | T | G | 157.009 | 33 | intronic | AIM1 | het | 0.250599 | rs9486406 | ||
| 107016731 | T | C | 225.009 | 70 | UTR3 | AIM1 (NM_001624:c.*290T>C) | het | 0.226637 | rs3747790 | ||
| 108192607 | G | T | 28.0137 | 29 | UTR3 | SEC63 (NM_007214:c.*301C>A) | het | - | - | ||
| 138192745 | G | A | 15.1417 | 4 | intronic | TNFAIP3 | het | 0.201278 | rs719149 | ||
| 138192761 | A | G | 5.4626 | 2 | intronic | TNFAIP3 | het | 0.205671 | rs719150 | ||
| 138195693 | T | C | 41.7648 | 2 | intronic | TNFAIP3 | hom | 0.742612 | rs643177 | ||
| 138195723 | C | G | 65.0073 | 9 | intronic | TNFAIP3 | het | 0.139577 | rs5029939 | ||
| 138196066 | T | G | 225.009 | 46 | exonic | TNFAIP3 | het | 0.139577 | rs2230926 | clinvar_20150629 CLINSIG=untested; CLNDBN= not_specified; CLNREVSTAT= no_assertion_provided; CLNACC= RCV000122149.1; CLNDSDB= MedGen; CLNDSDBID= CN169374 | |
| 138197331 | A | C | 111.008 | 27 | intronic | TNFAIP3 | het | 0.533546 | rs661561 | ||
| 138197824 | C | T | 5.46092 | 2 | intronic | TNFAIP3 | het | 0.741613 | rs582757 | ||
| 154344284 | C | T | 9.52546 | 1 | intronic | OPRM1 | hom | 0.10623 | rs12208137 | ||
| 154357986 | T | G | 11.3429 | 1 | intronic | OPRM1 | hom | 0.137979 | rs12210856 | ||
| 154357987 | A | C | 11.3429 | 1 | intronic | OPRM1 | hom | 0.137979 | rs12190259 | ||
| 154360569 | C | T | 165.009 | 23 | exonic | OPRM1 | het | 0.0325479 | rs17174638 | ||
| 154360696 | C | T | 221.999 | 21 | exonic | OPRM1 | hom | 0.0712859 | rs1799972 | ||
| 154382542 | A | G | 7.79993 | 1 | intronic | OPRM1 | hom | 0.29353 | rs495491 | ||
| 154382572 | G | T | 5.46137 | 2 | intronic | OPRM1 | het | 0.00658946 | rs75213204 | ||
| 154387317 | G | T | 39.765 | 2 | intronic | OPRM1 | hom | - | - | ||
| 154392675 | T | C | 11.3429 | 1 | intronic | OPRM1 | hom | 0.122804 | rs9478503 | ||
| 154402589 | C | T | 11.3429 | 1 | intronic | OPRM1 | hom | 0.014377 | rs141266915 | ||
| 154406316 | A | G | 10.4247 | 1 | intronic | OPRM1 | hom | 0.0147764 | rs73790422 | ||
| 154411847 | C | T | 50.0106 | 4 | intronic | OPRM1 | het | 0.014976 | rs17181213 | ||
| 154412385 | G | A | 225.009 | 59 | exonic | OPRM1 | het | 0.0091853 | rs1799975 | ||
| 154414446 | A | T | 221.999 | 85 | exonic | OPRM1 | hom | 0.882188 | rs540825 | ||
| 154414563 | A | G | 221.999 | 84 | exonic | OPRM1 | hom | 0.778754 | rs675026 | ||
| 154417209 | G | T | 6.20226 | 1 | intronic | OPRM1 | hom | 0.763978 | rs632457 | ||
| 154422744 | G | A | 7.79993 | 1 | intronic | OPRM1 | hom | 0.871006 | rs647192 | ||
| 154423714 | C | G | 6.20226 | 1 | intronic | OPRM1 | hom | - | - | ||
| 154426153 | T | C | 6.20226 | 1 | intronic | OPRM1 | hom | - | - | ||
| 154428537 | G | A | 73.0074 | 15 | intronic | OPRM1 | het | 0.152157 | rs650825 | ||
| 154428666 | C | T | 225.009 | 26 | exonic | OPRM1 | het | 0.14996 | rs677830 | ||
| 154428702 | A | G | 221.999 | 24 | UTR3 | OPRM1 (NM_001145286:c.*4A>G) | hom | 0.88778 | rs650245 | ||
| 154431393 | C | G | 221.999 | 20 | intronic | OPRM1 | hom | 0.941094 | rs497332 | ||
| 154431565 | G | A | 218.009 | 27 | UTR3 | OPRM1 (NM_001145282:c.*19G>A) | het | 0.150359 | rs606545 | ||
| 154431742 | G | T | 9.52546 | 1 | intronic | OPRM1 | hom | - | - | ||
| 154436236 | T | C | 10.4247 | 1 | intronic | OPRM1 | hom | 0.908946 | rs617648 | ||
| 154437252 | A | G | 11.3429 | 1 | intronic | OPRM1 | hom | 0.909145 | rs632395 |