Мотивы в белках
Консервативные мотивы в выравнивании
Для работы было выбрано семейство факторов рибозилирования АДФ (PF00025). Выравнивание seed для этого семейства содержит 20 последовательностей. В выравнивании на уровне 100% идентичности есть отдельные консервативные колонки и несколько мотивов. Для работы был взят второй мотив (см рис. 1).
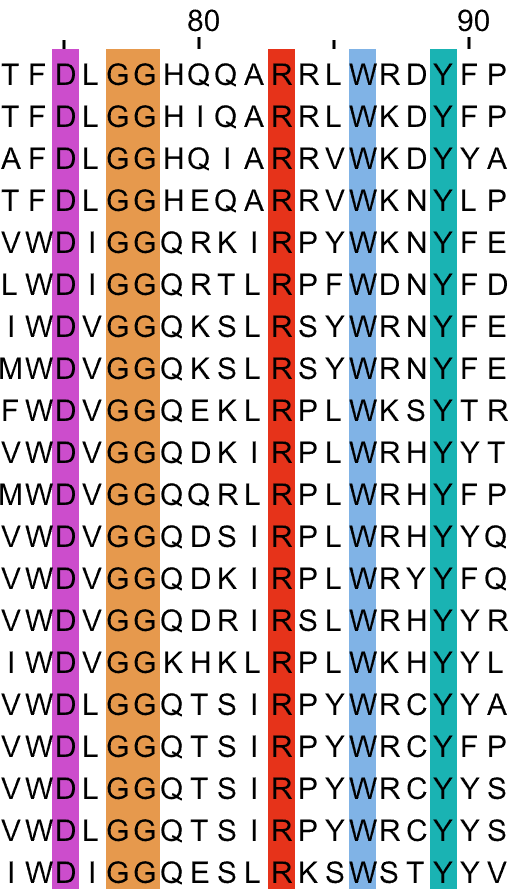
Для описания мотива был составлен следующий паттерн: D.GG.{4}R.{2}W.{2}Y или D-x-G-G-x(4)-R-X(2)-W-x(2)-Y
С помощью функции Find в Jalview я нашла данный паттерн во всех последовательностях по одному разу. Данный паттерн имеет высокое значение IC.
Далее я выполнила поиск по этому мотиву в базе данных SwissProt на сайте MyHints. Было найдено 206 находок. Большинство белков является белками семейства PF00025, кроме белков Guanine nucleotide-binding protein alpha, ubiquitin-protein ligase и их гомологов. Поэтому можно сказать сказать, что данный паттерн хорошо описывает белки из выбранного семейства.
Поиск мотива, специфичного для клады филогенетичсекого дерева
На основе выравнивания было построено дерево с помощью алгоритма neighbor-joining, представленное на рисунке 2.
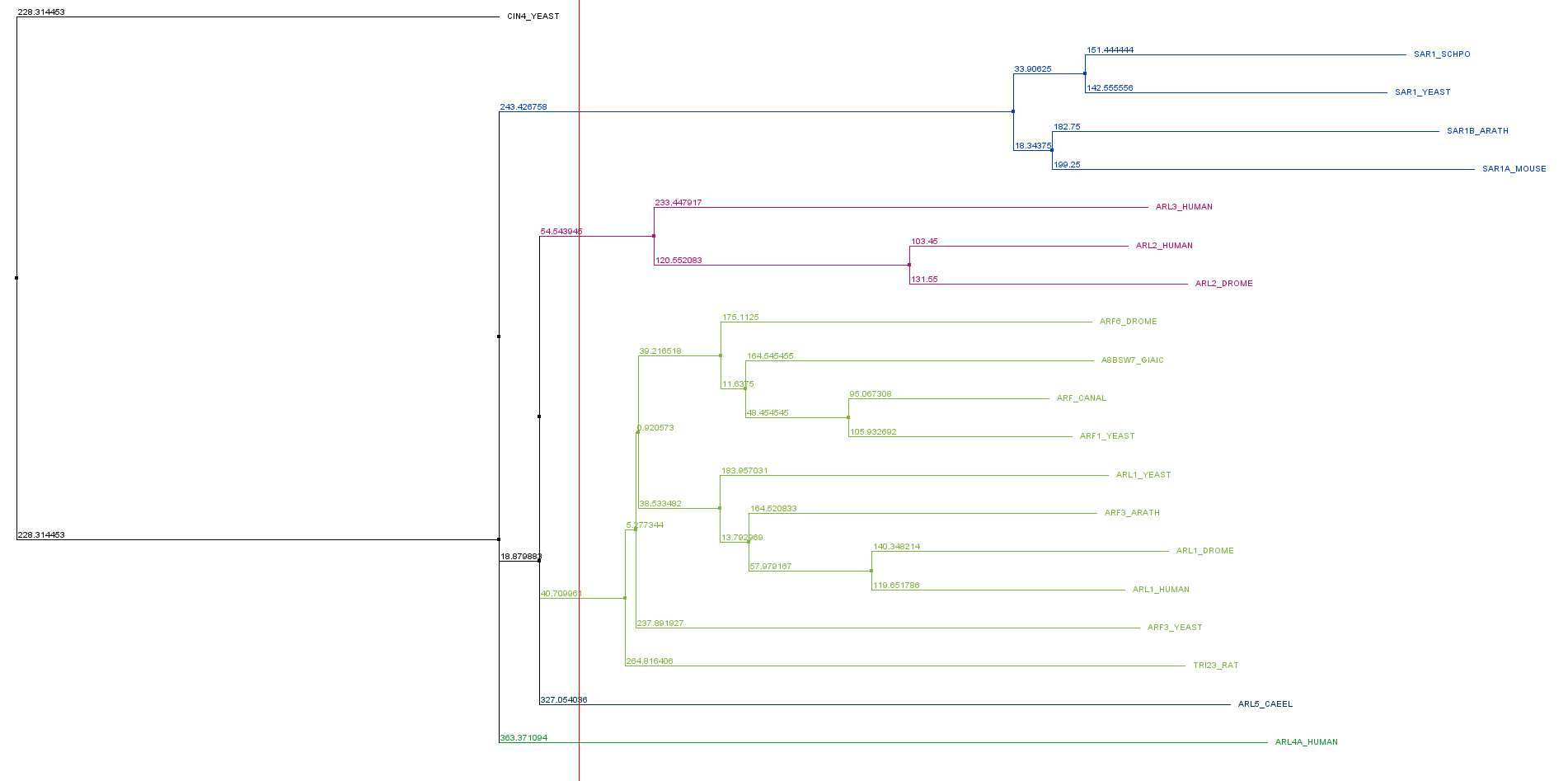
Было выделено 6 групп, три из них тривиальные. Далее я выбрала синию группу из 4 последовательностей, состоящую из SAR1 белков. В этом выравнивание был найден мотив: PT.HPTSEEL.I
Этот мотив встречается по одному разу во всех 4 последовательностях данной клады. Это говорит о том, что мотив специфичен для данной клады.
PSI-BLAST
Был выбран белок P0AD49. Во время стационарной фазы роста этот белок предотвращает образование рибосомы 70S, вероятно, для того, чтобы регулировать эффективность трансляции во время перехода между экспоненциальной и стационарной фазами роста. Таблица итераций PSI-BLAST представлена ниже.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 22 | Q49VV1.1 | 8,00E-04 | P19954.2 | 0,023 |
| 2 | 27 | O05886.4 | 3,00E-09 | NaN | NaN |
| 3 | 27 | O05886.4 | 2,00E-24 | NaN | NaN |
Как видно из таблицы, после второй итерации перестало меняться число находок, удовлетворяющее порогу e-value в 0,005. Разница e-value между худшей надпороговой находки и лучшей подпороговой составляет примерно 1 порядок, что говорит о плохой обособленности надпороговых находок и малой вероятности составления из них отдельного семейства гомологичных белков.