ПОИСК ДОМЕНА И СОЗДАНИЕ HMM-ПРОФИЛЯ
Я выбрала домен 7 transmembrane sweet-taste receptor of 3 GCPR PF00003 из 7 трансмембранных областей, которые формируют C-конец некоторых рецепторов подкласса 3 рецепторов G-связанных белков. Семь трансмембранных областей собираются таким образом, чтобы образовать стыковочный карман, в котором, как было обнаружено, связываются такие молекулы, как цикламат и лактизол, и, следовательно, придают вкусу сладость.
Выравненные 114 белков с доменной архитектурой PF00003-PF00003
Затем я почистила последовательности: Jalview => Edit => Remove redundancy порог 95% и убрала несколько последовательностей с большими вставками.
Выравненные оставшихся 20 последовательностей - обучающая выборка
Тут можно посмотреть файл со 114 белками с доменной архитектурой PF00003 - PF00003 (обучающая и положительная выборка) и со 103 белками с доменной архитектурой PF00003 - PF18455 (негативный контроль) Тык
Создание HMM-профиля
hmm2build -g out.txt cleaved.fa
Калиборовка HMM-профиля
hmm2calibrate out.txt
Поиск по общей выборке
hmm2search --cpu 1 out.txt full.fa > search.txt
Выдачи:
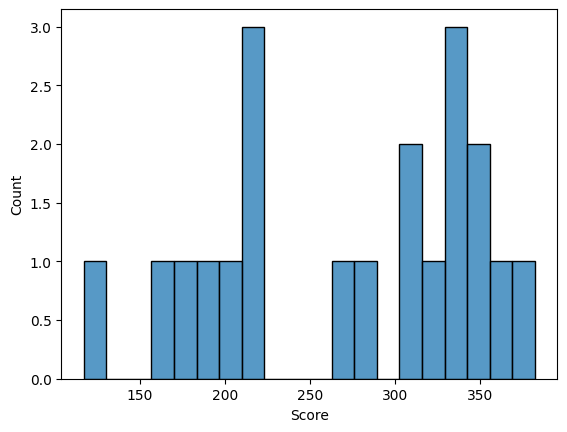
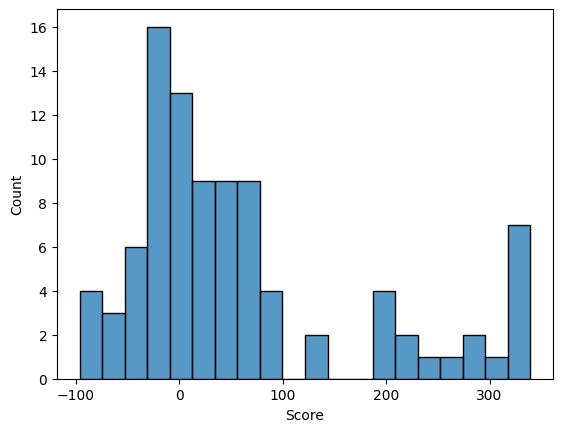
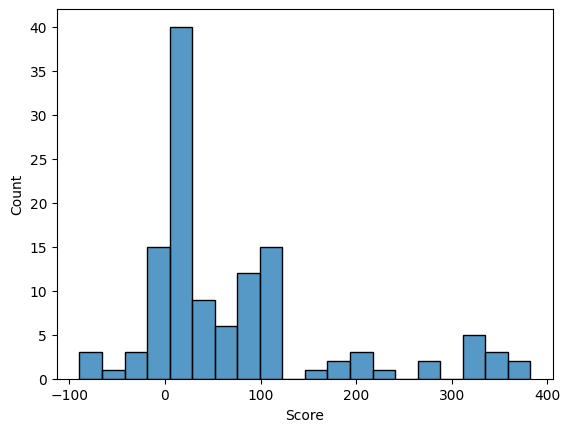
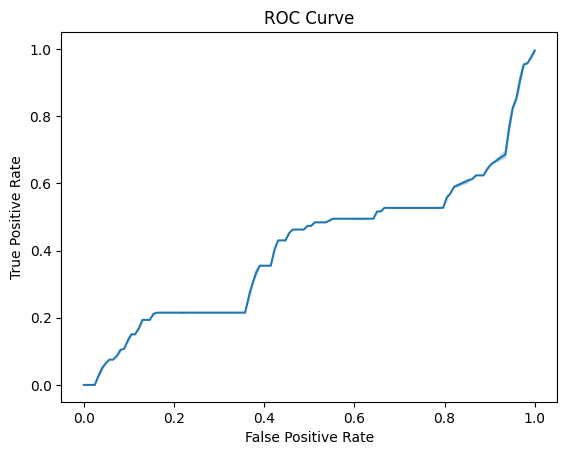
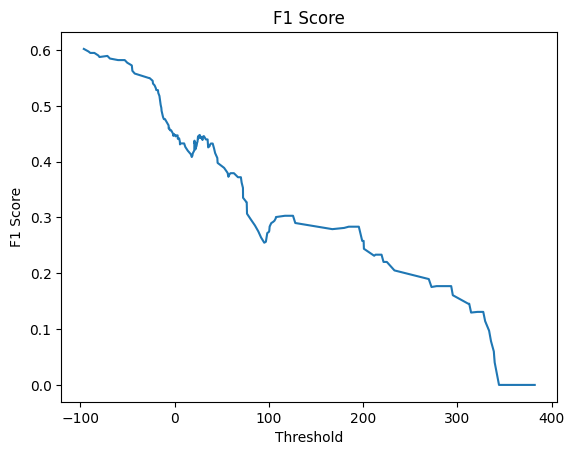
Затем с помощью скрипта Карины Каримовой были получены таблица, гистограммы весов выборок и графики ROC Curve и F1 Score. Видно, что положительная и негативная выборки плохо разделяются, скорее всего потому что выбранные мною доменные архитектуры слишком похожи.