1. Составление списка гомологичных белков, включающих паралоги.
С помощью команды я получила из списка fasta-файлов один:
cat THIDA.fasta SHEDO.fasta RHIME.fasta POLAQ.fasta ECOLI.fasta BRUSU.fasta AROAE.fasta>all1.fasta
Далее была создана база данных:
makeblastdb -in all1.fasta -dbtype prot -out ddbb1
Потом я искала гомологи белка CLPX_ECOLI, где P0A6H1.fasta это файл с последовательностью белка CLPX_ECOLI.
blastp -query P0A6H1.fasta -db ddbb1 -evalue 0.001 -out clpx_out1.txt
У меня получилось 21 находка, файл можно посмотреть по ссылке:
2. Реконструкция и визуализация.
Fasta-файл с последовательностями можно посмотреть здесь:
Далее было построено дерево, с помощью программы FastMe, формулу в Newick-формате можно посмотреть тут:
Пусть это дерево будет эталонным, выглядит оно так:
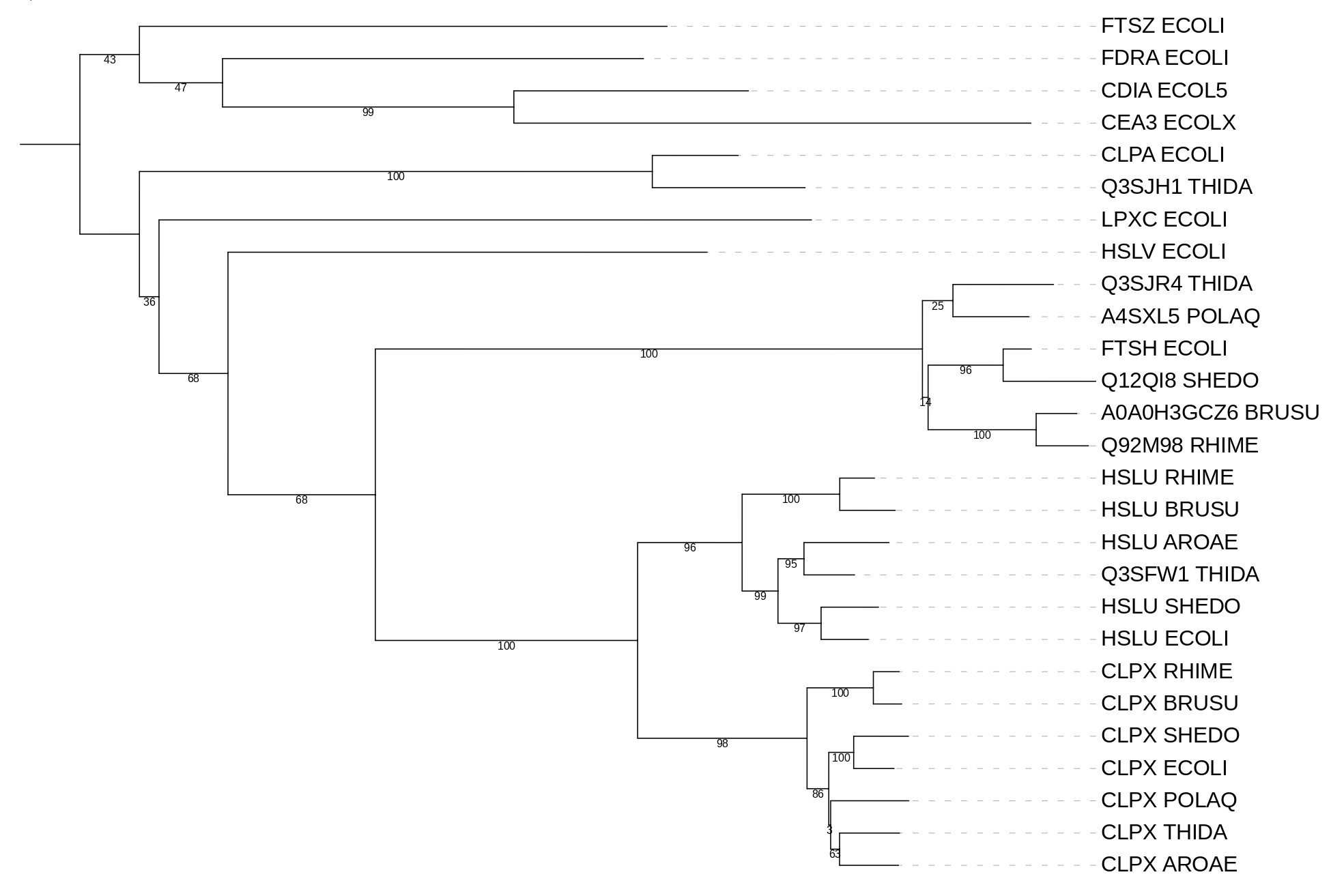
На изображении ниже я разделилиа белки по кладам по цветам, белки из кажодой клады являются ортологами, например, HSLU RHIME/HLSLU THIDA, CLPX ECOLI/CLPX SHEDO, HSLU ECOLI/HSLU BRUSU. Примеров паралогов являются белки из одной бактерии, например CLPX ECOLI и HSLU ECOLI, CLPX RHIME и HSLU RHIME, FITSH ECOLI и HSLU ECOLI.
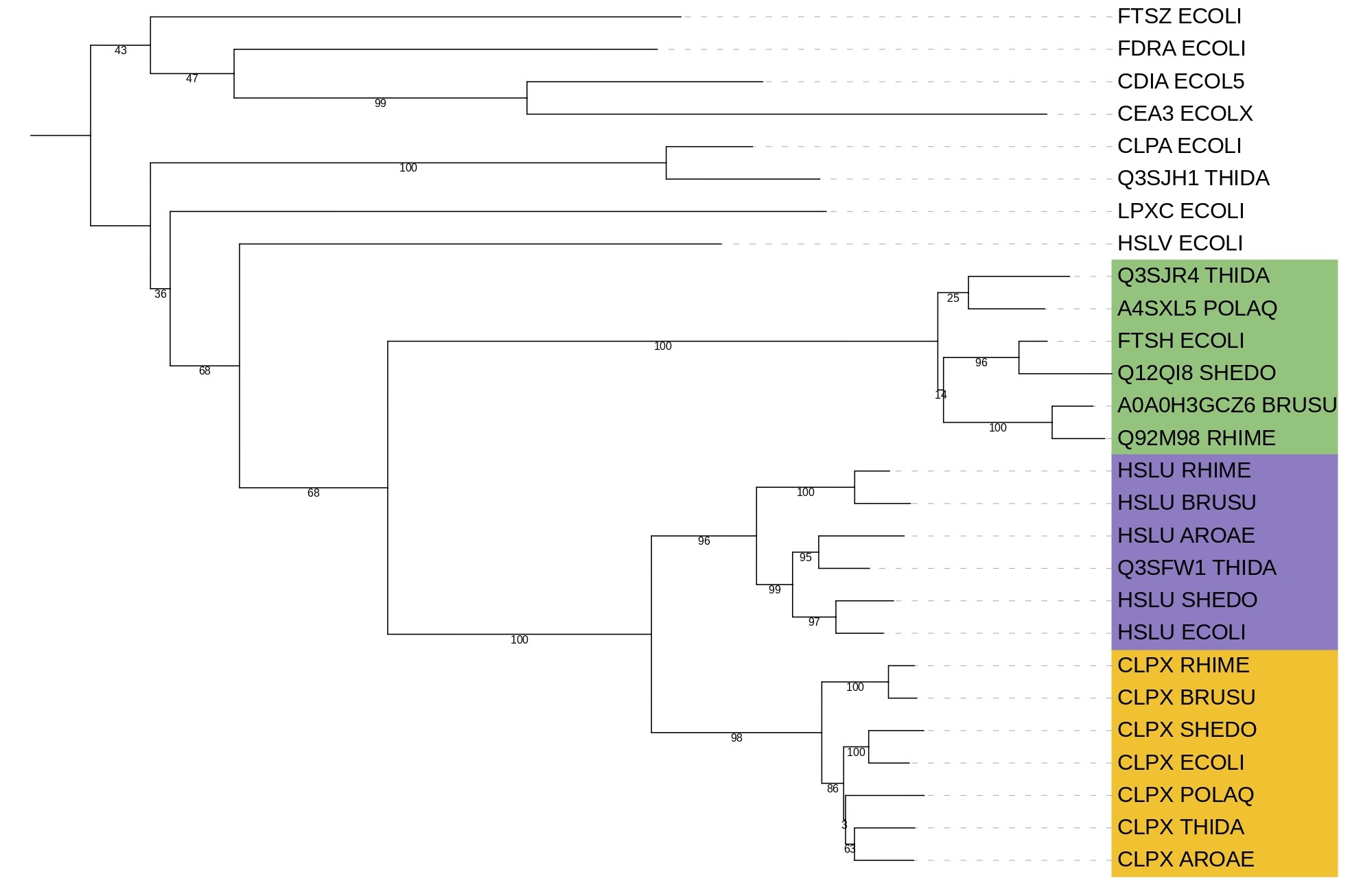
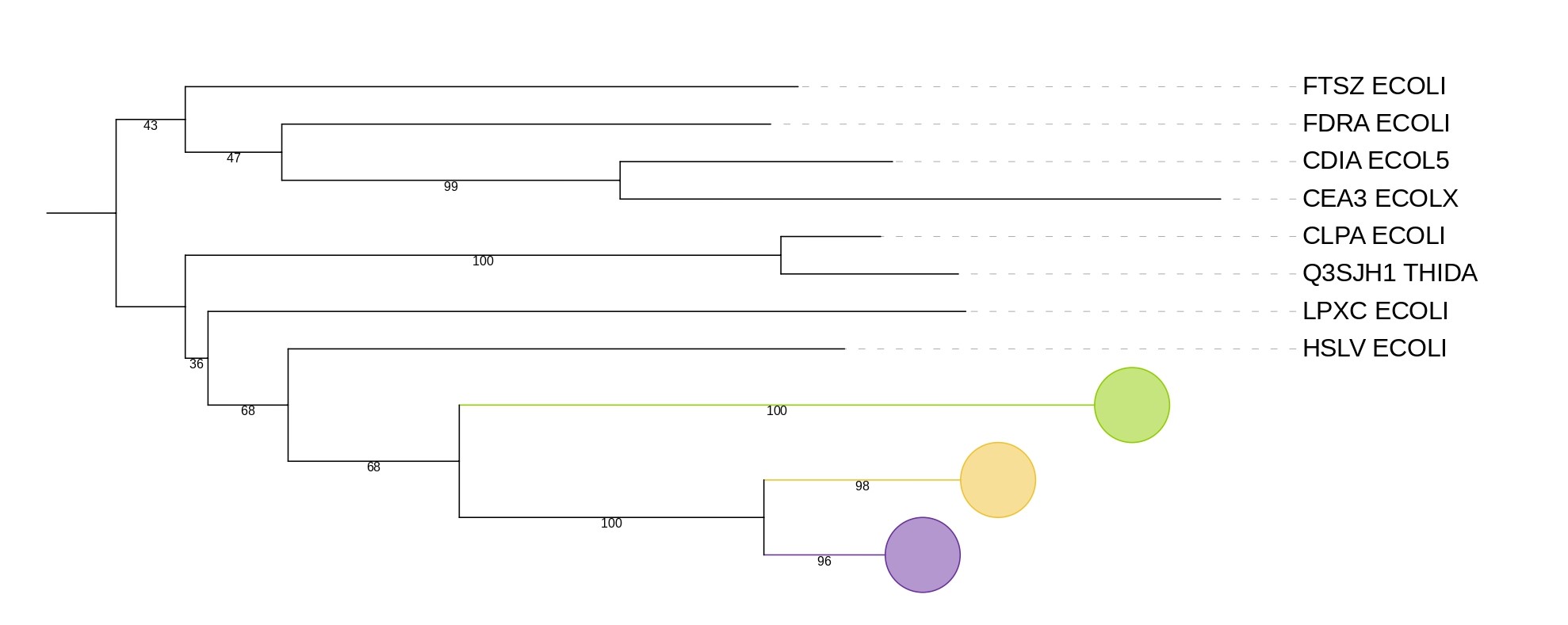
Далее, ветви содержащие более трёх последовательностей, были свёрнуты, можно посмотеть изображение ниже. Цвета сохранены в соответствии клад на рис.2.
Ниже представлено дерево построенное по генам 16S rRNA.
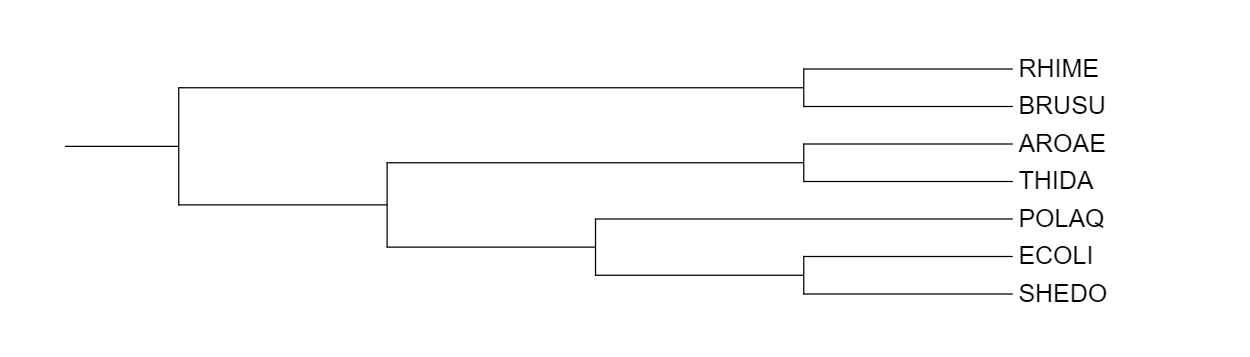
Если сравнивать реконструкцию филогении ортологических групп с филогенией бактерий, то отчётливо видно, что ортологическая группа отмеченная жёлтым(белок CLPX), в точности соответствует филогении бактерий как на рис.4. В данной кладе присудствуют все бактерии. В зеленой кладе (белок FTSH), отсутствуют AROAE, но филогения остальных тоже довольно близка к рис.4. В фиолетовой кладе (белок HSLU), отсутсвует POLAQ, но опять же, филогения других бактерий очень близка к рис.4. Таким образом, я сделала вывод, что реконструировання филогения ортологических групп у моих бактерий соответствует филогении самих бактерий.