Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке.
Название: Maltoporin(мальтопорин)
Организм: Escherichia coli
Функция: Участвует в транспортировке мальтозы и мальтодекстринов, селективная к ним часть белка находится на N-конце; также является рецептором для лямбда-фага
Выдача тут:OPM
Идентификатор UniProt: LAMB_ECOLI
Идентификатор PDB: 1AF6
Далее из UniProt взяла посоледовательность белка LAMB_ECOLI и запустила DeepTMHMM: текстовую выдачу можно увидеть тут Выдача TMHMM
Согласно ней, координаты бета-листов: 1(28-37), 2(64-72), 3(84-92), 4(104-113), 5(124-130), 6(149-157), 7(163-170), 8(196-201),9(211-118), 10(239-246), 11(252-259), 12(295-303), 13(310-317), 14(332-339), 15(346-352), 16(369-377), 17(382-393), 18(438-445).
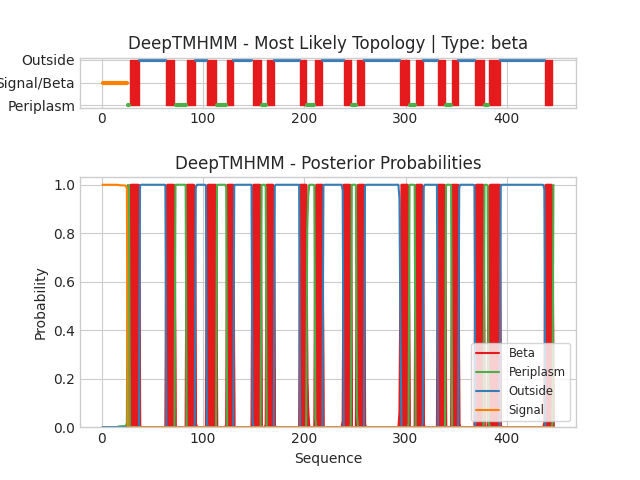
Координаты в соответствии с OPM:1(2-14), 2(40-53), 3(56-68), 4(80-89), 5(99-105), 6(110-113), 7(117-120), 8(124-133), 9(138-152), 10(165-182), 11(185-196), 12(211-223), 13(226-236), 14(252-254), 15(260-263), 16(267-280), 17(284-297), 18(303-315), 19(320-333), 20(339-352), 21(362-374), 22(376-378), 23(393-395), 24(407-420).
Таким образом, есть тяжи полностью не замеченные прграммой, например 2-14, 117-120, 226-236, 267-280,407-420. Другие участки имеют перекрытия от 1 до нескольких нуклеотидов, но ни один лист из OPM не совпадает польностью с предсказанием DeepTMHMM, что говорит о том, что предсказание довольно плохое.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был выдан белок Y2265_MYCTO,(NCBI_TaxID: 83331) который, предположительно, выполняет функцию транcмембранного переносчика. Название - Uncharacterized protein MT2327(Белок MT2327, с функцией, которая не опредлена), выделен из оргнизма Mycobacterium tuberculosis (strain CDC 1551 / Oshkosh).
UniProt: Y2265_MYCTO
Далее я запустила DeepTMHMM для Y2265_MYCTO, текстовую выдачу можно посмотреть тут: Выдача TMHMM
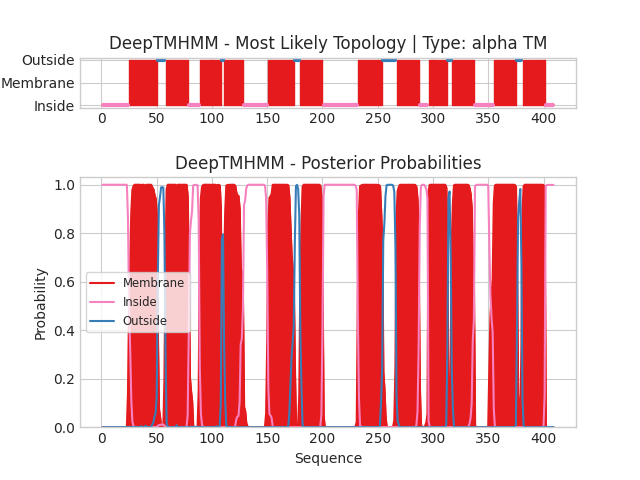
Предсказанные DeepTMHMM координаты трансмембранных спиралей: 1(25-50), 2(58-78), 3(89-108), 4(111-128), 5(151-174), 6(180-200), 7(232-254), 8(267-287), 9(296-313), 10(317-337), 11(355-375), 12(381-401).
После этого я скачала из раздела "Structure" SwissProt предсказание трёхмерной структуры Y2265_MYCTO и запустила PPM, параметры на рис.3. Я выбрала Topology(n-ter): in, потому что исходя из предсказания в DeepTMHMM N-конец расположен внутри клетки.

Здесь можно посмотреть выдачу PPM:PPM для Y2265_MYCTO
Тогда, согласно полученной топологии коордианты трансмембранных участков белка: 1( 26- 51), 2( 57- 78), 3( 90- 108), 4( 113- 131), 5( 152- 175), 6( 179- 200), 7( 234- 245), 8( 249- 257), 9( 264- 286),10( 294- 312),11( 317- 338),12( 355- 380),13( 381- 401)
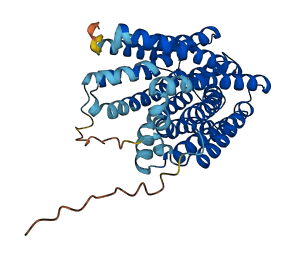
Теперь, если сравнить выдачи DeepTMHMM и PPM, то предсказания топологий имеют гораздо больше схожего в отличие от выдачи OPM и DeepTMHMM из задания 1. В данном случае у нас процент перекрытия каждой координаты горадо выше(часто отличие не более чем в 1-2 нуклеотида), и в выдаче DeepTMHMM не хватает лишь одной топологии (249 - 257). Таким образом, предсказание оказалось довольно достоверным. Если посмотеть на 3D-структуру Y2265_MYCTO из AlphaFold, где разными цветами указана степень достоверности данной модели в кажом отдельном участке(см.рис.4), то как раз примерно в середине белка(что соответствует участку (249-257)) достоверность низкая и очень низкая, тогда, можно предположить, что именно достоверность модели могла повлиять на достоверность PPM.