Базы данных
Мне был выдан список белков: Список ID
Я решила взять базу данных String
Данная база данных основывается на определении белков-белковых взаимодейтсвий. Она строит граф, на котором показывается тип взаимодействия между двумя белками, например, известые взаимодействия(из курируемых баз данных или экспериментально определенные взаимодействия) и предсказаннные взаимодействия(гены соседствуют, гены слияния(особый вид генов образованный в результате слияния двух независимых генов), совместное присутствие генов(гены не заимодействуют изолированно)). Таким образом я получила довольно объёмный граф из 72 узлов(один белок куда то пропал).
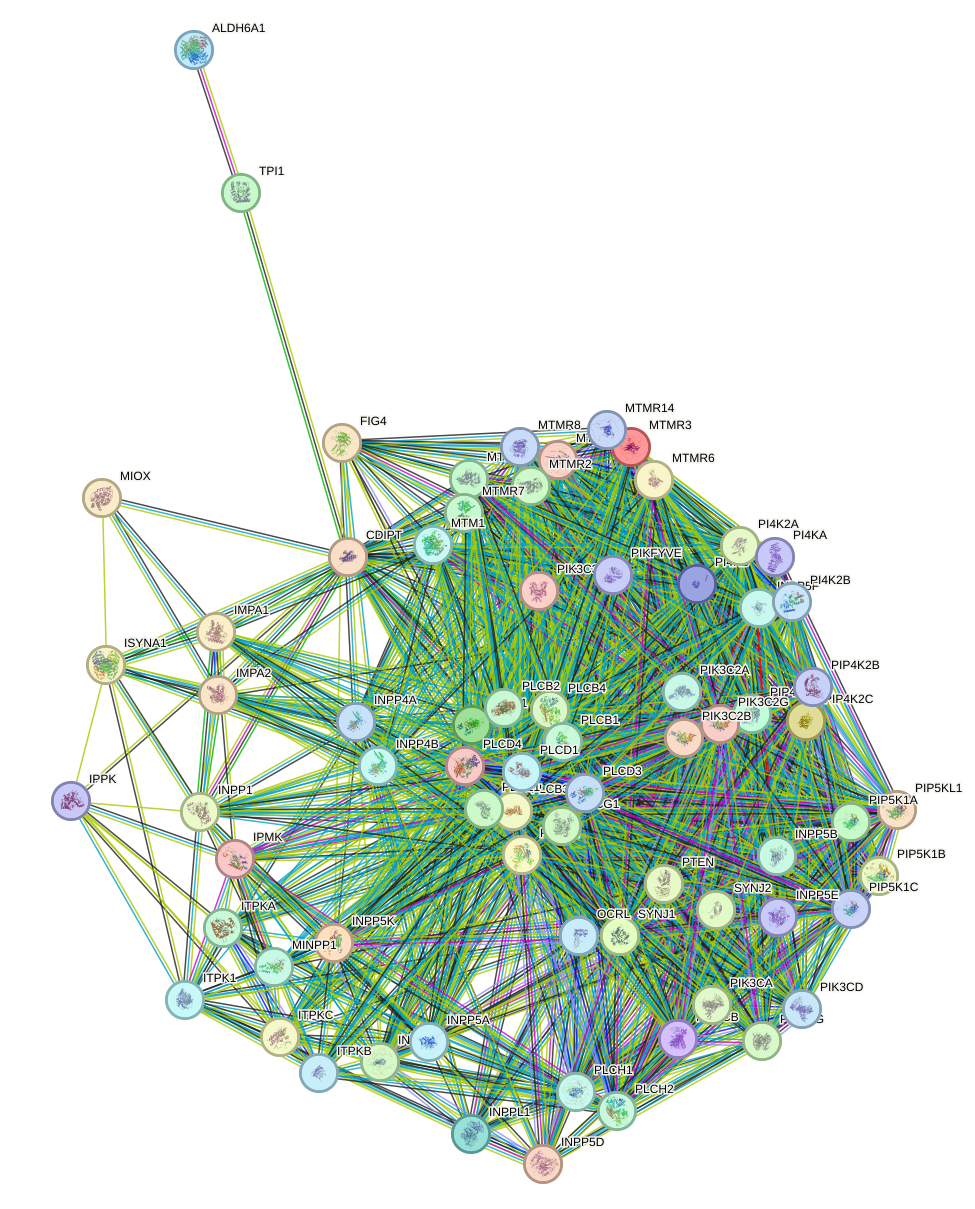
При этом, PPI enrichment p-value составлял < 1.0e-16, что означает, что гены составляют некотоую биологически осмысленную группу. То есть, данные белки имеют больше взаимодействий между собой, чем можно было бы ожидать от случайного набора белков одинакового размера и степени распределения, взятых из генома.
Далее я решила кластеризовать данный граф, по парметру клатеризации k-means clustering, то есть по центроиду(цендроид дерева - это вершина, после удаления которой граф подразделяется на множество поддеревьев). Теперь у меня получилась следующая картина:
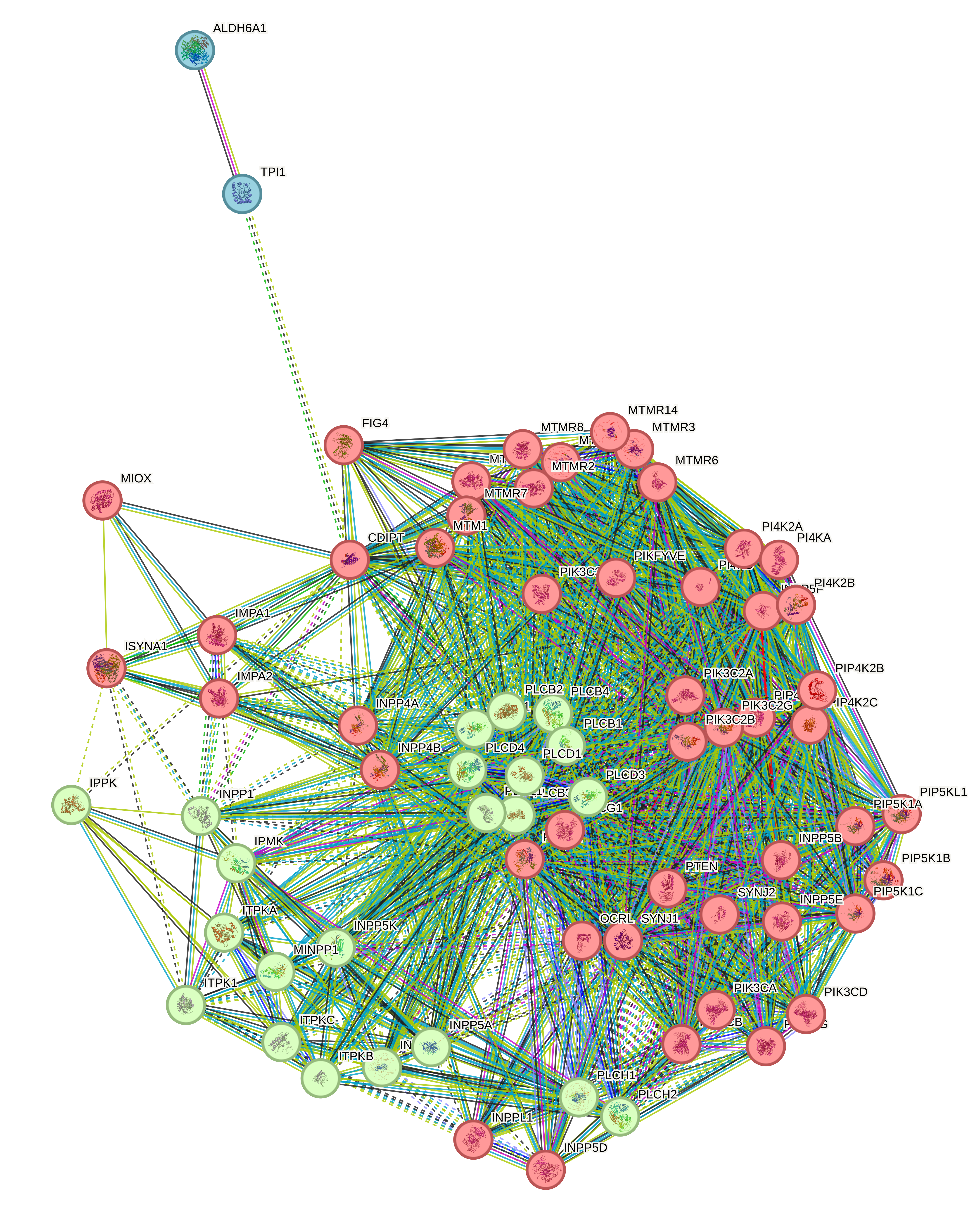
В параметрах я указала что хочу разделить на 3 клатера, и вот какое разделение получила:
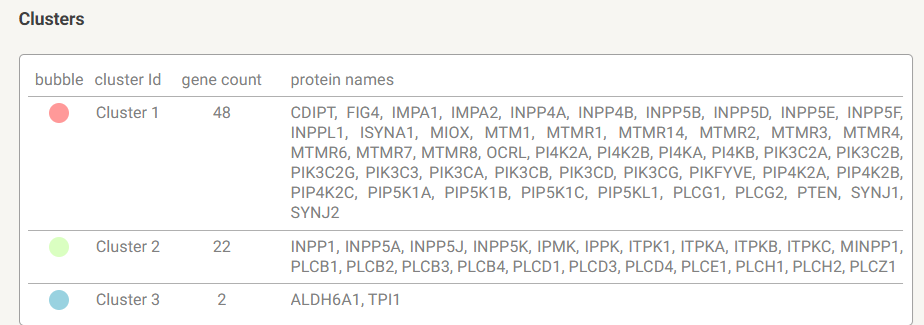
При этом в кластеры попало много гомологичных белков, например IMPA1, IMPA2 или PIP5K1A, PIP5K1B, PIP5K1C. Причем, если перейти в 3 полученных кластера(получается три разных графа), то исходя из записей KEGG, можно сделать вывод о том, что каждый из кластеров содержит белки связанные с метаболизмомом инозитола(шестиатомный спирт циклогексана), то есть можно сделать вывод о том, что все белки выданные мне и правда объединнены общей биологической функцией - учавствуют в синтезе инозитола, этот факт был предсказан ранее показателем PPI enrichment p-value.
Далее я решила провести анализ с базой данных GO. Загурзила туда список всех ID c параметром молеклярные функции, чтобы выявить специфическую активность генного продукта на молекулярном уровне. То есть сервис позволяет установить взаимоотноешние между сетью терминов и функций, и GO проводит тест на обогащение. За референс был взят Homo Sapiens(с ним будут сравниваться частоты терминов), а тест - Фишеровский.
В выдале лежат только P-value < 0,05, то есть только эти термины достоверны. И в данной выдаче, опять же, видно что есть очень сильно обогащённые термины - более 100, есть менее обогащённые - менее 0,01. Из выдачи терминов также видно, что данные белки связаны с метаболилзомом инозитола.
Вывод:
Таким образом, String удобен для поиска взаимодействий с белками, то есть с помощью него можно установить составляют ли белки биологически осмысленную группу. Причём формат выдачи у него очень удобный для восприятия формат выдачи. GO - позволяет проанализирвоать обогащение терминами, хотя формат выдачи у него менее удобный чем у String.