Поиск de novo сигналов в ДНК
Подготовка данных
Для выполнения задания с поиском сигнала посадки σ-субъединицы РНК-полимеразы была выбрана бактерия Actinomyces oris (референсный геном) Геном в формате Fasta
Далее, с помощью сервиса Operon-mapper был получен: список оперонов Промотором считаются 100 нуклеотидов перед опероном. В качестве материала для обучения было выбрано 50 случайных генов домашнего хозяйства. Для негативного контроля использованы случайные последовательности генома длиной 100 нуклеотидов.
Последовательности генов домашнего хозяйства
Здесь можно посмотреть файл для тестирвоания мотива:
последовательности тестирования
MEME
Была использована команда:
meme housekeeping.fasta -dna -mod zoops -nmotifs 3 -minw 6 -maxw 50 -maxsites 50
В результате работы программы был получен Негативный контроль . Найдено три мотива. Находки изображены на рисунках 1,2,3. Дальше решено было работать с находкой, у которой наименьшее E-value.
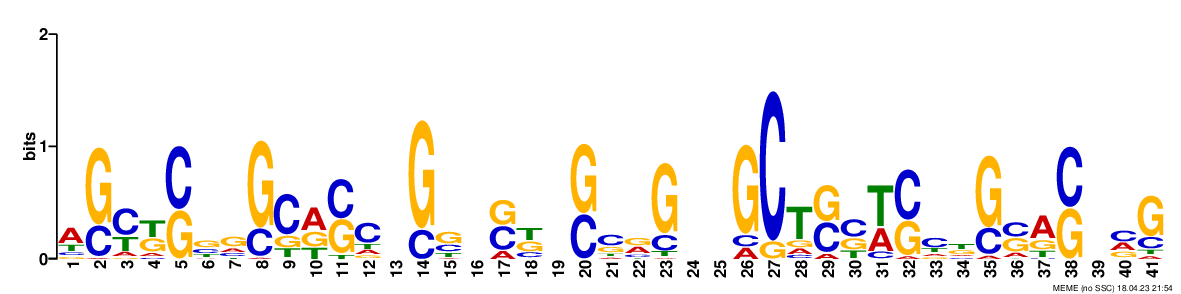

.png)
Поиск сигнала в материале для тестирования с помощью FIMO
Для поиска выбранного мотива в положительном и отрицательном контроле было установленно пороговое значение E-value = 0.0001. В позитивном контроле было найдено 1518 (из 2665), а в отрицательном - 29 (из 226)