Cравнительный анализ канонической ДНК и стеблей тРНК
Задание 1. Построение моделей структур А-, B- и Z-формы ДНК
Для построения моделей использовались следующие команды:
fiber -a -seq=GATCGATCGATCGATC gatc-a.pdb
fiber -b -seq=GATCGATCGATCGATC gatc-b.pdb
fiber -z -seq=GATCGATCGATCGATC gatc-z.pdb
Задание 2. Сравнение разных форм ДНК
На рисунке ниже изображён нуклеотид в составе ДНК и отдельно, нарисованный с помощью MarvinScetch. Атомы, обращённые в сторону большой бороздки покрашены в красный, в сторону малой - в синий.
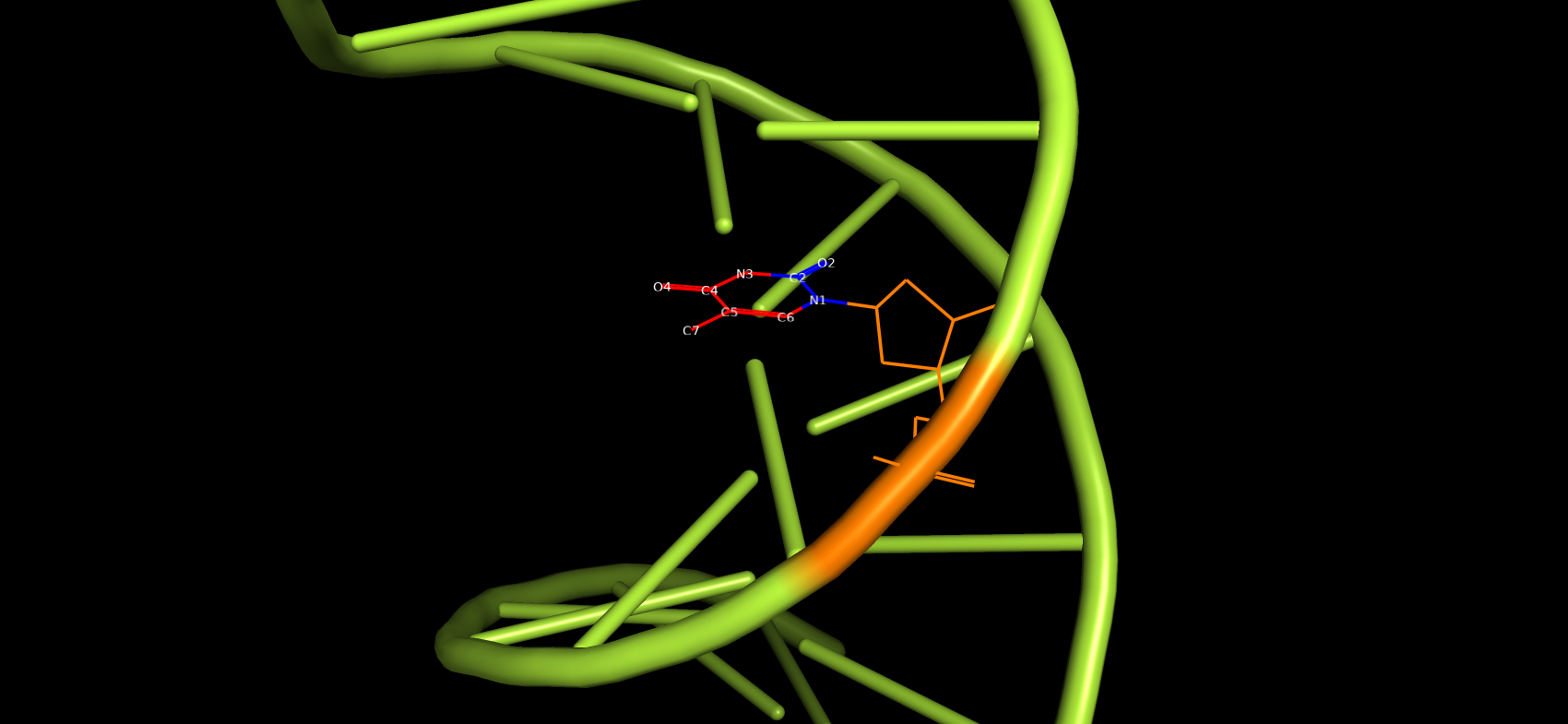
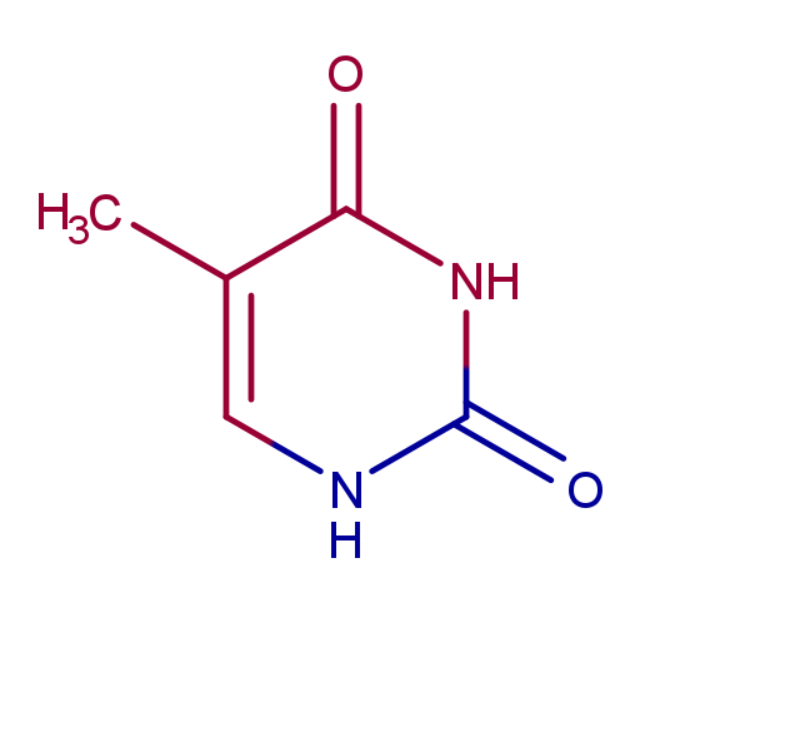
В сторону большой бороздки обращены атомы N3, C4, C5, C6, O4, O7.
В сторону малой бороздки обращены атомы O2, C2, N1.
| Форма ДНК | Тип спирали (правая или левая) | Шаг спирали | Число оснований на виток | Ширина большой бороздки | Ширина малой бороздки |
| А-форма | Правая | 28,7 А | 11 | 18,6 А | 9,5 А |
| B-форма | Правая | 33,7 А | 10 | 19,0 А | 11,7 А |
| Z-форма | Левая | 44,8 А | 12 | 28,8 А | 14,3 А |
Задание 3. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Для анализа была выбрана транспортная РНК дрожжей Saccharomyces cerevisiae (Entry: 3TRA). Ниже изображена её 3д-структура и развёрнутая схема, полученная с помощью модуля python forgi.

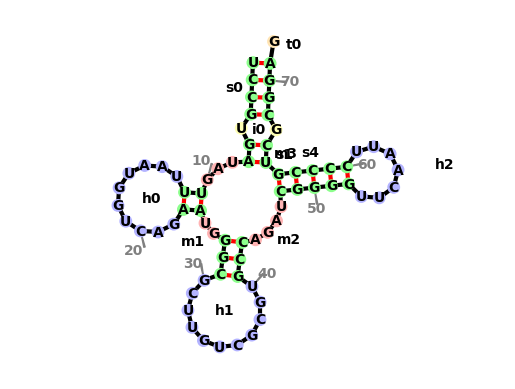
тРНК имеет 4 стебля. Их образуют следующие пары нуклеотидов:
1-7 c 71-65
48-52 c 64-60
41-43 c 29-27
11-12 c 24-23
Кроме того, в стабилизации структуры участвуют дополнительные водородные связи. Они образованы атомами нуклеотидов, образующих комплементарные пары, но не входящих в состав стеблей. Это 53 с 57, 54 с 17, 36 с 33, 14 с 8 и 15 с 47 нуклеотиды.
Из всех нуклеотидных пар 11 не являются каноническими:
Урацил(5) с гуанином(67)
Тимин(53) с аденином(57)
Псевдоуридин(54) с гуанином(17)
Цитозин(36) с урацилом(33)
Цитозин(38) с псевдоуридином(32)
Урацил(40) с гуанином(30)
Аденин(44) с гуанином(26)
Гуанин(10) с с урацилом(25)
Псевдоуридин(13) с гуанином(22)
Аденин(14) с урацилом(8)
Аденин(15) с урацилом(47)
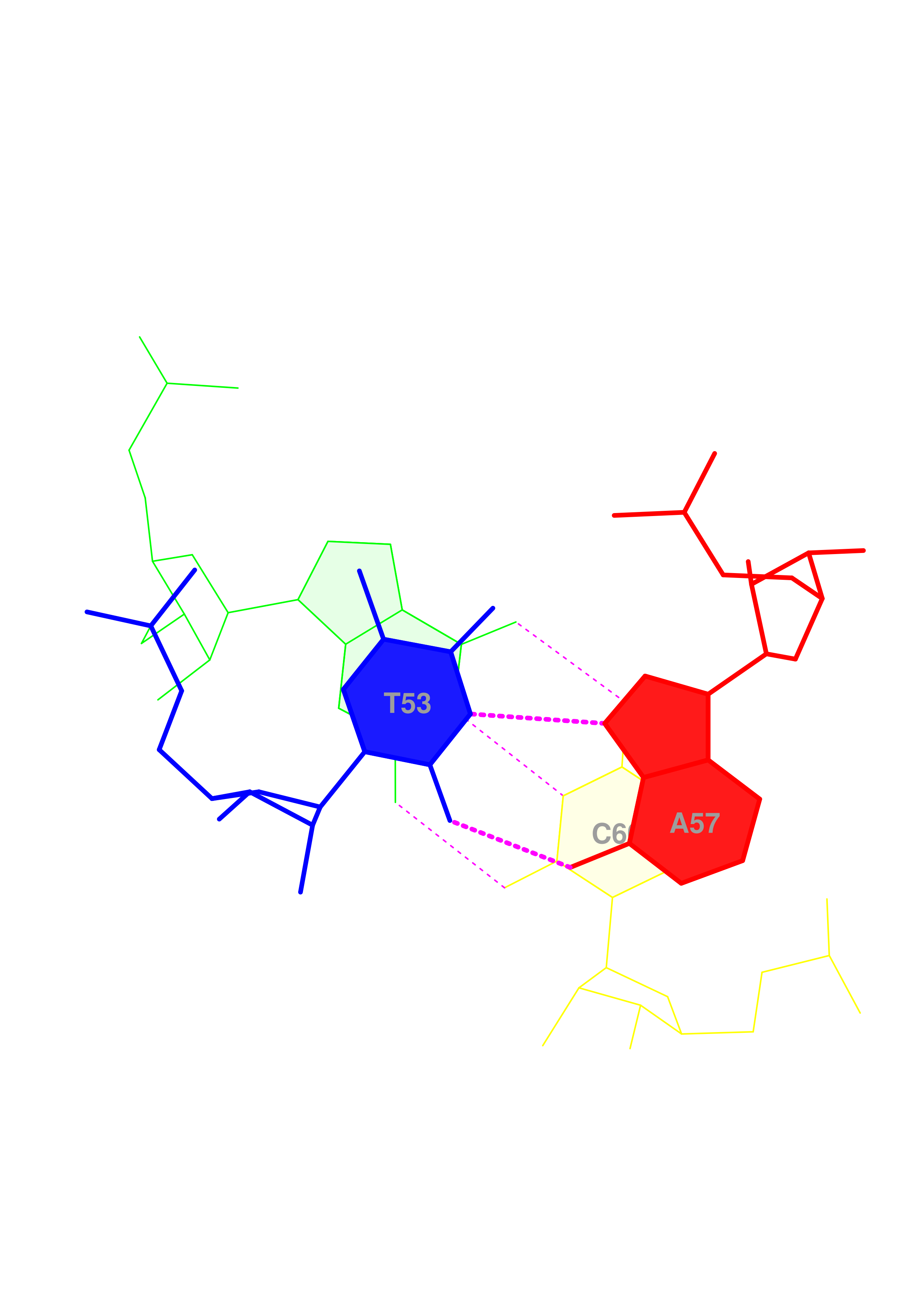
Наибольшая площадь перекрывания (8,41 квадратных ангстрем) наблюдается у тимина 53 с гуанином 52. На рисунке ниже показано стандартное изображение стекинг-взаимодействия между ними.
На рисунке показаны все торсионные углы. В таблице ниже представлены средние значение торсионных углов для трёх форм ДНК и исследуемой тРНК. Они были рассчитаны с помощью скрипта на питоне как среднее арифметическое для всех нуклеотидов, кроме краевых, так как для них некоторые углы неопределены. Получилось, что по значениям для разных углов тРНК ближе к разным формам ДНК. По значениям углов альфа, гамма и хи она ближе всего к В-форме, по значениям углов бета, дельта и зета - к А-форме, по значению угла эпсилон - к Z-форме.
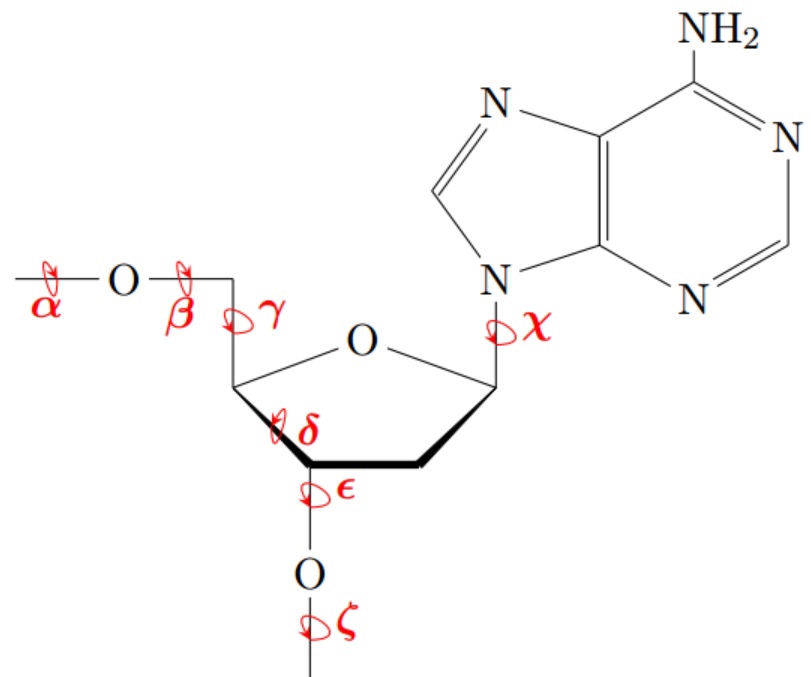
| Торсионный угол | alpha | beta | gamma | delta | epsilon | zeta | chi |
| тРНК | -42.47 | -12.02 | 61.16 | 82.96 | -97.55 | -69.7 | -119.22 |
| А-форма | -19.28 | -1.42 | 28.44 | 78.18 | -145.88 | -64.05 | -150.37 |
| B-форма | -54.33 | 55.93 | 50.51 | 112.85 | 42.06 | -60.45 | -107.43 |
| Z-форма | 31.16 | -61.14 | 22.16 | 87.66 | -99.32 | 28.78 | -42.3 |